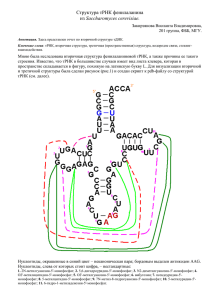
Структура пролиновой тРНК из бактерии Thermus
advertisement

Анализ структуры пролин-тРНК (CGG) из бактерии Thermus thermophilus. Ким Александр, гр. 201, ФББ МГУ Аннотация: биоинформатическими методами были проанализированы вторичная структура данной тРНК и внутримолекулярные контакты, поддерживающие ее третичную структуру. Ключевые слова: тРНК, вторичная структура тРНК, элементы вторичной структуры (акцепторный стебель, антикодоновый стебель, антикодон, DU-стебель, TψC-стебель, DU-петля, TψC –петля, вариабельная петля, антикодоновая петля), третичная (L-образная) структура тРНК, неканонические нуклеотидные пары, неспиральные стэкинг-взаимодействия, triple-взаимодействия. Введение: Транспортная РНК (тРНК) – это разновидность рибонуклеиновых кислот, представляющая полимерную макромолекулу, функцией которой является связывание аминокислоты, доставка ее к рибосоме и комплементарное взаимодействие с соответствующим кодоном мРНК, после чего данная аминокислота присоединяется к синтезируемой полипептидной цепи. Известно, что тРНК имеет вторичную структуру из четырех двуцепочечных стеблей и трех концевых петель, так называемый ”клеверный лист”, который в пространстве складывается в L-образную третичную структуру. Цель данной работы – построение вторичной структуры тРНК и поиск взаимодействий предполагаемо участвующих в формировании ее третичной структуры, при помощи обработки данных рентгено-структурного анализа исследуемой тРНК. Результаты: Следует отметить, что в исследуемом PDB файле нехватает 10 нуклеотидов со стороны акцепторного конца (6-и конечных комплементарных пар акцепторного стебля и 4-х нуклеотидов непосредственно акцепторного конца), к тому же существует ряд особенностей данного PDB файла, затрудняющих нормальную ориентацию в нем – 18-ый нуклеотид пронумерован как 17А, минорные нуклеотиды прописаны как гетероатомы, и т.д. Тем не менее мною была использована именно данная нумерация, чтобы избежать вохможной путаницы. Нуклеотидная последовательность цепи пролин-тРНК (участки раскрашены соответственно нижеприведенным таблицам 1 и 2): 5’ CGGGGAGUAGCGCAGCCCGGUAGCGCACCUCGUUCGGGACGAGGGGGGCGCUGG(5MU)(PSU)CAGAUCCAGUCUCCCCGACCA 3’ При помощи программ find_pair и analyze была построена таблица водородных связей между азотистыми основаниями на основе которой была оформлена таблица спиральных участков (Таблица 1): 4_:[..G]G-----C[..C]:..69 5_:[..G]G-----C[..C]:..68 6_:[..A]A-----U[..U]:..67 7_:[..G]G-----C[..C]:..66 10_:[..G]G-----C[..C]:..25 11_:[..C]C-----G[..G]:..24 12_:[..G]G-----C[..C]:..23 13_:[..C]C-----G[..G]:..22 26_:[..A]A-*---G[..G]:..44 27_:[..C]C-----G[..G]:..43 28_:[..C]C-----G[..G]:..42 29_:[..U]U-----A[..A]:..41 30_:[..C]C-----G[..G]:..40 31_:[..G]G-----C[..C]:..39 32_:[..U]U-----A[..A]:..38 49_:[..G]G-*---U[..U]:..65 50_:[..C]C-----G[..G]:..64 51_:[..U]U-----A[..A]:..63 52_:[..G]G-----C[..C]:..62 53_:[..G]G-----C[..C]:..61 Акцепторный стебель (4-7, 66-69) DU-стебель (10-11, 22-25) Антикодоновый стебель (26-32, 38-44) TψC -стебель (49-53, 61-65) По таблице 1 была построена схема вторичной структуры тРНК (раскраска в соответствии с данной таблицей): 3' – каноническая комплементарная связь – неканоническая комплементарная связь 5' @ C 3 @ G @ G G G A Акцепторный стебель DU-стебель DU-петля C C G C G G U A A A C G C G U Акцепторный конец G C C C C U TψC-стебель TψC-петля C U G A C C U A G A C G C U G G 5 P G C G G G G G псевдоуридин A G C G C A Антикодоновый стебель G @ A 3 @ C @ C A C C U C G C A G U U C G 5-метилуридин Вариабельная петля G G Антикодон Антикодоновая петля Так же по данным програм find_pair и analyze была построена таблица неспиральных водородных взаимодействий (комплементарные взаимодействия нуклеотидов, не образующие спирали; Таблица 2): 8_:[..U]U-**--A[..A]:..14 15_:[..G]G-**+-C[..C]:..48 18_:[..G]G-**+-P[PSU]:..55 19_:[..G]G-----C[..C]:..56 21_:[..A]A-**+-G[..G]:..47 54_:[5MU]u-**--G[..G]:..58 Анализом визуализации РСА-структуры тРНК в программе RasMol были определены triple-взаимодействия (водородные взаимодействия сразу трех нуклеотидов, лежащих в одной плоскости): 25 С 45 G 13 С 22 G 21 A 14 A 10 G 46 G 8U А так же неспиральные стэкинг-взаимодействия (гидрофобные взаимодействия лежащих друг под другом в параллельных плоскостях пар или одиночных нуклеотидов, не идущих друг за другом в цепи; раскраска в соответствии с таблицей 2): 15 G – DU-петля 19 G – DU-петля 45 G – вариаб. петля 14 A – DU-петля 59 A - TψC-петля 57 A - TψC-петля 9A 22 G – DU-стебель 18 G - DU-петля 46 G – вариаб. петля 58 G - TψC-петля 21 A – DU-петля 61 C – TψC-стебель 48 A – вариаб. петля Как обьединение полученных данных о трех вышеприведенных типа взаимодействий была построена схема взаимодействий отвечающих за стабильность пространственной структуры тРНК: 15 G – DU-петля // 59 A - TψC-петля // G C; A G G – неспиральные стэкинг-взаимодействия нуклеотидов A – неспиральные комплементарные взаимодействия (раскраска по таблице 2) – triple-взаимодействия G C G; A A U A C 19 G – DU-петля 5' 57 A - TψC-петля @ C 3 @ G @ G 18 G - DU-петля 58 G - TψC-петля 61 C – TψC-стебель C C G C G G U A A 45 G – вариаб. петля 9A 46 G – вариаб. петля 21 A – DU-петля 48 C – вариаб. петля A C G C G U C A G G A G C C C C U G C 15 G – DU-петля 59 A - TψC-петля U G A C C U A G A C G C U G G 5 P G C G G G G G A 14 A – DU-петля G 22 G – DU-стебель C A G C G C A C C U C G U U C G G G 8U 13 С – DU-стебель Так же были получены предсказания вторичной структуры данной тРНК программой mfold c использованием алгоритма Зукера. Нижеприведенный вариант является наиболее схожим со структурой, построенной на основании анализа водородных взаимодействий – показаны четыре стебля, причем акцепторный и TψC- стебли совпадают полностью; в отображении антикодонового стебля и петли, наличествует ряд незначительных отличий, связанных с тем, что программа, разорвав неканоническое взаимодействие A-G в начале стебля, добавила лишнюю неканоническую связь G-U в петле; при построении DU-петли mfold опять же добавил два лишних взаимодействия, одно из которых к тому же является неканоническим. В целом, на мой взгляд, алгоритм Зукера в данной реализации пригоден лишь для общего представления особенностей структуры тРНК, т.к. выявленные неточности могут влиять на корректность выводов, связывающих структурные особенности данной молекулы с функциональными. Обсуждение: найденные взаимодействия полностью соответствуют современным представлениям, по которым третичная L-структура тРНК формируется за счет сближения DU- и TψC- петель. Большое их разнообразие подчеркивает особую важность такой пространственной структуры для нормального функционирования тРНК – это может быть значимо для узнавания тРНК специфической ей аминоацилтРНК-синтетазой и взаимодействия с ней, а так же для процессов непосредственного синтеза полипептидной цепи на рибосомах. Сопроводительные материалы: В файле 1h4q содержится скрипт для Rasmol, позволяющий визуализировать основные элементы структуры тРНК из PDB записи 1h4q.pdb. Материалы и методы: пространственная структура и нуклеотидная последовательность пролин тРНК были взяты из базы данных PDB (Protein Data Bank), идентификационный номер структуры (PDB ID) – 1h4q. При подготовке материалов были использованы следующие инструменты: Программы find_pair и analyze пакета 3DNA для Unix (для получения таблицы водородных связей между азотистыми основаниями данной тРНК); Программа mfold пакета 3DNA (для построения схемы вторичной структуры тРНК на основании нуклеотидной последовательности по алгоритму Зукера); Программа RasMol (для изучения особенностей третичной структуры тРНК - выявление контактов нуклеотидов, отвечающих за стабильность пространственной конфигурации и др).