ДНК-штрихкодирование в изучении разнообразия и
advertisement

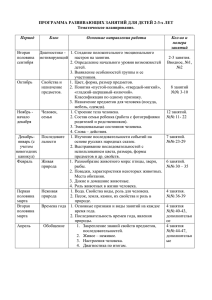
Современные методы исследования морских беспозвоночных Научно-популярный материал 1: ДНК-штрихкодирование в изучении морских беспозвоночных ВВЕДЕНИЕ Со времен первых экспедиций по изучению морских беспозвоночных прошло несколько столетий. Согласно Международному реестру морских видов (World Register of Marine Species- WoRMS; http://www.marinespecies.org/) в настоящий момент общая численность известных морских многоклеточных животных составляет более 215 000 видов, хотя потенциальное их число гораздо выше. Знания о морской фауне носят неполный характер, что прежде всего связано с неравномерностью изучения Мирового океана, отрывочными данными о подводном мире. Жак-Ив Кусто говорил об исследователях моря так: «Океанографы (дай им Бог здоровья!) - слепые нищие, ковыляющие на костылях из тросов» (Кусто, Даген, 1976). Оснащение исследовательских судов новыми технологическими приспособлениями способствует проникновению на новые глубины и в труднодоступные морские местообитания, наряду с этим все активнее осваиваются шельфовые районы Мирового океана. История морских исследований является наглядным примером эволюции методов изучения фауны - классические морфологические описания постепенно уступают место современным методам генетической идентификации видов. Происходит это не только из-за стремительного технологического прогресса, но и потому что только лишь морфологическими методами становится невозможным определить скрытые виды, учесть внутривидовую изменчивость, а также принять во внимание, что не все стадии организма или наличие обоих полов исследуемого животного возможно получить. Исследователю необходимо обрабатывать большие объемы информации для того, чтобы учесть все признаки вида, а это занимает немало времени и усилий. На помощь морским зоологам приходит генетический баркодинг (ДНК-баркодинг) - использование коротких маркеров, а именно последовательностей ДНК субъединицы I митохондриальной цитохром с оксидазы (COI) многоклеточных животных. ДНК-баркодинг лежит в основе таких крупных исследовательских проектов, как глобальные базы данных по видовому разнообразию the Consortium for the Barcode of Life (CBOL, http://www.barcodeoflife.org/) и the Barcode of Life Data Systems (BOLD, http://www.barcodinglife.org/). Также появились специализированные базы генетических данных - например, по тропическим экосиcтемам the Moorea Biocode Project, полярным экосистемам the Polar Barcode of Life (PolarBOL), по разным таксономическим группам животных и т.д. (см. приложение). Для морских биологов особый интерес представляет база данных по морским животным Marine Barcode of Life, где собрано более 50 тысяч последовательностей морских организмов. Базы генетических данных по морским беспозвоночным находят все более широкое применение на практике, их используют также для изучения популяционной генетики, в аквакультуре, мониторинге изменения окружающей среды, криминалистике и т.д. Почему же этот метод до сих пор не стал общепринятым при видовом определении морского биоразнообразия? ОПРЕДЕЛЕНИЕ И МЕТОД Генетический баркодинг (или ДНК-баркодинг) - это метод, в основе которого лежит использование коротких генетических маркеров для определения таксономического положения организма. В отличие от молекулярно-филогенетичеких методов ДНК-баркодинг не строит классификацию морских организмов, а определяет их таксономическую принадлежность в рамках (http://en.wikipedia.org/wiki/DNA_barcoding). уже В тоже известной время это классификации лишь способ для идентификации известных видов методом сравнения их ДНК последовательностей, но не характеристика для классификации таксонов (рис. 1). Рис. 1. Общая схема ДНК-баркодинга http://www.barcodeoflife.org/content/about/what-dna-barcoding). (с изменениями по Сама идея использования изменчивости на уровне нуклеотидных последовательностей для определения видового разнообразия не новая. Важный вклад в изучение архебактерий был сделан на основе анализа последовательностей рибосомальной ДНК, в то время как аллозимный метод и анализ последовательностей рибосомальной и митохондриальной ДНК легли в основу молекулярной систематики животных и растений. Предпосылкой для разработки нового подхода стали исследования Баклин и соавт. (1999), которые прочитали последовательности участка гена субъединицы I цитохром с оксидазы (COI) у восьми видов планктонных копепод и обнаружили возможность различать даже близкородственные виды с помощью этого генетического маркера. В 2003 году исследовательская группа Пола Хеберта (Paul D.N. Hebert) из Университета Гельфа в Канаде приступила к созданию единой библиотеки последовательностей митохондриальной ДНК (митДНК). В основе проекта лежала идея использования универсальных штрих-кодов, таких которые используют в супермаркетах для определения товаров. Научный проект Хеберта был призван унифицировать и облегчить процесс определения видов наряду с увеличением числа известных таксонов и удешевлением процесса чтения их генетических последовательностей. Модель использования универсальных генетических штрих-кодов для определения видов животных была успешно проверена учеными на 200 последовательностях участка гена COI чешуекрылых (Hebert et al., 2003), что позднее легло в основу генетической библиотеки CBOL. В чем состоит преимущество использования генетических маркеров перед морфологическими признаками в определении организмов? Прежде всего, эта информация представлена в универсальной и емкой форме. Генетические штрих-коды (англ. barcodes) стандартные последовательности из определенного региона генома животного - видоспецифичны, то есть они отличаются у разных видов морских животных. Генетические штрих-коды образованы лишь 4 альтернативными нуклеотидами в каждой позиций (A-G-TC), но набор сайтов для изучения остается огромным. Например, исследование всего 15 из таких нуклеотидных позиций дает вероятность образования 415 кодов, что уже в 100 раз больше числа всех предполагаемых таксонов! Не смотря на то, что в ДНК есть неизменные, важные в функциональном отношении, участки, а генетическая вариабельность проявляется даже на внутривидовом уровне, метод чтения ДНК последовательностей позволяет использовать важные для организмов белок-кодирующие последовательности. Принимая во внимание, что большинство замен в третьей нуклеотидной позиции кодонов слабо подвержено селекционному процессу из-за их четырех-кратной вырожденности, а длина изучаемых последовательностей достаточно велика, то полученная информация по генетическому отличию видов представляется достаточно убедительной. В перспективе предполагается чтение более длинных генетических последовательностей, поскольку это позволит обойти стороной некоторые биологические «тонкости» строения генома: известно, что у ракообразных нуклеотидный состав в третьей позиции кодона (триплета из нуклеотидов) чаще всего представлен парой А-Т, также у близкородственных видов большинство нуклеотидных позиций сходны по своему составу. Тем не менее, формулы, которая позволила бы рассчитать универсальную длину последовательности ДНК, идеально подходящую для определения видов, не существует, потому что сам процесс эволюции носит неравномерный характер в разных частях генома и между таксонами (Hebert et al., 2003). В основе ДНК баркодинга лежит простая идея: поскольку у большинства эукариотических организмов есть митохондрии, а мутационный процесс в митохондриальной ДНК протекает быстро, то у разных видов живых организмов накапливается достаточное количество отличий в последовательностях митДНК при относительно невысоком уровне вариации ДНК в пределах этих видов. За редким исключением у большинства известных видов в митохондриях (в отличие от ядерной ДНК) передача информации осуществляется от предка к потомку только по материнской линии, нет интронов, большое количество копий в каждой клетке и минимальный уровень рекомбинации (Radulovici et al., 2010). В качестве основной последовательности в ДНК-баркодинге используют участок длиной 658 пар нуклеотидов, локализованный в гене первой субъединицы цитохром с оксидазы (COI). Этот участок расположен на 5’-конце, его окружают консервативные последовательности ДНК. Благодаря этому стало возможным создание консервативных праймеров для проведения полимеразной цепной реакции (ПЦР). Наиболее широко используются «фолмеровские праймеры», которые получили свое название по фамилии исследователя, который их впервые синтезировал (Folmer et al., 1994). Важно, что у многоклеточных организмов различие в генетических расстояниях в пределах вида составляет менее 3%, в то время как между видами эта цифра колеблется в диапазоне 10-25%. Для ДНК-баркодинга интересным представляется люфт между этими значениями («barcoding gap»), в этих пределах корректно применять методы чтения генетических последовательностей и сравнивать их для определения «генетических границ вида» (рис. 2). Рис. 2. Диаграмма гипотетической чистоты распределений генетических расстояний на внутри- и межвидовом уровне (с изменениями по Bucklin et al., 2011, стр. 475, рис. 1а). Для подтверждения этого факта были использованы данные огромной базы генетических последовательностей Национального центра биотехнологической информации (NCBI) GenBank (http://www.ncbi.nlm.nih.gov/genbank). ПРЕИМУЩЕСТВА ДНК-БАРКОДИНГА Преимущества использования ДНК-баркодинга перед классическими методами определения видов морских беспозвоночных: - Возможность определения видовой принадлежности поврежденных особей, их фрагментов. Это позволяет сохранить уникальный образец животного практически полностью, взяв для анализа небольшую его часть. - Работа с любыми стадиями развития морского организма, в то время как многие классические определители построены на изучении определенных стадий жизненного цикла животного. - Идентификация видов-двойников и криптических (скрытых) видов, что крайне сложно или невозможно сделать только по морфологическим признакам. - Возможность изучать группы морских беспозвоночных с небогатыми морфологическими характеристиками. - Универсальность представления результатов в виде последовательностей, построенных из 4 нуклеотидов. - Быстрота определения видов, возможность единовременной идентификации группы организмов. - Управление большими объемами информации при относительно небольших затратах времени. - Общая доступность информации, ее использование в локальных и глобальных исследованиях. - Доступность метода для нетаксономистов. ОГРАНИЧЕНИЯ МЕТОДА: - Низкое разрешение метода при работе с гибридами, политипическими (генетически разнородными) и недавно возникшими видами, а также видами с небольшой скоростью эволюции. Наибольшие трудности в использовании ДНК-баркодинга возникают при работе с губками, гребневиками и некоторыми стрекающими (кораллы и актинии). Выяснено, что скорость эволюции митДНК у этих беспозвоночных в 10-20 раз ниже, чем у других морских многоклеточных (Huang et al., 2008). - Псевдогены (ядерные копии гена COI). Исследования пресноводных ракообразных рода Orconectes методом ДНК-баркодинга показали, что в ядрах их клеток содержатся копии генов COI, которые отличаются от последовательностей митДНК (Song et al., 2008). Если учитывать всё разнообразие псевдо-последовательностей, то по ошибке можно описать несуществующие виды морских организмов. - Контаминантные (чужеродные) генетические последовательности. Показано, например, что использование стандартных праймеров для ДНК-баркодинга при некоторых условиях может приводить к синтезу ДНК последовательностей морских холодноводных геммапротеобактерий (Sidall et al., 2009). - Межвидовая гибридизация (обмен генами между митохондриями разных видов). Так, установлено, что между двумя сосуществующими видами моллюсков Littorina fabalis и Littorina obtusata практически отсутствует отличие по митДНК, хотя по морфологическим признакам и другим генетическим маркерам эти виды хорошо различимы (Kemppainen et al., 2009). Кроме того, большая часть коллекций морских организмов, которая хранится в музеях по всему миру, была подготовлена с использованием формалина. Это вещество приводит к разрушению ДНК, и, соответственно, делает недоступным для использования музейные коллекции. Учитывая, что сбор морской фауны проводят в дорогостоящих экспедициях, часто сфокусированных лишь на определенных группах животных, пополнение коллекций новыми образцами оказывается довольно затратным мероприятием. В определении морских беспозвоночных по морфологическим признакам необходимо участие специалистовзоологов, число которых постоянно сокращается, а привлекать непрофессиональных наблюдателей к исследованию морской фауны оказывается практически невозможным. Несмотря на все эти ограничения, ДНК-баркодинг остается привлекательным методом для изучения биоразнообразия Мирового океана. ДНК-БАРКОДИНГ В МОРСКИХ ИССЛЕДОВАНИЯХ, ЕГО ПРИМЕНЕНИЕ НА ПРАКТИКЕ ДНК-баркодинг и определение видов морских беспозвоночных. С 2004 года по настоящее время самым крупным остается международная база данных на основе полученных последовательностей ДНК-баркодинга The Consortium for the Barcode Life (CBOL; http://barcoding.si.edu). Цель проекта - развитие ДНК-баркодинга в качестве стандарта для определения биологических видов, стимулирующего быстрый сбор высококачественных последовательностей ДНК в общедоступной библиотеке данных, а также развитие новых средств и методов для удешевления, быстроты и мобильности баркодинга. CBOL включает более 200 организаций-участников из 50 стран мира. В 2010 году учрежден международный проект The International Barcode of Life (iBOL; http://www.ibol.org), с помощью которого запланировано создать библиотеку баркодов для 500 тыс. видов до 2015 года. В сотрудничестве с The Census of Marine Life (CoML; http://www.coml.org) CBOL учредил международную программу по баркодингу морского биоразнообразия (MarBOL; http://www.marinebarcoding.org). MarBol собирает информацию по всем группам морских организмов. Расширенные базы данных последовательностей участков гена COI собраны для важных с научной и практической точки зрения морских видов (Bucklin et al., 2011). Наиболее изученными среди морских беспозвоночных по числу последовательностей гена COI являются моллюски и ракообразные. ДНК-баркодинг позволил создать обширную базу генетических данных и различить спорные виды в разных группах моллюсков (хитоны, брюхоногие, двустворчатые и головоногие) из разных местообитаний (от шельфа до глубоководных гидротермальных источников). Одними из первых исследований по пороговым значениям признаков и недостаточному числу проб при ДНК-баркодинге были проведены на моллюсках каури (род Cypraea). В результате анализа последовательностей их митДНК было установлено, что недостаточное количество образцов может привести к существенным ошибкам при генетическом определении видов (Meyer, Paulay, 2005). На планктонных брюхоногих моллюсках (Pteropoda, Heteropoda) был показан наибольший процент генетического различия (> 3%) внутри одних и тех же видов, обитающих в разных частях Мирового океана, что позволило предположить существование большего числа видов среди них, а, соответственно, и необходимость проведения дополнительной таксономической ревизии внутри этих таксонов (Jennings et al., 2010). Ракообразные, представленные во всех морских биотопах, играют важную роль в пищевых сетях. Costa и соавт. (2007) использовали последовательности 150 видов для видовой идентификации представителей разных семейств ракообразных, в пределах которых были обнаружены новые виды. Исследования по определению видов с использованием метода ДНКбаркодинга были проведены на личинках эуфаузиевых (Bucklin et al., 2007) и ротоногих (Barber, Boyce, 2006) раков. Исследования рифовой фауны ракообразных, представленной преимущественно десятиногими, ротоногими и перакаридами, во Французской Полинезии установили, что существенное число видов представлено там единичными экземплярами синглтонами (англ. singleton), обитающими на кораллах рода Pocillopora (Plaisance et al., 2009). Чтение последовательностей обнаруженных впервые видов существенно пополнило баркодинговых баз данных. Крупное исследование было проведено на иглокожих, результатом работы стало занесение в библиотеку последовательностей участка гена COI для 191 вида беспозвоночных (лишь 30% из них было позаимствовано в GenBank) (Ward et al., 2008). Интересно, что не только морские беспозвоночные, но и их паразиты, могут быть изучены с помощью секвенирования последовательностей митДНК: так, было обнаружено 20 видов трематод, обитающих в моллюсках, ракообразных и многощетинковых червях прибрежной полосы Новой Зеландии (Leung et al., 2009). Использование COI-последовательностей в других исследованиях по биологии. Накопленные в базах данных последовательности митДНК служат удобной основой для проведения исследований по популяционной генетике и филогеографическому анализу морской фауны. Например, сравнение популяций антарктического криля Euphausia superba из разных местообитаний показало генетическое сходство между ними, в то время как генетические характеристики внутри отдельных популяций ракообразных значительно отличались. Это объясняется тем, что криль способен выживать в зимний период и мигрировать на большие расстояния в Южном океане (Batta-Lona et al., 2011). Последовательности митДНК служат основой определения рациона питания морских животных и птиц. Интересно, что с помощью анализа последовательностей участка гена COI был установлен спектр питания глубоководных амфипод Scopelocheirus schellenbergi и Eurythenes gryllus, наблюдения за которыми в естественной среде не представляются возможными (Blankenship, Yayanos, 2005). Применение метода на практике. С развитием навигации во многих водоемах появились виды-вселенцы, многие из которых утратили свои диагностические характеристики после поселения в новых местообитаниях. Так, произошло с амфиподами из эстуария реки Св. Лаврентия в Канаде: образцы ракообразных были определены по морфологическим признакам как вид Marinogammarus obtusatus, после ДНК-баркодинга обнаруженных беспозвоночных оказалось, что это понто-каспийский вселенец Echinogammarus ischnus (Radulovici et al., 2009). Расшифрованные участки СOI-генов нашли широкое применение в определении контрафактных морепродуктов. Например, известны многочисленные случаи когда дорогие морепродукты заменяют их дешевыми вариантами. Это не только обман ради выгоды, но и потенциальная опасность для здоровья потребителей, поскольку многие морские обитатели способны выделять токсины и накапливать тяжелые металлы. С помощью ДНК-диагностики по митохондриальным последовательностям можно легко определить некачественный товар. Кроме того, с помощью метода ДНК-баркодинга можно выявить незаконно выловленные виды, которые находятся под охраной. ПЕРСПЕКТИВЫ ПРИМЕНЕНИЯ ДНК-БАРКОДИНГА Новые методы изучения труднодоступных глубинных районов Мирового океана непременно приведут к описанию новых видов морских беспозвоночных. Экспедиционные суда, оборудованные специальными лабораториями, позволят работать с нефиксированным материалом, что сделает возможным пополнять библиотеку баркодинговых последовательностей новыми качественными данными. Вероятно, можно говорить о появлении более специализированных баз данных последовательностей ДНК, сгруппированных не по таксономическому, а по экологическому признаку (например, для зоопланктона) (Machida et al., 2009). Привлечение других методов молекулярной биологии (например, ДНК-микрочипирование) позволит сделать процедуру определения видов морской фауны быстрой и более корректной (Forêt et al., 2007). По мере накопления генетических данных для массовых видов беспозвоночных открывается перспектива проведения мониторинга морских биотопов для исследования структуры природных сообществ. Вместе с тем, ДНК-баркодинг не приходит на замену морфологического метода определения видов морских беспозвоночных, а лишь является его дополнением. ЛИТЕРАТУРА 1. Кусто Ж.-И., Д. Даген, 1976. Живое море. Пер. с англ. - М.: Мысль. - 432с. 2. Barber P., S.L. Boyce, 2006. Estimating diversity of Indo-Pacific coral reef stomatopods through DNA barcoding of stomatopod larvae // Proc. R. Soc. Lond., B, Biol. Sci. 273: 20532061. 3. Batta-Lona P.G., A. Bucklin, P.H. Wiebe, T. Patarnello, N.J. Copley, 2011. Population genetic variation of the Southern Ocean krill, Euphausia superba, in the Western Antarctic Peninsula region based on mitochondrial single nucleotide polymorphisms (SNPs) // Deep Sea Research. Part II. 58: 1652-1661. 4. Blankenship L.E., A.A. Yayanos, 2005. Universal primers and PCR of gut contents to study marine invertebrate diets // Molecular Ecology 14: 891-899. 5. Bucklin A., M. Guarnieri, R.S. Hill, A.M. Bentley, S. Kaartvedt, 1999. Taxonomic and systematic assessment of planktonic copepods using mitochondrial COI sequence variation and competitive, species-specific PCR // Hydrobiologia 401: 239-254. 6. Bucklin A., P.H. Wiebe, S.B. Smolenack, N.J. Copley, J.G. Beaudet, K.G. Bonner, J. FarberLorda, J.J. Pierson, 2007. DNA barcodes for species identification of euphausiids (Euphausiacea, Crustacea) // J. Plankton Res. 29: 483-493. 7. Bucklin A., D. Steinke, L. Blanco-Bercial, 2011. DNA barcoding of marine metazoa // Annu. Rev. Mar. Sci. 3:471-508. 8. Costa F.O., J.R. deWaard, J. Boutillier, S. Ratnasingham, R.T. Dooh, M. Hajibabaei, P.D.N. Hebert, 2007. Biological identifications through DNA barcodes: the case of the Crustacea // Can. J. Fish. Aquat. Sci. 64: 272-295. 9. Farêt S., K. Kassahn, L. Grasso, D. Hayward, A. Iguchi, E. Ball, D. Miller, 2007. Genomic and microarray approaches to coral reef conservation biology // Coral Reefs 26(3): 475-486. 10. Folmer O., M. Black, W. Hoeh, R. Lutz, R. Vrijenhoek, 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase I from diverse metazoan invertebrates // Mol. Mar. Biol. Biotechnol. 3: 294-299. 11. Hebert P.D.N., A. Cywinska, S.L. Ball, J.R. deWaard, 2003. Biological identifications through DNA barcodes // Proc. R. Soc. Lond. B. 270: 313-321. 12. Huang D., R. Meyer, P.A. Todd, L.M. Chou, 2008. Slow mitochondrial COI sequence evolution at the base of the metazoan tree and its implications for DNA barcoding // J. Mol. Evol. 66: 167174. 13. Jennings R.M., A. Bucklin, H. Ossenbrügger, R.R. Hopcroft, 2010. Species diversity of planctonic gastropods (Pteropoda and Heteropoda) from six ocean basins based on DNA barcode analysis // Deep-Sea Research. Part II. 57: 24-26. 14. Kemppainen P., M. Panova, J. Hollander, K. Johannesson, 2009. Complete lack of mitochondrial divergence between two species of NE Atlantic marine intertidal gastropods // J. Evol. Biol. 22: 2000-2011. 15. Leung T.L.F., K.M. Donald, D.B. Keeney, A.V. Koehler, R.C. Peoples, R. Poulin, 2009. Trematode parasites of Otago Harbour (New Zealand) soft-sediment intertidal ecosystems: life cycles, ecological roles and DNA barcodes // N.Z. J. Mar. Freshwater Res. 43: 857-865. 16. Machida R.J., Y. Hashiguchi, M. S. Nishida, 2009. Zooplancton diversity analysis through using single-gene sequencing of a community sample // BMC Genomics 10: 438. 17. Meyer C.P., G. Paulay, 2005. DNA barcoding: error rates based on comprehensive sampling // PLoS Biology 3(12): e422. 18. Plaisance L., N. Knowlton, G. Paulay, C. Meyer, 2009. Reef-associated crustacean fauna: biodiversity estimates using semi-quantitative sampling and DNA barcoding // Coral Reefs 28: 977-986. 19. Radulovici A.E., B. Sainte-Marie, F. Dufresne, 2009. DNA barcoding of marine crustaceans from the Estuary and Gulf of St. Lawrence: a regional-scale approach // Mol. Ecol. Resour. 9: 181-187. 20. Radulovici A.E., P. Archambault, F. Dufresne, 2010. DNA barcodes for marine biodiversity: Moving fast forward? // Diversity 2: 450-472. 21. Sidall M.E., F.M. Fontanella, S.C. Watson, S. Kvist, C. Erséus C., 2009. Barcoding bamboozled by Bacteria: convergence to metazoan mitochondrial primer targets by matine microbes // Syst. Biol. 58: 445-451. 22. Song H., J.E. Buhay, M.F. Whiting, K.A. Crandall, 2008. Many species in one: DNA barcoding overestimates the number of species when nuclear mitochondrial pseudogenes are coamplified // Proc. Natl. Acad. Sci. USA 105: 13486-13491. 23. Ward R.D., T.S. Zemlak, B.H. Innes, P.R. Last, P.D.N. Hebert, 2008. DNA barcoding descriminates echinoderm species // Mol. Ecol. Resour. 8: 1202-1211. ПРИЛОЖЕНИЕ Список международных проектов по созданию баркодинговых библиотек The Consortium for the Barcode Life (CBOL) http://barcoding.si.edu The Barcode of Life Datasystems (BOLD) http://www.boldsystems.org The International Barcode of Life (iBOL) http://www.ibol.org CBOL Fungal Working Group http://www.fungalbarcoding.org/ Bee Barcode of Life Initiative (Bee-BOL) http://www.bee-bol.org/ Coral Reef Barcode of Life http://www.reefbarcoding.org/ European Cosortium for the Barcode of Life (ECBOL) http://www.ecbol.org/ Fish Barcode of Life Compaign (FISH-BOL) http://www.fishbol.org/ Health BOL http://www.healthbol.org/ Lepidoptera Barcode of Life http://lepbarcoding.org/ Mammalia Barcode of Life Сampaign http://www.mammaliabol.org/ Marine Barcode of Life (MarBol) http://www.marinebarcoding.org/ Moorea Biocode Project http://mooreabiocode.org/ Polar Barcode of Life (PolarBol) http://www.polarbarcoding.org/ Quarantine Barcode of Life (QBOL) http://www.qbol.org/UK/ Shark Barcode of Life (SharkBOL) http://www.sharkbol.org/ Sponge Barcoding Project (SpongeBOL) http://www.spongebarcoding.org/ Trichoptera Barcode of Life (TrichopteraBOL) http://trichopterabol.org/ ъ Е. Урюпова Кафедра зоологии беспозвоночных Биологического факультета МГУ им. М.В. Ломоносова Материал подготовлен в ходе выполнения проекта Министерства Образования и Науки (соглашение 8334)