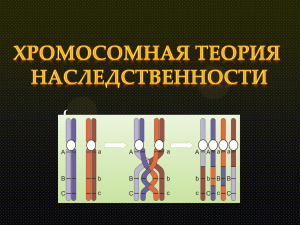
Оптимизация физического картирования генов с помощью
advertisement

ФГБОУ ВО РОССИЙСКИЙ ГОСУДАРСТВЕННЫЙ АГРАРНЫЙ УНИВЕРСИТЕТ – МСХА ИМЕНИ К.А. ТИМИРЯЗЕВА _____________________________________________________________________ На правах рукописи КИРОВ ИЛЬЯ ВЛАДИМИРОВИЧ РАЗРАБОТКА И ПРИМЕНЕНИЕ ВЫСОКОЧУВСТВИТЕЛЬНОГО МЕТОДА ФИЗИЧЕСКОГО КАРТИРОВАНИЯ ГЕНОВ НА ХРОМОСОМАХ РАСТЕНИЙ Специальность 03.02.07 – генетика Диссертация на соискание ученой степени кандидата биологических наук Научный руководитель – доктор биологических наук, профессор Хрусталева Л.И. Москва - 2015 2 Оглавление ВВЕДЕНИЕ ...................................................................................................................... 5 ГЛАВА 1. ОБЗОР ЛИТЕРАТУРЫ .............................................................................. 13 1.1 Физические и генетические карты растений ........................................................ 13 1.1.1 Достоинства и недостатки физические и генетических карт ....................... 13 1.1.2. Интегрированные физические и генетические карты.................................. 16 1.1.3 Картирование генов на хромосоме растений ................................................. 18 1.2 Создание физических карт растений..................................................................... 20 1.2.1 Подходы к физическому картированию геномов растений ......................... 21 1.2.2 FISH – главный инструмент физического картирования хромосом растений .................................................................................................... 23 1.2.3 Физическое картирование хромосом растений с помощью Tyramide-FISH........................................................................................ 26 1.3 Основные факторы успешного физического картирования генов с помощью FISH ................................................................................................................................ 28 1.3.1 Препараты хромосом ........................................................................................ 28 1.3.2 Системы детекции сайтов гибридизации ....................................................... 35 1.3.3. Цитогенетические маркеры хромосом растений .......................................... 35 1.4 Принцип Tyramide-FISH ......................................................................................... 38 1.4.1 Реакция тирамид с пероксидазой хрена ......................................................... 39 1.4.2. Способы модификации реакции для улучшения детекции антигена на мембране и в пробирке .............................................................................................. 41 1.4.3 Принцип Tyramide-FISH .................................................................................. 42 1.4.4 Системы детекции Tyramide-FISH .................................................................. 43 1.5 Характеристика объектов исследования............................................................... 48 1.5.1 Представители рода Allium .............................................................................. 48 1.5.2 Rosa wichurana ................................................................................................... 54 ГЛАВА 2. МАТЕРИАЛЫ И МЕТОДЫ ...................................................................... 58 2.1 Растительный материал .......................................................................................... 58 2.2 Приготовление препаратов хромосом .................................................................. 58 2.2.1 Митотические хромосомы ............................................................................... 58 3 2.2.2 Пахитенные хромосомы ................................................................................... 60 2.3 Выделение ДНК ....................................................................................................... 60 2.4 Получение проб для Tyramide-FISH и FISH......................................................... 61 2.5 FISH и ND-FISH (nondenaturating FISH) ............................................................... 64 2.6 Tyramide-FISH ......................................................................................................... 64 2.6.1 Прямой метод детекция.................................................................................... 64 2.6.2 Непрямой метод детекции ............................................................................... 65 2.6.3 Многоцветная Tyramide-FISH ......................................................................... 65 2.7 Флуоресцентная микроскопия и анализ изображения ........................................ 66 2.8 Изолирование повтора HAT58 ............................................................................... 66 2.9 Генетическое картирование ................................................................................... 66 ГЛАВА 3. РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ ............................................................ 68 3.1 SteamDrop – новый универсальный метод приготовления препаратов хромосом растений........................................................................................................ 68 3.1.1 Процесс разброса хромосом растений ............................................................ 68 3.1.2 Относительная влажность воздуха и температура – критические факторы разброса хромосом растений .................................................................................... 71 3.1.2 Метод «SteamDrop» («паровая капля») .......................................................... 74 3.2 Оптимизация Tyramide-FISH ................................................................................. 86 3.2.1 Оптимизация системы детекции ..................................................................... 86 3.2.1.1 Оптимизация прямой и непрямой систем детекции для физического картирования генов на мелких хромосомах R.wichurana ...................................... 86 3.2.1.2 Оптимизация прямой и непрямой систем детекции для физического картирования генов на крупных хромосомах Allium cepa..................................... 90 3.2.2 Химический синтез коньюгатов тирамид и их применение в Tyramide-FISH детекции на хромосомах Allium и R.wichurana ........................ 94 3.2.3 Влияние приготовления препаратов хромосом на результаты Tyramide-FISH ........................................................................................ 96 3.3 Поиск цитогенетичеких маркеров ....................................................................... 101 3.3.1 Поиск цитогенетических маркеров для хромосом Allium .......................... 102 3.3.2 Поиск цитогенетичесих маркеров для хромосом Rosa wichurana............. 106 4 3.4 Физическое картирование генов с помощью оптимизированных протоколов и построение интегрированных генетических и физических карт ........................... 111 3.4.1 Allium ................................................................................................................ 111 3.4.2 Rosa wichurana ................................................................................................ 120 ЗАКЛЮЧЕНИЕ ........................................................................................................... 133 ВЫВОДЫ ..................................................................................................................... 135 СПИСОК ЛИТЕРАТУРЫ........................................................................................... 137 5 ВВЕДЕНИЕ Актуальность. Положение гена на хромосоме играет важную роль в изменении признаков и эволюции организмов (Rockman et al., 2010). Знания о физическом положении генов на хромосоме позволяют прогнозировать возможность их переноса от одного генотипа к другому, ускорять процесс поиска и клонирования новых генов, судить о коллинеарности и синтении геномов, определять размеры селекционной популяции, время и экономические затраты на получение нужных форм. Геномы лишь небольшого числа растений были полностью секвенированы и собранны, предоставляя наиболее полную информацию о физическом положении генов. Для подавляющего большинства видов растений полный сиквенс генома пока остается недоступным. Более того, часто конечный вариант сиквенса генома имеет неточности в положении контигов и скэффолдов. Последние исследования показали, что 30% сравнительно небольшого генома томата (Solanum lycopersicum, 919 млн.п.н.), секвенированного при участии ученых из 14 стран, собрано неверно (Shearer et al., 2014). Эти неточности в de novo сборке геномов растений связаны с использованием генетических карт для упорядочивания контигов и скэффолдов. Генетические карты не показывают физическое положение генов/маркеров на хромосомах, а лишь предоставляют информацию о частоте рекомбинации между маркерами. Кроме этого, распределение точек рекомбнации вдоль хромосомы крайне неравномерно и один процент рекомбинации (сантиМорган, сМ) может быть эквивалентен физическому расстоянию от 10 тыс.п.н. до 100 млн.п.н. (Kunzel et al. 2000; Wu et al. 2003; Werner et al. 1997). В связи с этим, возможность прямой визуализация генов/контигов/скэффолдов/маркеров на физической хромосоме представляет практическую ценность и большой интерес как исследований, так и практической селекции растений. для фундаментальных 6 Методы молекулярной цитогенетики позволяют визуализировать и физически картировать на хромосомах различные ДНК последовательности. Физическое картирование коротких ДНК фрагментов, включая гены и маркеры, с помощью методов успешного коротких цитогенетики физического ДНК (молекулярно- фрагментов высококачественные затруднительно. на препараты Главные составляющие цитогенетического) картирования хромосомах хромосом; 2) растений система являются: детекции 1) сайтов гибридизации; 3) качество ДНК пробы, которая гибридизуется только в одном локусе; и 4) цитогенетические маркеры для идентификации индивидуальных хромосом. При этом определяющим фактором остаѐтся качество приготовленных препаратов хромосом, которое определяет 70% успеха физического картирования. Однако, современные методы приготовления препаратов требуют большого опыта и очень трудоѐмки, что делает процесс приготовления препаратов «больше искусством, чем наукой» (Barch et al., 1997). Флуоресцентная in situ гибридизация (FISH) – основной метод молекулярной цитогенетики. Главным недостатком FISH является низкая чувствительность, 5 – 10 тыс.п.н. (Valarik et al., 2004). В то время как средняя длина генов растений меньше 4 тыс.п.н. (Jaillon et al. 2007; Ling et al. 2013; Rice Chromosome 10 Sequencing Consortium., 2003). Для преодоления этого ограничения FISH был создан метод Tyramide-FISH (Raap et al. 1995), который позже был впервые применен для хромосом растений (Khrustaleva and Kik, 2001). Однако, из-за низкой частоты встречаемости сигнала по хромосомам и низкой воспроизводимости результатов в разных лабораториях и на разных видах растений (Jiang and Gill, 2006), Tyramide-FISH не был широко использован на хромосомах растений. Главными причинами этого являются ограниченные знания о факторах, влияющих на реакцию Tyramide-FISH, и отсутствие работ по оптимизации данного метода на хромосомах растений. Важным фактором в успешном физическом картировании генов с помощью методов цитогенетики является создание специфичных ДНК проб. Современные 7 технологии открывают целый набор инструментов для дизайна ДНК проб (зондов), не ограничиваясь в выборе только клонами из созданных ВАС, YAC, EST и других типов библиотек. Методы биоинформатики и существующие базы данных позволяют на своей платформе искать целевые ДНК последовательности in silico и разрабатывать специфичные ДНК зонды для физического картирования. Кроме этого, возможность поиска ДНК повторов внутри сиквенсов позволяет идентифицировать уникальные части последовательностей и подбирать локусспецифичные зонды (Schmutzer et al.; 2014). Сочетание методов биоинформатики и молекулярной цитогенетики позволяют проводить физическое картирование геномов растений на качественно новом уровне. Физическое картирование хромосом затруднительно без знания кариотипа растения и возможности проводить идентификацию отдельных хромосом кариотипа. Морфологические признаки хромосом (центромерный индекс и относительная длина) предоставляют ограниченную информацию, которая для многих растений (часто растений с мелкими хромосомами) не позволяет эффективно отличать хромосомы кариотипа. В связи с этим актуальной является задача поиска новых цитогенетических маркеров хромосом растений. Среди всех извесных типов цитогенетических маркеров FISH-маркеры хромосом растений открывают широкие возможности для специфичной идентификации хромосом. Сочетание чувствительной системы детекции сайтов гибридизации, методов приготовления качественных препаратов хромосом растений и ДНК проб совместно с использованием FISH-маркеров для идентификации хромосом растений формируют необходимую методологическую платформу для эффективного физического картирования генов на хромосома растений. Степень разработанности темы Теме физического картирования генов с помощью методов цитогенетики посвящены немногочисленные статьи и частично обзоры литературы, опубликованные в международных высокорейтинговых журналах. Однако, остаѐтся большое число методологических трудностей и вопросов, которые 8 препятствуют широкому использованию отдельных генов в качестве пробы для физического картирования с помощью FISH. К таким слабо изученным сторонам данной темы относятся проблемы приготовления препаратов хромосом растений, оптимизация системы детекции сайтов гибридизации и трудности идентификации хромосом растений. В связи с этими, оптимизация и изучение ключевых этапов физического картирования и практическое использование этого метода, особенно, на слабоизученных видах, является актуальной темой исследования. Цель работы: Повысить эффективность Tyramide-FISH, создать необходимую платформу (система детекции + метод приготовления препаратов + система цитогенетических маркеров) для физического картирования коротких фрагментов генов (1000 – 3000 п.н.), применить эту технологию на хромосомах видов Allium и Rosa wichuranа, создать физические (хромосомные) карты и провести интегрирование физических и генетических карт этих видов. Задачи: 1. Создать высокоэффективный и универсальный метод приготовления митотических метафазных и пахитенных хромосом растений c использованием видов с крупными геномами и хромосомами (виды рода Allium) и видов с мелкими хромосомами и малым размером генома (виды рода Rosa) 2. Провести клонирование фрагментов генов Rosa wichurana и Allium cepa длиной не менее 550 п.н. 3. Оценить влияние способа приготовления препаратов c использованием разных концентраций уксусной кислоты на частоту встречаемости сигнала после Tyramide-FISH с геном аллиназы корня 1 (1100 п.н.) на хромосомах A. cepa. 4. Оптимизировать и сравнить прямую и непрямую системы детекции TyramideFISH на хромосомах A.cepa и R.wichurana. 5. Провести оптимизацию высокоразрешающей и многоцветной Tyramide-FISH на хромосомах R.wichurana. 9 6. Провести картирование генов (гены семейства аллиназ и ген синтазы фактора слезотечения), вовлеченных в синтез сульфурорганических соединений, на хромосомах A.cepa, A.fsitulosum и их близкородственных видов (A.vavilovii, A.schoenoprasum, A.altaicum). 7. Провести Tyramide-FISH картирование генов, вовлеченных в ответ растения на биотический и абиотический стрессы, на митотических и пахитенных хромосомах R.wichurana. 8. Провести генетическое картирование генов R.wichurana c помощью метода высокоразрешающего плавления (HRM). 9. Провести интегрирование рекомбинационной и хромосомной карт A.cepa и R.wichurana. 10. Провести поиск молекулярно-цитогенетических маркеров для хромосом R.wichurana и A.fsitulosum с использованием данных секвенированния следующего поколения и биоинформатического анализа, и FISH выявления специфических патернов гибридизации. Научная новизна Автором впервые изучена динамика разброса хромосом растений в процессе приготовления препаратов и установлены факторы, влияющие на структуру и разброс хромосом. Впервые показано, что эффективность TyramideFISH зависит от концентрации уксусной кислоты, используемой для приготовления препаратов хромосом. Идентифицированы повторяющиеся ДНК последовательности и на их основе разработаны эффективные системы цитогенетического маркирования хромосом A.fistulosum (новый повтор HAT58, AfiT32, 5S рДНК) и R.wichurana (5S рДНК, 45S рДНК и теломерный повтор (GGGATTT)n). Впервые проведено интегрирование генетической и физической карт R.wichurana. С помощью Tyramide-FISH впервые показаны различия в гомеологичных хромосомах 4 A.cepa и A.fistulosum, перицентрической инверсией и транслокацией/делецией. обусловленные 10 Теоретическая и практическая значимость работы Разработанный высокочувствительнй метод позволит ускорить физическое картирования генов и интегрирование генетических и физических карт исследуемых видов. Данные результаты являются ценной информацией для проектов по секвенированию генома A.cepa (http://www.oniongenome.net/) и Rosa (Foucher et al. 2013), так как позволят ускорить и верифицировать сборку геномов. Эта информация послужит важной основой для изучения синтении и коллинеарности между близкими видами внутри рода Allium и семейства Rosaсeae, а также позволит более эффективно проводить клонирование генов, основанное на карте (от анг. map based cloning). Так же разработанный протокол многоцветной Tyramide-FISH с высоким разрешением позволяет существенно расширить возможности метода Tyramide-FISH. Разработанный метод приготовления препаратов митотических метафазных и пахитенных хромосом (―SteamDrop‖) был успешно испытан на 30 видах растений, что указывает на его высокую адаптивность и возможность использования для многих видов растений. Метод имеет ряд преимуществ, таких как хороший разброс хромосом, мягкий режим обработок, максимально сохранящий структуру хромосом, простота и быстрота приготовления препаратов, долгое хранение суспензии клеток без потерь качества препаратов. Разработанная система цитогенетических маркеров для A.fistulosum, основанная на FISH c новым тандемным повтором HAT58, а также AfiT32 и 5S рДНК, и для R.wichurana, основанная на FISH с 5S рДНК, 45S рДНК и (GGGATTT)8 теломерным повтором, делает возможным высокоразрешающее физическое картирование на мейотических хромосомах, идентификация которых у луков и роз невозможна без использования молекулярно-цитогенетических маркеров. Методология и методы диссертационного исследования Работа выполнена с использованием современного оборудования и молекулярно-биологических и молекулярно-цитогенетических методов, 11 разработанных ведущими учеными в мире. Результаты статистически обработаны. Положения, выносимые на защиту 1. Метод ―SteamDrop‖ – универсальный и эффективный метод приготовления хромосом растений, подходящих для in situ детекции уникальных и повторяющихся последовательностей ДНК 2. Использование пониженной концентрации уксусной кислоты для приготовления препаратов существенно повышает эффективность физического картирования генов Tyramide-FISH. 3. Фрагменты генов длиной от 1000 п.н. могут быть эффективно картированы на хромосомах R.wichurana и видов Allium, приготовленных с помощью ―SteamDrop‖, используя оптимизированные системы детекции Tyramide-FISH. 4. HAT58 – новый высококопийный (160.000 копий/1C) тандемный повтор генома A.fistulosum. 5. Созданы системы цитогенетических маркеров для хромосом R.wichurana и A.fistulosum. 6. С помощью Tyramide-FISH проведено картирование хозяйственно-ценных генов на крупных хромосомах A.cepa, A.fsitulosum, A.vavilovii, A.schoenoprasum, A.altaicum и мелких хромосомах R.wichurana. 7. Проведено интегрирование генетических и физических карт A.cepa и R.wichurana. 8. Инверсия участка хромосомы привела к различиям между гомеологичными хромосомами 4 A.cepa и A.fsitulosum. Степень достоверности и апробация результатов Результаты работы опубликованы в 35 печатных работах, в том числе 11 статьях в рецензируемых международных (8) и отечественных (3) журналах из списка ВАК. Ключевые результаты работы были доложены на международных и российских форумах и конференциях, в том числе: XI молодежная научная 12 конференция «биотехнология в растениеводстве, животноводстве и ветеринарии» (Москва, 2011); XVIII международная научная конференция студентов, аспирантов и молодых учѐных «Ломоносов» (Москва, 2011 г.); 35th conference of agricultural students and veterinary medicine with international participation (Сербия, 2011); XIV растениеводстве, молодежная научная животноводстве и конференция ветеринарии» «биотехнология (Москва, 2011); в 2-я Международная школа-конференция молодых учѐных (Звенигород, 2011 г.); 6th International symposium on Edible Alliaceae (Япония, 2012 г.); 19th International Chromosome Conference, (Италия, 2013); Journées Cytogénétique et Polyploïdie, (Франция, 2013); 7th International symposium on Edible Alliaceae (Турция, 2015 г.); Plant molecular cytogenetics in genomic and postgenomic era (Польша, 2014), 3-я Всероссийская научно-практическая конференция по геномному секвенированию (NGS-2015, Москва, 2015); XXVth International Symposium of the EUCARPIA Section Ornamentals ―Crossing borders‖ (Бельгия, 2015). 13 ГЛАВА 1. ОБЗОР ЛИТЕРАТУРЫ 1.1 Физические и генетические карты растений 1.1.1 Достоинства и недостатки физические и генетических карт Генетические карты показывают порядок маркеров относительно друг друга в группе сцепления. Расстояние между маркерами измеряется в процентах рекомбинации, или сантиМорганах (сМ). Начиная с 1913 года, когда была создана первая генетическая, или рекомбинационная карта Стѐртевантом (Sturtevant 1913, 1965), были сконструированы генетические карты для человека, многих животных и растений. Переход от довольно ограниченного полиморфизма морфологических признаков/мутантов и изоферментов (Brown, 1978) к использованию высополиморфных нейтральных ДНК маркеров (Poczai P. et al. 2013) расширяет возможности генетического картирования на многих видах растений. Конструирование подробных генетических карт с высоким уровнем покрытия генома является важным шагом на пути к применению молекулярных маркеров в селекции растений (маркер опосредованная селекция, МОС). Кроме этого сегодня генетические карты используются для: а) Детального генетического анализа качественных и количественных признаков, что позволяет локализовать гены или локусы количественных признаков (QTL) и провести их характеристику относительно эффекта на фенотип, плейотропии и эпистатических эффектов (Xiao et al. 1996; Mohan et al. 1997; Doerge , 2002; Yim et al. 2002) . б) Интрогрессии полезных генов или QTLs с помощью МОС (Dufey et al. 2015; Yohannes T. et al. 2015. в) Установления связи между генами и их функциями (Ahn et al. 1993; Paterson et al. 2002). г) Интегрирования с физическими картами (Yim et al. 2002; Kirov et al. 2014; Ariyadasa et al. 2014). д) Выделения и клонирования генов (Mohan et al. 1997; Mickelson-Young et al. 1995; Shimizu et al. 2015). 14 е) Сборки геномов (Ariyadasa et al. 2014; Liu et al. 2014; Deokar A. A. et al.2014; Dohm J. C. et al. 2014; Argyris et al. 2015; Wu P. et al. 2015) Генетическая карта должна отвечать техническим и методологическим критериям, таким как простота, надежность, и экономическая эффективность (Gill et al. 1996). Однако генетические карты имеют ряд недостатков, существенно затрудняющих их использование в сравнительной геномике и селекции растений. Во-первых, генетические карты не дают представления о действительном физическом расстоянии между генами. Гены могут быть физически удалены друг от друга на сотни миллионов пар оснований ДНК, но на генетической карте они могут стоять близко, например, в прицентромерных регионах, где рекомбинации почти не возникают (Sun et al. 2013). На сегодня достоверно известно, что один сМ может представлять на физической хромосоме расстояние от нескольких десятков тысяч до миллионов пар нуклеотидов (Schmidt et al. 1995; Gill et al. 1996; Werner et al. 1997; Kunzel et al. 2000; Wu et al. 2003; Blenda A. et al. 2012; Sun et al. 2013; Ariyadasa et al. 2014). Область супрессии рекомбинации может занимать половину хромосомы (Kunzel et al. 2000; Mayer et al. 2011), делая генетическое картирование в этих регионах малоэффективным. Во-вторых, положение маркеров на генетической карте сильно зависит от картируемой популяции, числа использованных маркеров и родительских форм. Одни и те же генетические маркеры могут быть локализованы в разных местах и даже группах сцепления в генетических картах, созданных на разных популяциях (Xu et al. 2008). Это серьезно затрудняет клонирование генов, основанное на положении в генетической карте и теоретические исследования по эволюции хромосом. Втретьих, события рекомбинации неравномерно распределены вдоль хромосом, поэтому генетические карты имеют низкое разрешение в местах супрессии рекомбинации, таких как гетерохроматин и прицентромерная область (Tanksley et al. 1992). Это приводит к неточностям в сборке геномов и усложняет клонирование генов (Karafiátová et al. 2013; Yang L. et al. 2012; Shearer et al. 15 2014). Так, например, 30% генов пшеницы находятся в местах супрессии рекомбинации, где общая частота рекомбинации всего 1сМ (Erayman et al. 2004). В отличие от генетических карт, расстояние между маркерами в физических картах обычно выражено в парах нуклеотидов для секвенированных геномов, и для цитогнетических карт, которые также можно отнести к физическим картам, в расстоянии от центромеры до маркера, выраженном в процентах или в микрометрах с возможным пересчетом в пары нуклеотидов, полагая одинаковую плотность компактизации хромосомы по всей длине. Физические карты полностью секвенированных и собранных геномов - очень информативны и, как правило, содержат большую часть последовательностей ДНК всего генома. Главным недостатком физических карт секвенированных геномов часто является неверное физическое положение контигов и скэффолдов в собранных псевдохромосомах. Данный недостаток является следствием сложности организации генома растений, который содержит множество повторяющихся ДНК последовательностей, как кодирующих (псевдогены, геныпаралоги, тандемно организованные гены (гены рРНК)), так и некодирующих (сателитная ДНК, мобильные элементы и т.д.). Кроме этого, геномы растений имеют высокое число дупликаций, а многие сельскохозяйственные растения являются аллополиплоидами. Большинство неточностей в сборке геномов растений локализованы в местах с низкой частотой рекомбинации и/или с высокой плотностью повторяющихся последовательностей ДНК (центромерная область, гетерохроматин, субтеломерная область). Связь между качеством сборки генома и распределением точек рекомбинации по геному обусловлена необходимостью использовать генетические карты, насыщенные маркерами, для выстраивания контигов («заякоривание»). Для преодоления трудностей сборки геномов, основанной на генетических картах, должны быть использованы другие подходы для картирования, не зависящие от частоты мейотической рекомбинации. Один из таких подходов – это построение карт при помощи панели радиационных гибридов (Riera-Lizarazu O. et al. 2000; Kalavacharla V. et al. 16 2006; Kumar A. et al. 2014; Kobayashi et al. 2015). Этот подход позволяет построить карты, в которых расстояние между маркерами выражено в санти Рейах (centi Rays (cR)). 1сR – это один разрыв хромосомы на 100 линий. Распределение частоты образования хромосомных разрывов происходит более равномерно, чем распределение частоты мейотической рекомбинации. Хотя карты, построенные при помощи панелей радиационных гибридов, могут быть использованы для улучшения сборки генома, они не являются физическими, т.к. не показывают реального физического расстояние между генами. Установить точное положение на хромосоме и определить физическое (выраженное в парах нуклеотидов) расстояние между ДНК последовательностями, не используя генетические карты и панели радиационных гибридов, позволяют цитогенетические карты и молекулярно-цитогенетические методы. В отличие от карт секвенированных геномов, цитогенетические карты карты, как правило, картированных содержат существенно последовательностей, что меньшее обусловлено число физически трудоѐмкостью их создания. Несомненными достоинствами цитогенетических карт являются: (1) они показывают реальное и достоверное положение ДНК последовательностей на физической хромосоме; (2) позволяют проводить физическое картирование относительно важных хромосомных участков (гетерохроматин, эухроматин, центромера, теломера); (3) короткое время создания и относительно низкая цена; (4) не требуют создания специальных коллекций растений с хромосомными нарушениями; (5) успех физического картирования с помощью методов цитогенетики (FISH) не зависит от положения ДНК последовательности в геноме, что позволяет проводить картирование в местах супрессии рекомбинации, и в участках генома, насыщенных повторяющиемися последовательностями ДНК. 1.1.2. Интегрированные физические и генетические карты Приведенные достоинства и недостатки генетических и физических карт указывают, что эти два типа карт могут дополнять друг друга при их 17 интегрировании. Построение интегрированных карт геномов растения может ускорить процесс секвенирования геномов и точнее проводить сборку геномов, а так же аннотирование повторяющихся последовательностей генома. Комбинирование данных из генетических карт, карт секвенированных геномов и цитогенетических карт не только позволяет упорядочить и интегрировать генетическую, молекулярную и цитологическую информацию, но и способствуют лучшему пониманию организации генома в контексте хромосом (Islam-Faridi et al. 2002). Интегрирование генетических и цитогенетических карт карт проводится для (а) установления распределения событий рекомбинаций вдоль физической хромосомы; (б) установления физического расстояния между генетически картированными маркерами; (в) определения порядка генов/маркеров в местах супрессии рекомбинации; (г) привязки («заякоривание») групп сцепления индивидуальным хромосомам; (д) установление физического к положения маркеров/генов/QTL относительно мест генома с высокой и низкой частотой рекомбинации для оценки эффективности индивидуального переноса генов/признаков между сортами. Молекулярные карты сцепления быстро развиваются с добавлением новых типов маркеров, слиянием разных видоспецифичных карт и сравнительным отображением полученных маркеров карт, между требует геномами. детального Эффективное понимания использование взаимосвязи между генетическим и физическим расстоянием. Например, для сельскохозяйственных растений с крупными геномами, успех позиционного клонирования (клонирование по карте) генов зависит от возможности определения геномных регионов, в которых физическое расстояние между связанными локусами достаточное для его преодоления молекулярно-биологическими методами (Tanksley et al. 1992). Различные стратегии могут быть использованы для соотношения генетического расстояния к физическому. Для гексаплоидной пшеницы была разработана система получения терминальных делеций хромосом (Endo, 1988; Endo and Gill, 1996). Используя определенные делеции, были 18 получены результаты по распределению маркеров и событий рекомбинации на субрегионах хромосомы (Werner et al. 1992; Hohmann et al. 1994; Delaney et al. 1995; Gadaleta et al. 2014). Кроме того, подавление рекомбинации в проксимальных участках хромосом и частая рекомбинация в дистальных участках хромосом, локальные горячие точки рекомбинации и плотности генов были определены для небольших участков хромосом (Gill et al. 1993; 1996). Однако, изза трудностей в получении делеционных линий для диплоидных видов растения и отсутствия хромосомных маркеров для детекции хромосомных реорганизаций, такие линии не могут быть созданы для многих видов культурных растений (например, ячмень и лук) (Howell et al. 2002). Так как создание цитогенетических карт проще, чем физических карт секвенированных геномов, то наиболее часто проводят интегрирование именно генетических и интегрирования цитогенетических генетических и карт. Самым цитогенетических эффективным карт является методом прямая визуализация маркера/гена на хромосоме с помощью FISH (Jiang and Gill, 2006). За последние 5 лет интегрирование FISH-основанных цитогенетических карт с генетическим картами было проведено для целого ряда видов, включая Brassica rapa (Xiong et al. 2000), Allium cepa (Masamura et al. 2012; Романов, 2012); Cucumis sativus (Han et al. 2011; Lou et al. 2013), Zea mays (Figueroa and Bass, 2012), Gossypium hirsutum (Wang et al. 2010), Gossypium herbaceum (Cui et al. 2015), Phaseolus vulgaris (Fonsêca et al. 2010), Vicia faba (Ruiz-Rodriguez et al. 2014), Beta vulgaris (Paesold S. et al. 2012), Brachypodium distachyon (Febrer et al. 2010), Lotus japonicus (Ohmido et al. 2010), Carica papaya (Zhang et al. 2010; Wai et al. 2012), Rosa wichurana (Kirov et al. 2014, 2015a, 2015b). 1.1.3 Картирование генов на хромосоме растений Физическое картирование отдельных генов растений с помощью FISH является перспективным направлением в молекулярной цитогенетике растений (Jiang and Gill, 2006), т.к. позволяет напрямую визуализировать гены на хромосоме, сравнивать их положение между видами и проводить интегрирование 19 генетических и физических карт значительно быстрее. Результаты многочисленных работ посвященных физическому картированию генов с помощью FISH свидетельствуют, что порог чувствительности для обычной FISH составляет 3000 – 10000 п.н. (Jiang and Gill, 2006; Lamb et al. 2007; Danilova and Birchler, 2008; Karafiátová et al. 2013; Danilova et al. 2014). FISH с ДНК пробами, длина которых меньше 3000 п.н., дает низкую частоту встречаемости сигналов и трудно воспроизводима (Jiang and Gill, 2006). За редким исключением, как правило, работы по FISH картированию коротких фрагментов ДНК проводятся на растениях (кукуруза, ячмень, пшеница, лук), для которых имеются хорошо отработанные цитогенетические протоколы. Достижение положительных результатов в картировании коротких фрагментов на хромосомах других видов растений очень проблематично. Можно выделить несколько факторов, затрудняющих возможность визуализации небольших фрагментов ДНК на хромосомах растений. Первый фактор - это трудности в приготовлении высококачественных препаратов хромосом. Это связано, прежде всего, с наличием клеточной стенки, затрудняющей получение препаратов с малым содержанием цитоплазмы и высокой доступностью хроматина для гибридизации. Цитоплазма снижает доступность хроматина для гибридизации и препятствует прохождению ДНК пробы к ДНК хромосомы. Второй фактор заключается в плотной компактизации хроматина в хромосомах растений. Оба фактора увеличивают интенсивность фона, снижая отношение интенсивности сигнала к фону. Но, несмотря на это, на сегодня предпринято множество попыток картирования коротких последовательностей ДНК. При этом применяются несколько подходов: (1) использование специальной CCD (cooled charge coupled device camera) камеры, позволяющей увеличить интенсивность сигнала в 30 раз (Khrustaleva and Kik, 2001); (2) PRINS (primed in situ labeling) метод (Koch et al. 1989; Kubaláková et al. 2001); (3) создание пробы с помощью ник-трансляции, содержащей флуорохроммеченные нуклеотиды и повышенную концентрацию ДНК полимеразы I E.coli 20 (Kato et al. 2006) и (4) Tyramide-FISH (Khrustaleva and Kik, 2001). Принцип PRINS метода заключается в достраивании Taq полимеразой коротких олигонуклеотидов, гибридизованных с ДНК хромосомы, в присутствии меченных нуклеотидов. Преимущества PRINS по сравнению со стандартной FISH состоит в использовании немеченой пробы, что делает возможным применение больших концентраций пробы без усиления фона. Кроме этого, PRINS занимает меньше времени и поэтому минимизирует разрушение хромосом. Метод был успешно применен для картирования на хромосомах растений. Так, была определена локализация кДНК и гена устойчивости к антракнозу у люпина (Kaczmarek et al. 2007) и локализация кластера генов В-гордеинов у ячменя (Abbo et al. 1993). Tong Zhu c соавторами (Zhu et al. 1995) визуализировал фрагменты ДНК (RFLP маркеры) меньше 1 тыс. п.н. Но большей популярностью этот метод пользуется в детекции повторяющейся ДНК и высококопийных генов 45S и 5S рРНК генов (Kubalakova et al. 1997; Menke et al. 1998). Работа по увеличению чувствительности FISH на хромосоме растений посредством улучшения эффективности мечения ДНК (число встроенных меченых нуклеотидов) была опубликована Kato с соавторами в 2006 году. Авторы работы провели оптимизацию метода ник-трансляции и выявили ключевые компоненты реакции. Главным итогом данной работы было выявление прямой связи между качеством мечения ДНК и концентрацией ДНК полимеразы I в реакции. Авторы установили, что из 3 главных компонентов реакции никтрансляции (ДНК-полимераза I, ДНКаза, меченый нуклеотид) только увеличение концентрации ДНК-полимеразы I приводит к значительному увеличению эффективности мечения ДНК (Kato et al. 2006). Разработанный протокол мечения ДНК в последующем был успешно апробирован для FISH картирования ДНК фрагментов и кДНК размером от 2600 п.н. (Danilova and Birchler, 2008; AcevedoGarcia et al. 2013; Danilova et al. 2012, 2014). 1.2 Создание физических карт растений 21 1.2.1 Подходы к физическому картированию геномов растений На сегодня существует три подхода к разработке физических карт (Jiang and Gill, 2006): 1) полногеномное секвенирование и построение физических карт секвенированных геномов; 2) физическое картирование с помощью коллекций растений с хромосомными нарушениями (анеуплоиды, делеционные линии, линии с замещениями и т.д.); 3) физическое картирование с помощью методов молекулярной цитогенетики (цитогенетические карты). Создание карт секвенированных геномов - это результат бурного развития методов секвенирования (секвенирование следующего поколения) и биоинформационных методов анализа и хранения данных. За 15 лет с момента секвенирования первого генома высшего растения (Arabidopsis thaliana, Arabidopsis Genome Initiative, 2000), список секвенированных и собранных геномов растений значительно увеличился (Bolger et al. 2014) и пополнился важными сельскохозяйственными культурами, такими как рис (Goff et al., 2002), томат (Tomato Genome Consortium, 2012), хлопок (Wang et al. 2012), виноград (Jaillon O. et al. 2007), картофель (Potato Genome Sequencing Consortium, 2011)), сорго (Paterson et al. 2009), кукуруза (Schnable et al. 2009), огурец (Huang et al. 2009). Однако, качественный уровень информации, полученной от последних геномных проектов растений, остаѐтся не таким высоким, как, например, для проекта по секвенированию генома A. thaliana. Исключение составляет только проект по секвенированию генома риса (Feuillet et al. 2010). В секвенировании геномов различных растений было использовано три основных подхода: (1) секвенирование перекрывающихся ВАС (Bacterial Artificial Chromosomes) клонов (рис, A. thaliana (Goff et al. 2002; Arabidopsis Genome Initiative, 2000)); (2) секвенирование методом фрагментации генома (большинство секвенированных геномов растений (Bolger et al. 2014)); (3) секвенирование индивидуальных хромосом (пшеница (Poursarebani et al. 2014), ячмень (Mayer et al. 2011)). Разработка методов секвенирования следующего поколения и их интегрирование с подходом секвенирования методом фрагментации генома значительно ускорили 22 и удешевили процесс секвенирования генома и построения физических карт. Первым растительным геномом, секвенированым с использование только методов секвенирования следующего поколения, стал геном земляники (Shulaev et al. 2011). Однако, большинство карт секвенированных геномов растений создаются с помощью комбинирования различных методов секвенирования (Bolger et al. 2014). Условно весь процесс создания карт геномов растений можно свести к следующему: (1) секвенирование генома (как правило, с помощью методов секвенирования следующего поколения с большим покрытием генома короткими секвенированными фрагментами (ридами, или чтениями)); (2) сборка контигов из коротких ридов; (3) сборка скэффолдов (упорядоченные контиги); (4) создание генетических карт с высокой плотностью маркеров; и (5) упорядочивание контигов и скэффолдов в псевдохромосомы, используя генетические карты. Несмотря на значительный прогресс в разработке эффективных и экономичных методов секвенирования, процесс полного секвенирования генома для большинства растений остается весьма трудоѐмким. Физическое картирование с помощью коллекций растений с хромосомными нарушениями заключается в определении (например, методом ПЦР) присутствия или отсутствия маркера у растений коллекции. Затем результаты такого скрининга сопоставляются с данными о локализации хромосомных перестройках у каждого растения и делается вывод о физическом положении маркера. Используя коллекции растений с хромосомными нарушениями, физическое картирование было проведено для целого ряда сельскохозяйственно-важных культур, таких как кукуруза (Weber et al. 1989; Riera-Lizarazu O. et al. 2000), пшеница (Sears 1954, 1956, 1966; Werner et al. 1992; Gill et al. 1993; Kumar A. et al. 2014), ячмень (Künzel et al. 2000) и лук (Shigyo et al. 1996; Van Heusden et al. 2000; Martin W. J. et al. 2005; Baldwin S. et al. 2014). Цитогенетические карты появились благодаря возможности использования таких молекулярно цитогенетических методов как флуоресцентная (FISH) и геномная (GISH) in situ гибридизация (Islam-Faridi et al. 2002; Khrustaleva L. I. et 23 al. 2005; Jiang and Gill, 2006; Kirov et al. 2014). Большинство цитогенетических карт созданы с использованием метода FISH. FISH картирование включает несколько этапов: 1) Приготовление препаратов; 2) Создание пробы; 3) Проведение гибридизации in situ; 4) детекция сайтов гибридизации и 5) анализ препаратов и детекция сигнала с помощью флуоресцентного микроскопа. Принцип и модификации FISH будут подробно рассмотрены в главе 1.2.2. 1.2.2 FISH – главный инструмент физического картирования хромосом растений FISH - наиболее часто используемый метод для создания физических (цитогенетических) карт, как растений, так и животных. Принцип метода заключается в гибридизации меченого фрагмента ДНК с ДНК хромосомы с последующей визуализацией сайтов гибридизации. Широкое применение данного метода обусловлено тем, что он позволяет напрямую визуализировать интересующие исследователя последовательности ДНК на хромосомах. С помощью FISH были проведено физическое картирование на хромосомах растений различных ДНК последовательностей, включая повторяющиеся элементы генома (рРНК гены, тандемные повторы, мобильные элементы), ВАС клоны и уникальные последовательности (гены) (Figueroa and Bass, 2010; Jiang and Gill, 2006). Однако, чувствительность традиционной FISH не позволяет рутинно визуализировать последовательности меньше 3-10 тыс.п.н. (Valarik et al. 2004). Но на сегодня созданы различные модификации метода, благодаря которым во многом удаѐтся избежать проблем, связанных с этим ограничением, о которых подробно будет изложено в разделе 1.2.3. Наряду с чуствительностью метода FISH важным параметром для успешного цитогенетического катирования генов является разрешающая способность метода, т.е. наименьшее расстояние между двумя соседними последовательностями ДНК, при котором флуоресцентные сигналы от этих двух последовательностей не перекрываются. FISH обладает низкой разрешающей 24 способностью метафазных на компактных митотических метафазных хромосом хромосомах. картофеля было Например для показано, что чувствительность FISH составляет - 12 млн.п.н., а для гетерохроматина и эухроматина менее компактных пахитенных хромосом - 1,2 млн.п.н. и 120 тыс.п.н. соответственно (Lavania et al. 2004). Разрешающая способность зависит от уровня упаковки ДНК митотические в цитологическом материале: интерфазные ядра, прометафазные и метафазные хромосомы, растянутые митотические метафазные хромосомы, мейотические пахитенные хромосомы и растянутые ДНК нити. Для картирования, наиболее часто используются митотические метафазные хромосомы из-за доступности меристематических клеток практически для всех видов растений (Jiang and Gill, 2006). Для преодоления ограничений чувствительности метода FISH часто используют YAC (Yeast Artificial Chromosomes) клоны, BAC (Bacterial Artificial Chromosomes) клоны и космиды. Эти клоны содержат протяженные фрагменты ДНК: 400-600 (YAC), 100-150 (BAC) и 40 т.п.н. (космиды). BAC векторы используются намного чаще для физического картирования хромосом, чем космиды и YAC. Это объясняется большим размером возможной вставки чужеродной ДНК для клонирования в BAC, чем в космиды и меньшей способностью к химеризму, характерному для YAC векторов (Mohan et al. 1997). Большие фрагменты ДНК BAC клонов, несущие интересующий ген легко детектируются на хромосомах с помощью обычной FISH. Используя BAC клон и космиду, Nobuko Ohmido с соавторами (1998) удалось с помощью FISH физически картировать гены устойчивости к пирикуляриозу и пятнистости листьев риса (Ohmido et al. 1998). Данные гены устойчивости были локализованы на хромосомах разновидностей риса, отличающихся от разновидностей, из которых получены пробы (BAC клон и космида) для картирования. С помощью BAC-FISH на пахитенных хромосомах томата удалось установить точную физическую локализацию гена устойчивости к нематоде (Mi) и гена фосфатазы 1 (Aps-1) и рассчитать расстояние между этими генами в парах нуклеотидов равное 25 40 млн. п.н. Интересно отметить, что, генетическое расстояние между этими генами составило всего лишь 1.1 сМ, в то время как физически гены были удалены на десятки млн. п.н., и более того эти гены были расположены в разных плечах хромосомы, т.е. гены разделяла область центромеры. Работы по цитогенетическому картированию генов устойчивости с помощью BAC-FISH были проведены на рисе (Jiang et al. 1999), картофеле (Dong et al. 2000), яблоне (Cevik and King, 2002), перце (Kang et al. 2001). Кроме генов устойчивости, с помощью BAC-FISH были физически картированы многие другие гены растений, например, ген восстановления фертильности канолы (Feng et al., 2009), ген Bronze1 (Bz1) кукурузы (Koumbaris and Bass, 2003), локус Sun томата (Van der Knaapet al. 2004). BAC-FISH нашла применение и в уточнении генетических карт. Так, два маркера генома сорго, Xtxs1176.1 и Xtxa2344, были ранее картированны в группе сцепления А (LG-A), однако, их положение относительно друг друга было неясно (Islam-Faridi et al. 2002). BAC-FISH позволила не только установить их физическое положение, но и определить принадлежность LG-A хромосоме 1. Кроме этого, используя BAC клоны, несущие ранее генетически картированные маркеры LG-A, и FISH на пахитенных хромосомах, авторы создали цитогенетическую карту более 20 маркеров и установили положение центромеры на генетической карте (Islam-Faridi et al. 2002). Однако, BAC-FISH имеет несколько существенных недостатков. Вопервых, это трудоѐмкость создания и скрининга BAC библиотеки. Во-вторых, BAC клоны растений содержат повторяющиеся последовательности, которые маскируют сигнал, исходящий от интересующего нас гена, из-за кроссгибридизации меченной ДНК пробы с многими участками хромосом (Islam-Faridi et al. 2002; Khrustaleva and Kik, 2001; Suzuki et al. 2001). Предотвратить кроссгибридизацию помогает использование С0t-1 или С0t-100 фракций геномной ДНК содержащие высоко- и умеренноповторяющиеся последовательности ДНК в качестве блока (немеченая ДНК), концентрация которой в десятки раз больше 26 меченой ДНК BAС клона (проба) (Wang et al. 2006; Ohmido et al. 1998). В качестве блока так же используют фрагментированную ДНК из спермы лосося (Ohmido et al. 1998). 1.2.3 Физическое картирование хромосом растений с помощью TyramideFISH Первая работа с применением Tyramide-FISH на растениях была опубликована в 2001 году (Khrustaleva and Kik, 2001). В данной работе авторы продемонстрировали, что с помощью Tyramide-FISH возможно детектировать ДНК фрагменты длиной менее 1000 п.н.. Так же было показано, что при детекции последовательности Т-ДНК размером 710 п.н. на хромосомах Allium cepa, сигнал был детектирован без использования CCD камеры на 36% метафаз, в то время как при применении стандартной FISH даже вместе с ССD камерой не было детектировано ни одного сигнала. По сравнению cо стандартной FISH, TyramideFISH имеет ряд явных преимуществ для детекции коротких последовательностей: 1) высокая чувствительность; 2) более высокая частота встречаемости сигнала и 3) короткое время детекции (3-5 мин.) (Khrustaleva and Kik, 2001). Цитогенетическое картирование хромосом с использованием Tyramide-FISH было проведено на нескольких видах растений, в том числе на луке (Khrustaleva and Kik, 2001; Khrustaleva et al. 2012; Романов, 2012; Романов и др., 2014), ячмене (Stephens et al., 2004), пшенице (Perez et al. 2009), овсе (Sanz et al., 2011) и розе (Kirov et al. 2014a, 2015a). В работе на пшенице (Perez et al. 2009), Perez с соавторами локализовали ген Rad50 на коротком плече хромосом 5А, 5B и 5D. Используя тирамиды, конъюгированные с биотином, с последующей детекцией последнего с помощью стрептавидина, конъюгированного с флуорохромом, авторы визуализировали фрагмент последовательности гена размером 2000 п.н.. Что бы оценить чувствительность метода на хромосомах пшеницы, были созданы и детектированы фрагменты Rad50 различной длины (12, 8, 4 и 2 тыс. п.н.). 27 Частота встречаемости клеток с сигналом уменьшалась, в зависимости от размера пробы, с 57,5% до 37,5%. При детекции пробы 2 тыс.п.н. чувствительность метода увеличивалась в 30 раз, если применяли CCD камеру (Perez et al. 2009). Возможность последовательностей использования ДНК на Tyramide-FISH хромосомах для детекции растений коротких была ярко продемонстрирована в работе Stephens с соавторами (Stephens et al., 2004) при цитогенетическом картировании RFLP клонов на хромосомах ячменя. Из 24 (0.5 3 тыс.п.н.) клонов была визуализирована локализация 14. Уникальность этой работы состоит в том, что авторы картировали не просто маркеры, а индивидуальные гены в виде кДНК. Так, было показано, что три используемых пробы (кДНК убиквитина, алкогольдегидрогеназы и гистона) имели несколько сайтов локализации, а оставшиеся 11 кДНК проб гибридизовались с хромосомами в единичных сайтах. Авторами этой работы удалось не только ещѐ раз показать ценность метода Tyramide-FISH, как инструмента для картирования отдельных генов растений, но и для интегрирования генетической и физической карт. Использование Tyramide-FISH позволило установить хромосомную локализацию предполагаемых генов устойчивости (RGA) на хромосомах овса (Avena sativa) и родственных видов (A.strigosa, A.eriantha, A.byzantina) (Sanz et al., 2011). Результаты позволили подтвердить родство на уровне хромосом между диплоидными (A.strigosa, A.eriantha) и гексаплоидными видами (A.sativa, A.byzantina), интегрировать генетические и физические карты, а также выявить эволюционные хромосомные различия между видами (Sanz et al., 2011). Соотнесение групп сцепления с физическими хромосомами – это первый этап в интегрировании карт. При этом, использование Tyramide-FISH значительно облегчает этот процесс, т.к. приготовление пробы не требует создания специальных библиотек (YAC, ВАС, EST клонов) и сводится к ПЦР амплификации целевого фрагмента и клонированию ПЦР продукта. TyramideFISH был использован с данной целью на хромосомах овса (Sanz et al., 2011), розы (Kirov et al. 2014) и лука (Romanov et al. 2015). 28 Другим направлением использования Tyramide-FISH является картирование и идентификация генов локализованных на половых хромосомах. Подобные работы были проведены на хромосомах Silena latifolia (Nakao et al. 2002). Авторы использовали Tyramide-FISH для детекции как коротких (<3000 п.н.), так и длинных фрагментов ДНК. Главными причинами сравнительно небольшого числа публикаций, посвященных картированию генов на хромосомах растений с помощью TyramideFISH, являются слабая воспроизводимость данного метода на разных видах растений. Это обусловлено трудоѐмкостью приготовления препаратов хромосом растений, удовлетворяющих требованиям метода, и слабая изученность факторов, влияющих на процесс Tyramide-FISH детекции. 1.3 Основные факторы успешного физического картирования генов с помощью FISH Успех физического картирования коротких фрагментов ДНК (генов, маркеров) во многом определяется несколькими факторами: 1) качеством приготовленных препаратов хромосом; 2) выбранной системой детекции сайтов гибридизации (С-PRINS, Tyramide-FISH, прямой метод детекции); 3) наличием цитогенетических маркеров для идентификации индивидуальных хромосом (особенно критично для видов с мелкими хромосомами и при картировании пахитенных хромосом); 4) дизайном и созданием локус-специфичной пробы. 1.3.1 Препараты хромосом Ключевым шагом, определяющим 70-80% успеха, в любых цитологических и молекулярно-цитогенетических качественных цитологических молекулярно-цитогенетических исследованиях препаратов, является подходящих исследованиях растений для приготовление FISH. В материалом для препаратов чаще всего выступают митотические метафазные или пахитенные хромосомы. Приготовление препаратов митотических метафазных хромосом 29 растений осложняется главным образом наличием у растений жѐсткой клеточной стенки, препятствующей гидролизу цитоплазмы и получению подходящего разброса хромосом. Главные требования, предъявляемые к препаратам хромосом, являются: отсутствие цитоплазмы и клеточных остатков вокруг и на хромосомах, сохранение структуры хромосом, хороший разброс и отсутствие перекрытий хромосом (Ambros et al. 1986; Schwarzacher et al. 1989). Как правило, результаты молекулярно-цитогенетических исследований, проведѐнных на препаратах, не удовлетворяющих этим требованиям, не высоки и невоспроизводимы. Основными этапами, предшествующими приготовлению препаратов, являются: 1) Предобработка цитостатиками (колхицин, 1-бромнафталин, N20, ледяная вода и др.); 2) Фиксация (как правило, в 3:1, 100% этанол:100% уксусная кислота); 3) Промывка и Мацерация. Мацерация ткани позволяет гидролизовать межклеточное вещество и клеточную стенку и проводится двумя способами: с помощью кислот (уксусная (Clark and Karp, 1989; Karp, 1994; Mirzaghaderi, 2010), соляная (Marks, 1973)) и ферментов (Schwarzacher and Leitch, 1994) или последовательно с помощью кислоты и ферментов (Ma et al.1996). Ферментативный гидролиз - наиболее предпочтителен для высокочувствительных молекулярно-цитогенетических исследований, где даже небольшие клеточные остатки могу привести к высокому уровню фонового сигнала. Каждый из существующих методов приготовления митотических метафазных хромосом растений имеет ряд недостатков, требует большого опыта и навыков владения, и подходит для ограниченного числа видов растений. На животных клетках разработаны методы, позволяющие быстро и эффективно приготовить качественные препараты хромосом. Методы приготовления препаратов митотических метафазных хромосом животных и человека рутинно 30 применяются в научных исследованиях и диагностике генетически наследуемых заболеваний и приобретенных патологий, например, онкологических заболеваний, обусловленных крупными хромосомными перестройками (Stephens et al. 2011). Это стало возможным, благодаря не только тому, что у животных клеток отсутствует клеточная многочисленным работам, стенка, но, главным образом, благодаря посвященным изучению физико-химических процессов, протекающих во время приготовления препаратов, и факторов, влияющих на качество разброса хромосом (Spurbeck J. L. et al. 1996; Hliscs et al. 1997; Henegariu et al. 1999; Claussen U. et al., 2002; Deng et al. 2003; Ami et al. 2014). Данные процессы и факторы не были достаточно исследованы на растениях. Сегодня существуют три основных метода приготовления митотических метафазных хромосом растений: метод давления (с англ. squash), метод распластывания (с англ. spread) и метод капли (drop method). Приготовления пахитенных хромосом проводится только методом давления и распластывания. Из всех вышеперечисленных методов приготовления препаратов растительных хромосомсравнительно новым является метод капли, который, благодаря ряду работ по его модификации, набирает все большую популярность. Работы по созданию метода капли для приготовления препаратов митотических метафазных хромосом растений были начаты Mouras с соавторами (Mouras et al., 1978) и Murata (Murata, 1983) более 30 лет назад. Mouras с соавторами (1978) использовали живые корни для обработки ферментами и получения клеточной суспензии, которую затем фиксировали и раскапывали на стѐкла. Murata (1983) адаптировал к растительной клетке метод приготовления препаратов, широко используемый для клеток животных и человека. Ранее, для преодоления проблем, связанных с жѐсткой клеточной стенкой и тканью, в целом, у растений использовали суспензию протопластов: Murata (1983) на шпинате, и Rayburn и Gold (1983) на капусте и кукурузе (Rayburn и Gold 1983). После гипотонии (0.075М KCl) и серии промывок в фиксаторе (3:1), суспензию протопластов 31 раскапывали на влажное предметное стекло и высушивали. Эти основные этапы метода капли используются с незначительными модификациями и сегодня. Kato (2004) разработал метод капли, который имеет ряд важных преимуществ: 1) ядра и хромосомы образуют монослой и менее повреждены, чем при других методах; 2) одинаковое качество препаратов; 3) относительная простота и быстрота приготовления препаратов. Среди недостатков метода выделяются: a) для приготовления препаратов необходимо большое количество материала с высоким числом метафаз, и, как следствие б) невозможность приготовления препаратов из одного корня (Schwarzacher and Leitch, 1994); в) метод более подходит для мелких хромосом, так как крупные хромосомы плохо поддаются разбросу (Schwarzacher and Leitch, 1994). Данный метод был применѐн для хромосом кукурузы (Kato, 1997, 1999; Kato et al. 2004), ячменя, пшеницы (Busch et al., 1994; Komuro S. et al. 2013), риса (Abbasi F. M. et al. 2012), роз (Ma et al. 1996), берѐзы (Anamthawat-Jónsson, 2004) и других видов (Geber and Schweizer, 1988). Модификации метода капли, проведѐнные рядом авторов, позволили преодолеть некоторые ограничения (Kato, 1997, 1999; Andras S. C. et al. 1999). Kato (1997, 1999) оптимизировал метод капли для кукурузы и ряда других культур, и для увеличения разброса предложил увеличить концентрацию уксусной кислоты и использовать раствор 9:1 (уксусная кислота:спирт) или 100% уксусною кислоту вместо раствора 3:1. Эта модификация совместно с применением веселящего газа (N2O) в качестве цитостатика позволили эффективно использовать данный метод на кукурузе и пшенице (Kato, 1997; 1999; Kato et al. 2004; Komuro S. et al. 2013). Однако, применение N2O требует специального оборудования и, возможно, из-за его наркотического действия, будет скоро ограничено в России. Стоит отметить, что применение N2O на пшенице не позволяет получить большого числа хорошо разбросанных метафаз (Komuro et al. 2013). Для увеличения общего числа метафаз на стекло, Busch с соавторами (1994) провели синхронизацию клеток с помощью гидроксимочевины с последующей обработкой высокоэффективным цитостатиком – 32 амипрофосметил (АПМ). Так же авторы модифицировали метод капли для использования его в приготовлении хромосом для высокочувствительной сканирующей микроскопии. Однако, высокое число метафаз, получаемое после синхронизации, не гарантирует хорошего разброса. Таким образом, спустя более 30 лет после первой публикации с использованием метода капли на растительной клетке, его основные этапы, как и недостатки, остались практически без изменения. Главными причинами этого остаѐтся неизученность физико-химических процессов, протекающих при приготовлении препаратов митотических метафазных хромосом растений. Физико-химические процессы, протекающие при разбросе хромосом, были хорошо изучены на клетках человека и животных (Spurbeck J. L. et al. 1996; Hliscs et al. 1997; Henegariu et al. 1999; Claussen U. et al., 2002; Deng et al. 2003; Ami et al. 2014). Результаты этих исследований показали динамичность и сложность процессов, протекающих при разбросе хромосом. Было показано, что качество препарата, приготовленного методом капли, зависти от многих факторов: температуры окружающей среды, влажности, высоты с которой проводится капание, ширины капли ,количества фиксатора, температуры и наклона предметного стекла. Причины разброса хромосом и влияние каждого из этих факторов на качество разброса разными учѐными рассматривается по-разному и одна гипотеза сменяет другую. Изначально предполагалось, что сам процесс разброса хромосом происходит из-за резкого «взрыва» клеток в суспензии вследствие еѐ падения (в виде капли) на стекло. Однако, более поздние работы (Henegariu et al. 1999; Deng et al.,2003) опровергли это предположение. Всю неоднозначность современных представлений о значении перечисленных выше факторов и трудоѐмкость приготовления препаратов хромосом Bursch с соавторами (1994) выразили следующими словами о приготовлении препаратов: «it is more of an ―art‖ than science» («это больше искусство, чем наука»). 33 Claussen с соавторами (2002) наблюдали за клетками, нанесѐнными на стекло, после добавления гипотонического раствора, фиксатора (3:1, уксусная кислота: спирт), уксусной кислоты и воды. Добавление воды после фиксатора и уксусной кислоты приводило к резкому набуханию митотических клеток (Рисунок 3). Рисунок 3. Морфологические изменения митотических клеток амниотической жидкости после добавления гипотонического раствора (a), фиксатора (b), уксусной кислоты (с) и воды (d-e) (Claussen et al. 2002). Позже Deng с соавторами (2003), наблюдая за поведением хромосом, выделил этапы, предшествующие разбросу хромосом. Эти этапы отражают динамичность процессов, проходящих при разбросе хромосом: 1) Первые 10 секунд: клетки распределяются по поверхности стекла и иммобилизируются; 2) От 10 до 25 секунд: не происходит видимых изменений в движении клеток, а к концу этого периода хромосомы начинают быть видимыми в фазовый микроскоп; 34 3) 25-50 секунд: фиксатор продолжает испаряться, клетки увеличиваются в размере, и происходит разброс. В течение этой фазы разброс происходит быстро (в отличии от того, что предполагали ранее (Hliscs et al. 1997)) и преимущественно за первые 5 секунд. 4) 50-90 секунд: фиксатор испаряется, и хромосомы прикрепляются к стеклу. Хромосомы в области с замедленным испарением фиксатора часто имеют переразброс и вымываются. Проведѐнные Deng с соавторами (2003) детальные микроскопические исследования поведения хромосом человека во время приготовления препаратов, позволили сформулировать следующие выводы: 1) Высота капания, изначальная влажность и наклон стекла, соотношение спирта и кислоты в фиксаторе и время сушки от 40 до 140 секунд не существенно влияют на разброс; 2) Главный фактор, влияющий на разброс, - это влажность воздуха. Таким образом, мнение многих авторов сходится в том, что вода играет главную роль в разбросе хромосом, поступая в клетку и вызывая еѐ набухание и разброс хромосом. Предполагается, что это происходит следующим образом: фиксатор распределяется по стеклу и, в силу своей высокой гигроскопичности, абсорбирует влагу из воздуха, при этом высвобождается свободная энергия, которая и является движущей силой хромосом, приводящей к их разбросу (Deng et al. 2003). Согласно Claussen с соавторами (2002), абсорбция воды происходит не только фиксатором, но и ацетатом аммония, образовавшимся в результате ацетилирования белков клетки уксусной кислотой. Другим фактором, который достоверно влияет на разброс, является температура (Spurberck et al. 1996). На основе анализа большого числа клеток человека и животных было показано, что разброс хромосом – это функция от температуры и относительной влажности (Spurberck et al. 1996). Нами впервые был проведен анализ динамики разброса хромосом растений. Результаты этих исследований показывают, что динамика разброса хромосом 35 клеток растений, человека и животных после нанесения суспензии клеток в фиксаторе на стекло имеет много общего. Проведенные нами исследования позволили усовершенствовать метод капли и создать новый универсальный метод приготовления хромосом растений, названный ―SteamDrop‖ (Kirov et al. 2014b). 1.3.2 Системы детекции сайтов гибридизации Наиболее часто используемые системы детекции сайтов гибридизации можно объединить в две группы: прямая детекция, когда пробу непосредственно метят флуорохромами, и непрямая детекция, нефлуоресцирующими соединениями - гаптенами диоксигенин. При непрямой системе детекции когда пробу метят такими как биотин или визуализацию сайтов гибридизации осуществляют с помощью антител и/или ферментов (TyramideFISH), конъюгированных с флуорохромами. Различные системы детекции сайтов in situ гибридизации рассмотрены также в других главах диссертации (1.2.3, 1.4). 1.3.3. Цитогенетические маркеры хромосом растений Физическое картирование с помощью молекулярно-цитогенетических методов осложняется трудностью идентификации хромосом, несущих сигналы. Попытки распознать индивидуальные хромосомы начались с момента открытия хромосомы в 1882 году немецким анатомом В. Флемингом. С тех пор учѐные занимаются описанием кариотипов живых организмов (кариотипирование). Кариотипирование подразумевает описание числа хромосом, их размеров, положение центромеры (метацентрическое, субметацентрическое, акроцентрическое, телоцентрическое) и вторичной перетяжки. Однако, кариотипирование занимает много времени и сильно затруднено на видах, хромосомы которых схожи по размеру и центромерному индексу, а так же на видах с большим числом хромосом и сложной конституцией генома (аллополиплоиды). Для преодоления этих трудностей сегодня разрабатываются системы цитогенетических индивидуальные хромосомы маркеров, в позволяющие кариотипе даже без легко различать кариотипирования. 36 Использование цитогенетических маркеров началось с разработки методов дифференциального окрашивания хромосом, позволяющих идентифицировать индивидуальные хромосомы и их отдельные регионы по специфической картине окрашивания хромосом. Примерами такого окрашивания может служить С – бэндинг с использованием красителя Гимза; люминисцентное окрашивание: хромомицином А3 (СмÀ3), 4,6-диамидино-2-фенилиндолом (DAPI), окрашивание серебром (Badaeva E. D. et al. 1986; Gill et al. 1991; Olin-Fatih and Heneen, 1992; Cheng et al. 1995; Snowdon et al. 2000; Hasterok R. and Maluszynska, 2000; Koo et al. 2005; Muravenko O. V. et al. 2009; Bolsheva et al. 2014; She et al 2015; Bolsheva et al. 2015). С введением бендинга хромосом в семидесятых годах, была облегчена идентификация индивидуальных хромосом. Но на некоторых растительных объектах бэндирование не дает специфической картины для распознавания отдельных хромосом (напр. виды рода Allium). Новым витком в разработке цитогенетических маркеров было создание метода FISH. FISH позволяет эффективно проводить идентификации индивидуальных хромосом и геномов (GISH). Благодаря FISH появилось такое определение как «chromosome painting», или окрашивание индивидуальных хромосом, впервые описанное Pinkel с соавторами (Pinkel et al., 1988). Растительные геномы обогащены повторяющимися последовательностями, некоторые из которых локализуются в определѐнных частях хромосомы (центромера, теломера, гетерохроматин) и организованны тандемно, занимая миллионы пар нуклеотидов в геноме. Локализация повторяющихся последовательностей может различаться между отдельными парами хромосом в кариотипе, что делает их перспективными кандидатами для разработки FISH цитогенетических маркеров (Olin-Fatih and Heneen, 1992; Cheng et al. 1995; Tsujimoto et al. 1997; Snowdon et al. 2000; Hasterok R. and Maluszynska, 2000; Navratilova et al. 2003; Koo et al. 2005; Muravenko O. V. et al. 2009; Bolsheva et al. 2014; She et al 2015; Schmidt and Heslop-Harrison, 1998). 37 Пробы для FISH маркирования хромосом могут быть получены из консервативных повторяющихся последовательностей генов, таких как 45S (18S5.8S-25S) рРНК, 5S рРНК генов и теломерных повторов. 45S и 5S рРНК гены были успешно использованы для дифференциации хромосом растений (Kato A. et al. 2005). В качестве пробы для FISH маркирования могут выступать и тандемные повторы, чья функция в геноме остается неизвестной. По сравнению с рРНК генами, такие повторы часто встречаются на большем числе геномных локусов, и поэтому могут давать сигналы, характерные для каждой отдельной пары хромосом в кариотипе, как это было показано, например, для Vicia faba (Macas et al., 2003), V. sativa (Navratilova et al. 2003) и видов Triticum (Friebe et al. 1991; Komuro et al. 2013). Кроме разного рода повторяющихся ДНК, источником цитогенетических маркеров могут выступать уникальные последовательности генома. Но это возможно лишь, если размер уникальных последовательностей ДНК выше порога чувствительности FISH, т.е. более 10 000 п.н. Такие последовательности ДНК получают из ДНК библиотек (BAC, YAC, космиды). ВАС клоны, несущие большие вставки геномной ДНК (более 100 000 п.н.) показали свою высокую эффективность, как хромосом-специфичные цитогенетические ДНК маркеры, особенно на видах с небольшим размером генома (Dong et al.. 2000). Так с помощью BAC-FISH были маркированы половые хромосомы и некоторые аутосомы Silene latifolia (Lengerova M. et al., 2004). Библиотеку BAC клонов использовали при создании цитогенетических маркеров для Silene и Lotus japonicus (Pedrosa et al. 2002), Pinus (Hizume M. et al. 2002) и Picea abies (Vischi M. et al. 2003). Однако на видах с крупным геномом использование ВАС клонов в качестве хромосом-специфичных цитогенетических маркеров затруднительно изза наличия большого числа семейств повторяющихся ДНК, которые гибридизуется с ДНК всех хромосом и тем самым затрудняет идентификацию хромосом (Zhang P. et al. 2004). 38 Ввиду значимости цитогенетического маркирования хромосом растений, методы поиска ДНК повторов, которые могут быть использованы для FISH маркирования хромосом, многообразны. Например, некоторые ДНК маркеры (SSR, ISSR, RAPD), основанные на повторах ДНК, могут быть эффективно использованы для выделения FISH цитогенетических маркеров из генома (Cuadrado and Jouve, 2010). Так, на Cucumis sativus использование RAPD позволило выделить повторяющиеся ДНК, которые стали эффективными цитогенетическими маркерами, способными дифференцировать все 7 пар хромосом (Koo et al. 2005). Самым современным и наиболее эффективным методом поиска повторов в геномах растений является биоинформационный метод, разработанный в лаборатории Jiri Macas (Чехия). Принцип этого метод заключается в использовании ридов после секвенирования следующего поколения и их попарное сравнение (каждый с каждым). Затем, риды отображаются в 3D пространстве как «точки», соединенные линиями, а расстояние между ними тем меньше, чем больше их сходство. Схожие риды образуют скопления, или кластеры ридов. Форма кластера определяется геномной организацией ДНК повтора. Например, тандемные повторы образуют глобулы (плотные кластеры) и кольца, а кластеры, соответствующие ретротранспозонам, имеют форму линии (Novak et al. 2010, 2013). Кроме всех достоинств данного метода (высокая производительность, информативность, экономичность), его использование облегчается существованием Интернет сайта (http://www.repeatexplorer.org/), где можно проанализировать любой набор ридов. Всего за несколько лет данный метод был успешно применен для выделения и характеристики повторяющейся ДНК на многих видах растений (http://www.repeatexplorer.org/). 1.4 Принцип Tyramide-FISH 39 1.4.1 Реакция тирамид с пероксидазой хрена Пероксидаза хрена (ПХ) имеет целый ряд субстратов, с которыми она может вступать в реакцию с образованием продукта реакции. Наибольшее освещение в литературе получили реакции полимеризации фенольных соединений. Это связано, прежде всего, с высокой ценностью данного типа реакция для химического и пищевого производства. Способность пероксидазы обуславливать полимеризацию ярко продемострирована целым рядом статей (обозрено у Gross et al. 2001, Рисунок 7). Рисунок 7. Полимеризация фенола и его производных, вызванное пероксидазой хрена (Gross et al. 2001). В реакцию полимеризации, катализируемую пероксидазой, могут вступать не только тирозин и тирамин, но другие молекулы (феруловая кислота, 3-(4гидроксифенил пропионовая кислота и др.). Эти и другие работы показывают (Akkara et al. 1991; Kobayashi and Higashimura, 2003; Tonami et al. 1999; Zhang et al. 2012; Turac and Sahmetlioglu, 2010; Zhou et al. 2013), что ПХ – эффективный биокатализатор для полимеризации фенольных соединений. В присутствие перикиси водорода ПХ может окислять тирамины и тирамиды (тирамин, коньюгированный с гаптеном) в хинон-подобные структуры, несущие радикал на втором атоме углерода в их фенольном кольце. Как и многие другие производные фенола тирамин в реакции с пероксидазой может образовывать ди-, три- и полимеры (Yamaoka et al. 1995). Всего за минуту ПХ может «активизировать» около 107 молекул субстрата (Zhang et al., 2013). Из-за короткого времени существования тирамид в активизированной форме, они 40 вступают в реакцию с другими тирамидами в непосредственной близости от пероксидазы. Эти знания позволили использовать ПХ в гистохимии и молекулярной цитогенетике (Tyramide-FISH, CARD, TSA-FISH). Однако, ни сам тирамин, ни его полимер не имеют специфической окраски, поэтому были использованы модифицированные тирамиды, представляющие собой коньюгаты с флуорохромами (FITC, Cy3 и др.) или гаптеном (биотин, дигоксигенин и др.). Коньюгирование тирамидов не мешает прохождению реакции полимеризации. Что бы применить данную реакцию для гистохимических исследований ПХ была химически коньюгирована с антителом или стрептавидином. Это позволило доставить ПХ к целевой молекуле ДНК, РНК или белка в клетке, зафиксированной на стекле. Последующее инкубирование препарата с раствором тирамид-коньюгатов приводит к образованию димеров с фенольными соединениями в непосредственной близости от сайта локализации ПХ. Эти соединения являются: 1) фенольными соединениями, присутствующими в ткани; 2) ранее иммобилизированными тирамидами (тримеризация и т.д.); 3) тирамидами, присутствующими в растворе в качестве субстрата. Теоретически, в качестве фенольных соединений, присутствующих в ткани и способных вступать в реакцию с активированными тирамидами, могут выступать остатки аминокислоты тирозина (Van Gijlswijk et al. 1996) (Рисунок 8). 41 Рисунок 8. Реакция между тирамидами, коньюнированными с тетраметил родамином (Tyramide-TMR), и пероксидазой хрена (Watabe S. et al. 2011). Результатом реакции является присоединение меченных тирамидов к тирозину белка. 1.4.2. Способы модификации реакции для улучшения детекции антигена на мембране и в пробирке Широкое применение реакции ПХ с тирамидами в иммуно-ферментном анализе побуждало к созданию новых, более чувствительных модификаций этого метода. Одной из таких работ, проведенной на мембране, является работа Bhattacharya с соавторами (1999). Авторы выдвинули предположение, что увеличение числа фенольных соединений вокруг ПХ, с которыми могут вступать в реакцию активизированные тирамиды, позволит увеличить чувствительность метода. Чтобы проверить эту гипотезу авторы пришили («обогатили») к белку (бычий сывороточный альбумин.), используемому для блокировки неспецифических сайтов на мембране, остатки 3-(4-гидроксифенил) пропионовой кислоты (p-OH-ppa) и создали «электрон-богатый» белок (Рисунок 8). Для детекции иммуноглобулинов кролика, авторы использовали DOT-ELISA коньюгированными с биотином. иммобилизированных на мембране, метод с детекцией тирамидами, 42 Рисунок 8. Схема обогащения белка остатками 3-(4-гидроксифенил) пропионовой кислоты (p-OH-ppa) (Bhattacharya et al.,1999). Получившийся белок был использован вместо белка для блокировки. Результаты этого эксперимента показали, что применение электрон-богатого белка позволило увеличить чувствительность метода в 10 000 раз. Последующие работы так же успешно применили данный подход для конкретных исследований (Pal and Dhar, 2004; Zhang et al., 2011; Huang X. et al. 2013).Таким образом, на мембране было подтверждена гипотеза о том, что окисленные ПХ тирамиды вступают в реакцию с окружающими ПХ фенольными остатками. Подобные работы не проводились для улучшения чувствительности метода на биологических препаратах. Это связано, прежде всего, с трудностью доставки и закрепления такого белка в месте локализации ПХ, т.к. в отличие от мембраны, после нанесения «электрон-богатого» белка на препарат, он будет смыт последующими промывками. 1.4.3 Принцип Tyramide-FISH Tyramide-FISH объединяет преимущества ферментной (ПХ) депонирования многих молекул тирамид с более высокой чувствительностью флуоресцентной детекции. Принцип метода заключается в детекции пробы, гибридизованной с ДНК хромосомы, с помощью антител, коньюгированных с пероксидазой хрена (ПХ). В присутствии перекиси водорода ПХ способна активизировать фенольные 43 соединения, тирамиды, меченные флуорохромом, и вызывать их депонирование в сайте локализации пероксидазы (Рисунок 9). Рисунок 9. Принцип Tyramide-FISH детекции Чувствительность Tyramide-FISH в 100 раз превышает стандартную FISH. Впервые, метод был применѐн в иммунологическом анализе, а позже в 1995 году был оптимизирован для FISH, проводимой на животной клетке (Raap et al. 1995). Метод нашѐл применение в детекции отдельных генов на хромосомах человека (Schmidt et al. 1997) и в 2001 году впервые был адаптирован для хромосом растений (Khrustaleva & Kik 2001). 1.4.4 Системы детекции Tyramide-FISH Разнообразие антител, флуорохромов и гаптенов позволяют разрабатывать всѐ новые системы детекции. Система детекции Tyramide-FISH так же имеет ряд разновидностей, которые можно объединить в две большие группы: прямой метод детекции и непрямой метод детекции. К первому относятся системы, использующие тирамиды, коньюгированные с флуорохромами. Это позволяет проводить визуализацию без последующей детекции антителами, 44 коньюгированные с флуорохромами . Прямая система детекции включает в себя и способ детекции с использованием пробы непосредственно меченой пероксидазой вместо гаптенов (Van Gijlswijk et al. 1996). В непрямых системах детекции используются тирамиды, коньюгированные с гаптенами - химическими соединениями, которые не испускают флуоресценцию (биотин, дигоксигенин). Поэтому при непрямой системе детекции проводится дополнительная детекция антителами, коньюгированными с флуорохромами. Число последующих этапов детекции варьирует и зависит как от используемого типа препарата (тканевые срезы, хромосомы), так и от размера и типа (ДНК, мРНК) детектируемой последовательности. Для физического картирования уникальных ДНК последовательностей на хромосомах растений использовалась как прямая, так и непрямая системы детекции (Таблица 1). Концентрации тирамид и антител (в случае непрямой детекции) не изменялись и использовались в соответствии с протоколами производителя. 45 Таблица 1. Системы детекции Tyramide-FISH, использованные на хромосомах растений для детекции коротких фрагментов ДНК Вид Способ детекции Allium cepa Прямой Используемые Размер ЧВС1 Тирамиды пробы, п.н. Тирамид-FITC 710 36.5% (1:50) Ссылка Khrustaleva and Kik, 2001; Khrustaleva et al., 2012 Triticum Непрямой ТирамидОт 37.5% Perez et al. aestivum Биотин (1:50) 2000 2009 Hordeum Непрямой ТирамидОт 500 Не Stephens et al. vulgare Биотни (1:50) (кДНК опреде 2004 ) ляли Avena sativa Прямой Тирамид-Cy3 От >37.5 Sanz et al., (1:50) 2000 % 2011 Silene latifolia Непрямой ТирамидОт Не Nakao S. et al. биотин, 2236 опреде 2002 Стрептавидинляли HRP, Тирамидбиотин Rosa Непрямой ТирамидОт 30-40 Kirov et al. wichurana Биотин (1:25) 1100 2014 1 Частота встречаемости сигнала При прямой системе детекции для Tyramide-FISH не требуется проводить дополнительную детекцию тирамид-коньюгатов антителами, так как тирамиды коньюгированы непосредственно с флуорохромами (Рисунок 10). 46 Рисунок 10. Принцип прямой системы детекции Tyramide-FISH. Большую работу по оптимизации прямой системы детекции провели van Gijlswijk с соавторами (1996) на срезах тканей крысы и хромосомах человека. Авторы проверили тирамиды, меченные флуорохромами с волной испускания в голубом зелѐном и красном диапозоне спектра, на возможность их использования в иммуноцитохимических, иммуногистохимических исследованиях. Из 12 флуорохромов два, а именно давали плохие результаты в FISH экспериментах. была использована в Tyramide-FISH и X-Rhodamin, Texas Red, Прямая для цитогенетических система детекции детекции уникальных последовательностей на хромосомах растений (Sanz et al. 2012; Khrustaleva and Kik, 2001). В отличие от прямой системы детекции в непрямой системе после реакции тирамид с пероксидазой, тирамиды должны быть детектированы с помощью антител к гаптену, коньюгированных с флуорохромом (Рисунок 11). В роли гаптена выступают нефлюоресцирующие соединения (дигоксигенин, биотин и др.). Широкое разнообразие гаптенов, которые могут быть коньюгированы с тирамидами, детекции. определяет доступность современных коммерческих систем 47 Рисунок 11. Принцип непрямой системы детекции. Если для хромосом и гистологических срезов млекопитающих оптимизированы разные варианты непрямой системы детекции, то для хромосом растений оптимизаций систем детекции проведено не было. Среди работ, где использовалась непрямая система детекции, можно выделить два типа таких систем: 1) Тирамид-биотин, Стрептавидин-TexRed (Stephens et al. 2004; Perez et al 2009; Kirov et al. 2014); 2) Тирамид-биотин, Стрептавидин-HRP, Тирамид-биотин, Стрептавидин-FITC (Nakao et al. 2002). Было проведено сравнение прямой системы детекции (тирамид-Cy3 и тирамид-FITC) физического и непрямой картирования (Тирамид-биотин, коротких Стрептавидин-TexRed) последовательностей для на хромосомах человека (Schriml et al. 1999). Результаты картирования ПЦР продуктов 319 п.н., 689 п.н. и 855 п.н. показали, что только непрямой метод детекции давал чѐткие сигналы на одной или обоих гомологах. В то же время, детекция сайтов гибридизации прямым методом давала высокий фоновый сигнал. Сравнение прямой и непрямой систем детекции в эффективности визуализировать короткие 48 последовательности на хромосомах растений было проведено на хромосомах R.wichurana (Kirov et al. 2014a). Было показано, что прямая система детекции не позволяла картировать гены и приводила к высокому уровня фонового сигнала. А с помощью оптимизированной непрямой системы детекции были успешно картированы фрагменты генов размером 1100 – 1700 п.н. Главными достоинствами прямой системы детекции являются короткое время детекции и более низкая, по сравнению с непрямой детекцией, стоимость. А главными недостатками – более высокий бэграунд и более низкая чувствительность. 1.5 Характеристика объектов исследования 1.5.1 Представители рода Allium 1.5.1.1 Филогенетическое положение и хозяйственное значение Род Allium входит в подсемейство Allioideae семейства Alliaceae порядка Asparagales и насчитывает около 780 видов (Friesen et al. 2006). Asparagales и Commelinanae, включающие наиболее важный род однодольных – Poaceae, являются родственными таксонами с множеством сходных морфологических и ДНК характеристик (Havey et al. 2006). Большинство видов рода Allium диплоиды и имеют гаплоидное число хромосом x=8 (Евразия и Средиземноморье), но встречаются виды и с x=7 (Северная Америка) и x=9 (Евразия). Новая классификация рода Allium была разработана Friesen с соавторами (2006). Изучив ITS фрагменты ядерной рибосомальной ДНК 196 видов рода, авторы пришли к выводу, что род состоит из 15 монофилетичных подродов и 56 секций. Кроме этого авторы отметили очень большое (>40%) генетическое расстояние внутри рода. Подобное значение в других подсемействах характерно для наиболее дивергировавших родов и даже семейств. Согласно этой классификации A. cepa, A. fistulosum, A. vavilovii и A. altaicum входят в состав одной секции - Cepa, а A. schoenoprasum в состав секции Schoenoprasum подрода Cepa (по традиционной классификации Rhirizideum). 49 Род Allium включает важные продовольственные культуры: лук репчатый (Allium cepa), чеснок (A. sativum), лук-порей (A. ampeloprasum), шнитт-лук (A. schoenoprasum) и лук-батун (A. fistulosum). Лук репчатый – это вторая по важности овощная культура в мире после томата (United Nations Food and Agricultural Organization (FAO, 2010)). Лук репчатый употребляется повсеместно в пищу в самом разнообразном виде. Повышенным вниманием относительно других культур A.cepa пользуется и благодаря возросшему интересу к правильному питании. Лук батун, A. fistulosum, является не только широко возделываемой культурой, но и потенциальным источником генов устойчивости к ржавчине (Urocystis cepulae Frost) (Jones et al. 1963), шейковой гнили (Botrytis squamosa Walker), луковой мухе (Hylemyia antique Bouche), корневой гнили (Phoma terrestris E. M. Hans.) (Porter and Jones, 1933), устойчивости к холоду, a также генов, контролирующих высокое содержание сухого вещества (Van der Meer, 1978). Шнитт-лук (A. schoenoprasum) – самый небольшой по габитусу представитель рода. Интересной особенностью A. schoenoprasum является малый размер генома (7.7 пг/1С), второй в роде луковых после A.altyncolicum Friesen. Геном A. schoenoprasum в два раза меньше генома A.cepa (16 пг/1С) и в полтора A.fistulosum (11.7 пг/1С), несмотря на их очень близкое родство (Friesen et al. 2006). A. schoenoprasum обладает важнымии фармацевтическими свойствами сходными со свойствами чеснока. A. schoenoprasum обладает положительным действием на кровеносную систему (Rattanachaikunsopon and Phumkhachorn, 2008) и имеет высокую антиоксидантную активность (Štajner et al. 2011). С точки зрения агрономически ценных признаков шнитт-лук имеет высокую устойчивость к фузариозу донца лука (возб. Fusarium oxysporum) (Galván et al. 2008) и некоторым абиотическим факторам (холод и засуха). В связи с высокой адаптационной способностью шнитт-лук распространѐн практически по всему Северному полушарию (Кокорева и Титова, 2007). A.vavilovii и A.altaicum являются ближайшими родственниками A.cepa и A.fistulosum, соответственно. Предполагается, что A.fistulosum образовался из предковой формы A.altaicum, в 50 связи с чем A.altaicum представляет большой интерес для исследования фундаментальных вопросов (Friesen et al. 1999) видообразования, эволюции и доместикации представителей рода Allium. Наличие широкого спектра хозяйственно-ценных признаков у диких (напр. A. roylei, A. vavilovii) и культурных (A.fistulosum, A.schoenoprasum) видов побуждают к проведению работ по открытию и секвенированию новых генов, что позволило бы создавать маркеры и вести МОС - селекцию. Последняя, в свою очередь, создаст платформу для более эффективного и быстрого получения новых сортов и гибридов между A. cepa и другими видами и сортами. 1.5.1.2 Сравнительная характеристика геномов A.cepa и A.fistulosum Даже немногочисленные проведенные на сегодня исследования позволяют сделать вывод о существенной разнице в организации и структуре генома A.cepa и A.fistulosum. Эти данные получены на основе молекулярного и хромосомного анализа генов и повторяющейся ДНК, сравнения генетических карт и анализа мейоза у межвидовых гибридов. Наиболее изученными генами в плане их организации у различных представителей рода луковых являются гены рРНК, а именно 5S и 45S рРНК локусы. Как правило, гены 45S рРНК локализуются в части хромосомы, отделѐнной вторичной перетяжкой (NOR, или спутник). Кластеры 5S и 45S рРНК генов весьма мобильные и достаточно протяжѐнные и могут быть легко визуализированы с помощью методов молекулярной (FISH) и классической (Ag окрашивание) цитогенетики. В связи с этим они являются популярным инструментом сравнительной геномики (Schubert and Wobus, 1985; Schubert, 1984). A. cepa и A. fistulosum различаются по количеству и месту локализации локусов 5S и 45S рРНК. A. cepa имеет два локуса 5S, локализованные на хромосоме 7. Кроме этого, Shibata с соавторами (2002) доказали, что сиквенсы, содержащиеся в этих двух локусах имеют разную длину и состав, что обусловлено частичной дупликацией нетраскрибируемого спейсера и инсерцией 51 уникальной последовательности. В то время как A. fistulosum имеет только один локус на той же хромосоме (Shibata and Hizume, 2002). Дупликации затронули и локус 45S рРНК, который так же находится на двух хромосомах A. cepa и A. fistulosum (хромосомы 6 и 8), одна из которых (хромосома 6) спутничная. Размер единицы повтора отличается между видами и составляет 11.8 и 13.1 тыс.п.н. для A. cepa и A. fistulosum, соответственно (Havey, 1992). По данным работы других авторов A. fistulosum не имеет дополнительного сайта на хромосоме 8 (Ricroch et al. 1992). Такое разногласие может быть связано с разным материалом, используемым для исследований, и высокой мобильностью кластеров рРНК генов (Schubert and Wobus, 1985). По генетическому картированию ценных признаков лука репчатого A.cepa интенсивно ведутся исследования преимущественно группами из США, Новой Зеландии и Японии (alliumgenetics.org). Для A. cepa разработаны несколько генетических карт, содержащих различные типы маркеров: SNP, EST-SSR, SSR (Martin et al. 2005), RFLP, RAPD, (King et al. 1998), AFLP (Van Heusden et al., 2000) и TSA (маркеры на основе полиморфизма в собранных из ридов транскриптах) (Duangjit et al. 2013). Генетические карты для A. fistulosum до последнего времени ограничивались существенно меньшим числом и типом маркеров, чем для A.cepa (Song et al., 2004; Tsukazaki et al., 2008; Ohara et al., 2005). Созданы AFLP карта, содержащая так же CAPS и 13 SSR маркеров (Ohara et al., 2005), и генетическая карта, состоящая исключительно из SSR маркеров (Tsukazaki et al., 2008). Современные методы секвенирования следующего поколения и программное обеспечение позволяют получать и анализировать большие объемы данных как генома, так и транскриптома. В 2015 году Tsukazaki с соавторами впервые провели секвенирование транскриптома A. fistulosum, что позволило им «собрать» более 50 000 сиквенсов транскриптов. Полученные данные были использованы для поиска SNP и построения генетической карты, содержащей 364 маркера, из которых 190 - это картированные транскрипты A. fistulosum (Tsukazaki et al. 2015). К сожалению, недостаточное количество 52 ортологичных маркеров в этой генетической карте и генетической карте A.cepa (Duangjit et al. 2013) не позволили сравнить генетическое положение генов и определить степень синтении между двумя геномами. Более ранние попытки сравнения организации геномов A. cepa и A. fistulosum на основе карт генов были предприняты в 1994 году при картировании гена глутамат оксалоацетат трансаминазы (Got) с помощью RFLP и моносомик дополненной линии (Shigyo et al., 1994). Позднее полный набор дополненных линий A. cepa с хромосомами A. fistulosum (Shigyo et al. 1996) и A. fistulosum c хромосомами A. cepa (Yaguchi et al., 2009), стал доступен, что позволило проводить сравнительный анализ локализации отдельных генов в группах сцепления и ассоциировать группы сцепления с конкретной хромосомой. Благодаря созданию в 2008 году карты сцепления A. fistulosum с использованием 42 общих для A. cepa и A. fistulosum маркеров, появилась возможность более подробного сравнения организации геномов этих двух видов (Tsukazaki et al. 2008). 212 A. fistulosum и 42 A. cepa маркеров были распределены между 17 группами сцепления. Для соотнесения групп сцепления и 8 пар хромосом A. fistulosum авторы использованы следующие два подхода: 1) скрининг моносомнодополненных линий (A. fistulosum - A. cepa) с картированными маркерами обоих видов, что позволило 12 из 17 групп сцепления отнести к 7 хромосомам A. cepa; 2) скрининг аллотриплоидных A. fistulosum моносомно дополненных линий с 139 из 212 маркерами A. fistulosum, что позволило 11 из 17 групп сцепления отнести к 5 хромосомам A. fistulosum. Полученные результаты свидетельствуют о значительной макросинтении между этими видами. Среди 16 групп, соотнесѐнных с хромосомами A. fistulosum, только 11 были соотнесены, используя линии, дополненные одиночными хромосомами A. fistulosum. Оставшиеся 5 групп сцепления, были соотнесены только с хромосомами A. cepa, и на основе этого был сделан вывод об их принадлежности к соответствующим хромосомам A. fistulosum, т.е. авторы полагали, что макросинтения между двумя этими видами распространяется на все группы сцепления. Однако, для 53 подтверждения этого предположения, необходимо детальное физическое картирование общих последовательностей. Прямое доказательство присутствия различий между двумя исследуемыми видами обеспечивают результаты анализа мейоза, как самих этих видов, так и гибридов между ними. Отличие мейоза A. fistulosum от A. cepa и большинства других растений заключается в особенном распределении у A. fistulosum точек рекомбинации вдоль хромосомы, проявляющихся в виде хиазм. У лука репчатого хиазмы распределены случайно в каждом биваленте. У A. fistulosum хиазмы локализованы строго прицентромерно. Такое расположение хиазм у A. fistulosum обуславливает формирование кросс бивалентов. Подобные биваленты встречаются в 94% случаев, оставшиеся 6% приходятся на биваленты с рандомизированным распределением хиазм, число которых зависит от сорта (Emsweller and Jones, 1945). Гибриды между двумя этими видами были получены разными авторами с использованием разных сортов (Levan, 1941; Emswellers and Jones, 1935; Maeda, 1937). В F1 детектируются нарушения в синапсисе хромосом, которые свидетельствуют о значительных интра- и интерхромосомных реорганизациях, отличающих геномы этих двух видов (Levan, 1941). Частота образования бивалентов сильно различалась между гибридами, полученными Emsweller и Jones (1935) и Maeda (1937) и Levan (1941). У первых двух авторов этот показатель был 70%, в то время как у Levan (1941) 2% и значительно больше мультивалентов. Emsweller и Jones (1945) продемонстрировали, что все биваленты в F1 были ассиметричны и имели рандомизированное распределение хиазм. Позже, более подробный анализ, проведѐнный с помощью GISH (Genomic in situ hybridization), показал, что частота хиазм составляет в среднем 11 на клетку и преобладающее большинство хиазм локализовано в дистальной и интерстициальной части хромосомы (Stevenson et al. 1998). Мнения всех авторов сходятся в том, что распределение точек рекомбинации у гибридов F1 ближе к A. cepa (Stevenson et al., 1998). Сравнение типа распределения хиазм у BC1 растений показало, что 10 из 17 растений имели строго локализованные хиазмы, как у A. 54 fistulosum, у 7 растений распределение хиазм было рандомизированное. Аналогичный анализ F2 показал следующие расщепление: 9 растений имели все биваленты с рандомизированным распределением хиазм, 2 растения имели 6 бивалентов с рандомизированным распределением и 2 с локализованным распределением хиазм, 2 растения имели 5 бивалентов с рандомизированным распределением и 3 с локализованным распределением хиазм (Emsweller и Jones, 1935). Обнаруженное расщепление послужило поводом выдвижения гипотезы о генетическом детерминировании локализации хиазм у этих видов (Emsweller и Jones, 1935). Вторая гипотеза, объясняющая кросс-биваленты, связана с уникальной организацией хромосом A. fistulosum, которая заключается в проксимальном расположении ген-богатых областей и наличии в дистальной области крупного видоспецифичного повтора (Khrustaleva et al. 2012). Таким образом, различия в распределении хиазм, так же как и нарушения в мейозе у гибридов F1 свидетельствуют о хромосомных реорганизациях, отличающих геномы A.cepa и A.fistulosum. Среди них разные авторы выделяют крупные делеции, инверсии и транслокации (Emsweller & Jones, 1938; Levan, 1941; Peffley, 1986). С другой стороны, данные генетического картирования ортологичных SSR маркеров указывают на высокий уровень макросинтении между двумя геномами. Дальнейшие исследования по генетическому и физическому картированию ортологичных маркеров и генов позволят пролить свет на тип хромосомных перестроек и их количество. 1.5.2 Rosa wichurana 1.5.2.1 Филогенетическое положение и преимущества для цитогенетических исследований Род Rosa относится к семейству Rosaceae, которое содержит многие значимые виды растений, относящиеся к родам Malus, Prunus, Pyrus, Fragaria, Rubus, Sorbus, Cotoneaster and Crataegus. Внутри рода Rosa ботаники выделяют около 200 видов и более 20000 сортов (Wissemann, 2003). Однако, только 8 – 20 55 видов были вовлечены в образование всех современных сортов (Vries and Dubois, 1995; Reynders-Aloisi and Bollereau, 1995; Gudin, 2000). Розы являются наиболее широко культивируемыми декоративными растениями в мире. Rosa wichurana относится к подроду Rosa, секции Synstylae. Данный вид очень удобен для цитогенетических исследований, т.к., в отличие от многих других видов рода Rosa, R. wichurana является диплоидным видов (2n=14, Рисунок 9B), имеет интенсивно растущие и крупные апикальные меристемы (Рисунок 9А) и долгий период цветения (2-3 месяца). Кроме этого, цветки имеют простой венчик и большое число пыльников (Рисунок 12С), что облегчает изучение мейоза в материнских клетках пыльцы (Kirov et al. 2015b). Рисунок 12. Важные особенности Rosa wichurana для цитогенетических исследований: А. интенсивно растущие апикальные меристемы; B. диплоидное число хромосом; C. Цветки с десятками пыльников. R. wichurana – один из немногочисленных видов, вовлеченных в образование более 20000 современных сортов розы (Vries and Dubois, 1995). Еще одной причиной выбора данного вида для исследований является его интенсивное использование и в современных селекционных программах, т.к. R. wichurana является ценным источником генов устойчивость к мучнистой росе (Dugo et al. 2005; Leus et al. 2009; Moghaddam et al. 2012). 56 1.5.2.2 Цитогенетические исследования генома Цитогенетическое изучение представителей рода Rosa ограничивается лишь исследованиями кариотипа с помощью подсчета числа хромосом, измерения центромерного индекса, размера хромосом и построением идиограмм (Hurst, 1925, 1927; Wylie, 1954; Darlington and Wylie, 1955; Price at al., 1981; Liu and Li, 1985; Subramanian, 1987; Ma and Chen, 1991, 1992; Ma et al., 1997a, b; FernandezRomero et al., 2001; Akasaka et al., 2002, 2003; Liu et al., 2008, Jian et al. , 2013a, 2013b). Несмотря на высокое хозяйственное значение, молекулярно- цитогенетические исследования видов рода Rosa с использованием метода FISH немногочисленны. Это обусловлено несколькими факторами: 1) все исследованные виды имеют достаточно маленькие и трудно различимые хромосомы (Akasaka et al., 2002, 2003; Kirov et al., 2014); 2) большинство видов – полиплоиды с числом хромосом 2n=4x=28 до 2n=10x=70 (Vamosi and Dickinson, 2006; Roberts et al., 2009; Jian et al., 2010); 3) низкий митотический индекс в апикальных и корневых меристемах (Ma et al., 1996). Все исследования хромосом с помощью FISH были проведены с использованием генов 45S и 5S в качестве пробы (Ma et al., 1997b; Fernandez-Romero et al., 2001; Akasaka et al., 2002, 2003; Lim et al. 2005; Jian et al. 2012; Kirov et al. 2014a, 2015a, 2015b). Этими исследованиями было показано, что кариотип практически всех исследованных диплоидных видов содержит одну пару хромосом, несущую 45S гены рРНК в ядрышко-образующем регионе (ЯОР), или спутнике (Ma et al., 1997; FernándezRomero et al., 2001; Jian et al. 2012; Kirov et al., 2014a). Исключение составляет картиотип R. foliolosa, содержащий три пары хромосом с 45S рРНК (Akasaka et al. 2003). Fernández-Romero et al. (2001) использовали FISH с генами 45S и 5S рРНК для исследования происхождения и эволюции диплоидных, триплоидных и тетраплоидных предковых видов подрода Rosa. Авторы показали, что виды R. chinensis и R. gallica являются автотриплоидом и аллотетраплоидом, соответственно (Fernandez-Romera et al. 2001). В отличие от 45S, гены 5S рРНК локализованы в двух парах хромосом у видов из «группы А» (Akasaka et al., 2002). Один из локусов 5S локализуется на той же хромосоме, что 45S (Akasaka et al., 57 2002; Lim et al. 2005). Одновременное использование двух проб в одном FISH эксперименте позволяет эффективно проводить идентификацию нескольких хромосом у видов рода Rosa. Так, двуцветная FISH была успешно использована в изучении поведения хромосом R.canina (2n=5x=35) в мейозе (Lim et al. 2005). Авторами было показано, что два из пяти субгеномов вовлечены в образование бивалентов и рекомбинация не возникает между хромосомами остальных трех субгеномов. Это исследование ясно продемонстрировало ценность цитогенетических маркеров, основанных на FISH, в изучении хромосом видов Rosa. Для преодоления трудностей молекулярно-цитогенетических исследования геномов видов Rosa, связанных со сложностью приготовления препаратов хромосом, был разработан и успешно протестирован на хромосомах R.wichurana метод ―SteamDrop‖ (Kirov et al. 2014a,b). Данный метод совместно с оптимизированным протоколом Tyramide-FISH позволили расширить возможности молекулярно-цитогенетического изучения генома рода Rosa и провести картирование коротких фрагментов ДНК, размером от 1.1 тыс.п.н. (Kirov et al. 2014a, 2015a). 58 ГЛАВА 2. МАТЕРИАЛЫ И МЕТОДЫ 2.1 Растительный материал Растения R. wichurana были выращены в условиях теплицы без искусственного контроля света и температуры. Семена A.cepa (сорт Халцедон), A.fistulosum (сорт Русский зимний), A.schoenoprasum (сорт Медонос) были куплены в фирме «Гавриш». Семена A.altaicum Альвес были куплены в фирме Седек. Растения A.vavilovii, F1 AcepaxA.vavilovii и A.proliferum были предоставлены ВНИИСОК. Растения A.wakegi были предоставлены Dr. Masyoshi Shygio (Yamaguchi University, Faculty of Agriculture, Japan). 2.2 Приготовление препаратов хромосом 2.2.1 Митотические хромосомы Для приготовления преператов хромосом семена A. cepa, A. fistulosum, A.altaicum и A.schoenoprasum были выращены на фильтровальной бумаге при температуре 24С. Затем проросшие семена (длина корня около 1см) обработывали гидроксимочевиной: помещали в раствор 0,75мМ гидроксимочевины на 18-20 часов; затем промывали и переносили в воду на 4 часа и затем переносили в 0,05% раствор колхицина на 4 часа, после чего проростки фиксировали фиксаторе в 3:1 (100% этанол:ледяная уксусная кислота) в течение 30 мин. и переносили в 70% спирт для последующего использования или хранения на 20°С. Для A.vavilovii, F1 Acepa x A.vavilovii, A.proliferum и A.wakegi интенсивно растущие корни были собраны со взрослых растений в теплице и предобработаны закисью азота в течении 3-4 часов при давлении 1атм. Затем корни фиксировали 30 мин. в 3:1 (100% этанол : ледяная уксусная кислота) и перенесены в 70% спирт для последующего использования или хранения на -20°С. Для приготовления препаратов хромосом R.wichurana были собраны апикальные меристемы и обработаны согласно протоколу Ma et al. (1996). 59 Фиксированные корни (апикальные меристемы) были промыты в воде 20 мин. и в 10 мМ цитратном буфере (рН 4.8) и выдержаны в смеси ферментов 0.5% целлулаза, 0.5% пектиназа и 0.5% цитогеликаза в 10 мМ цитратном буфере при 37°С. Время обработки варьировало в зависимости от вида (Таблица 2). Таблица 2. Время обработки ферментами для используемых видов Вид Allium виды F1 A.cepa A.vavilovii R. wichurana Время обработка Источник ферментами метафаз 75 мин Корни из проросших семяна х 90 мин Корни с молодых растений 120 мин Апикальные меристемы Препараты метафазных хромосом были приготовлены ―SteamDrop‖ согласно следующему протоколу (Kirov et al. 2014b): методом 1. Сделать клеточную суспензию, аккуратно ударив несколько раз по пробирке 2. Добавить 500 мкл воды и перемешать 3. Центрифугировать 45 сек. 12000 rpm 4. Удалить супернатант с помощью пастерки 5. Добавить 500 мкл. 96% этанола и перемешать 6. Центрифугировать 45 сек. 12000 rpm 7. Удалить супернатант 8. Добавить 40-200 мкл (в зависимости от плотности клеточной суспензии) 100% спирта или 550 мкл для хранения (-20С) 9. Капнуть 10 -11 мкл клеточной суспензии на предметное стекло 10. Когда поверхность станет гранулярной, капнуть 20 мкл фиксатора (9:1, 5:1, 3:1, 2:1 или 1:1 (смотри текст). 11. Спустя 35-45 сек выдержать стекло над водяной баней нижней стороной вниз от 1 (для мелких хромосом) - 8 (для крупных хромосом) секунд 12. Подсушить 2 секунды и капнуть 5 мкл более концентрированного фиксатора. 60 13. Подождать15-20 сек, провести над баней нижней стороной вниз и быстро высушить под потоком воздуха. 2.2.2 Пахитенные хромосомы Для приготовление пахитенных хромосом A.cepa были отобраны пыльники в подходящей стадии и фиксированы в 3:1 (100% этанол : ледяная уксусная кислота) в течение 4-5 часов при комнатной температуре и перенесены в 70% спирт для последующего использования или хранения на -20°С. Препараты готовили методом ―SteamDrop‖ согласно вышеизложенному протоколу с использованием фиксаторов 1:1/100% уксусная кислота. Для приготовление пахитенных хромосом R.wichurana с бутонов с подходящим размером гипантия были удалены лепестки и чашелистики и проведена фиксация в 3:1 (100% этанол : ледяная уксусная кислота) в течение 6-8 часов при комнатной температуре, а затем бутоны были перенесены в 70% спирт для последующего использования или хранения на -20°С. Препараты готовили модифицированным методом ―SteamDrop‖ с использованием фиксаторов 1:1/100% уксусная кислота. Модификация заключалась в добавлении капли (400 мкл) 60% или 45% уксусной кислоты после последнего действия пара, не позволяя препарату высохнуть. Затем препарат помещался на нагревательный столик (42°С) и капля уксусной кислоты перемещалась с помощью препаровальной иглы в течении 15-30 секунд. После этого, остатки уксусной кислоты сливали с препарата и препараты выставляли вертикально на фильтровальную бумагу для подсыхания при комнатной температуре. 2.3 Выделение ДНК ДНК выделяли согласно протоколу Rogers and Bendich (1985) из молодых листьев для R.wichurana и проросших корней для Allium. 61 2.4 Получение проб для Tyramide-FISH и FISH Для создания проб для физического картирования генов на хромосомах видов Allium и Rosa wichurana были подобраны праймеры на целевые гены с помощью программы Primer 3.0 Plus (http://www.bioinformatics.nl/cgi- bin/primer3plus/primer3plus.cgi). Последовательности генов и их номера в NCBI, которые были использованы для подбора праймеров, представлены в Таблице 1. Температура отжига для всех праймеров составила 58°С. Фрагменты генов PAL и P5CS были изолированы согласно протоколу Razavi et al. (2011), используя EST Prunus persica из базы данных Rosaceae (Genome Database of Rosaceae, Jung et al. 2008). Для генов MDAR, AD1 и Nar праймеры были подобраны на сиквенсы, найденные в базе данных (https://iant.toulouse.inra.fr/plants/rosa/FATAL/cgi/ROSCH.cgi) с помощью BLAST с EST сиквенсами из генетической карты тетраплоидной розы (Gar et al. 2012, Таблица 3). 62 Таблица 3. Информация о праймерах Вид Ген Allium cepa Allium cepa Allium cepa Allium cepa Allium cepa Rosa wichura na Rosa wichura na Rosa wichura na Rosa wichura na Rosa wichura na Rosa wichura na Rosa wichura na Rosa wichura na Rosa wichura na Аллиназа корня I (AOB249) Аллиназа луковицы Rosa wichura na Rosa wichura na Праймеры, 5‘-3‘ CAGAATTCACCAAGGCGAAGGG ACCGGATAATAAGGGGCATGAG GGTCATCTCCCTTTCACCAA TGATCAAACTCAAACGCAC CCACGGGTCGAGATGGGATGGA AAAAACGCCCTTCGCCTTGGTG AAAATGGAGCTAAATCCTGGTG CATAATGCATCACAGCACTGAA TCCATGATCAGGAAACGCTAC CGAACGCACCCATTAACAAT TGCACTACCAATCCATCCAA TGCCAAGTAACATTTGGCTTT Номер в NCBI Локус устойчивости к мучнистой росе О 1 (MLO1) Локус устойчивости к мучнистой росе О 2 (MLO2) Локус устойчивости к мучнистой росе О 3 (MLO3) Пирролинe-5Карбоксилат Синтаза (P5CS) Фенилаланин аммония лиаза (PAL) TCAAAGCCACCAAAAATTCA TGCTTCTTAGCCGTGTGATG JX847131.1 AGGATTTCAAGGTCGTGGTG TGGTCGGCTAGCATTTTTCT JX847132.1 AAAACACCAACATGGGCAGT TTCCGAAAATCAAAGGTCGT JX847133.1 GCTGGCATCCCTGTTGTTAT CTTCGGATCGCTAATGAAGC FC865594.1 ACCACTGGKTTTGGTGCWAC CCYTTGAASCCATAATCCAA АТФаза (AAA2) GTTCCCTTTGTCATTGCAG ACGGCCTCTTCATCAATT Монодегидроаскорб ат редуктаза (MDAR) Маннозил гликопротеин эндобета-маннозидаза (MGM) Аннексин D1 (AD1) GAGGCGGTATGGTTAATTT AAACTTGGGCTTTGGTGA Scaffold_2:20,819, 497..20,820,563 (Prunus persica) LG7: 21485846..2148109 0 (Fragaria vesca) CF349908.1 Нарингенин (Nar) Аллиназа корня II Синтаза фактора слезотечения (LFS) Халкон синтетаза (CHS) Орцинол-О-метил трансераза (OOMT) AA451570.1 L48614.1 AB111058.1 AB094593 AY221245.1 AJ786302 CGGCATGGAAAATGAGTCAA GAACAAAGGGATCTGCCA LG6: 5180627..5186123 TAAAGTACCAGCATGCGTT CCATAAACAACACCAACTCA EC586738.1 AATTCCGATCATTTCCCTCTC CCTTGCTCATTCTTCTTCTT BQ105585.1 Фрагменты генов были амплифицированы с помощью следующей ПЦР программы: 63 ПЦР программа: 1) 94º - 5 мин 2) 94º - 30 сек 3) 58 º - 45 сек х 35 4) 72º - 60 сек 5) 72º - 5 мин 6) 4º - ∞ ПЦР продукты были клонированы в pGEM-T easy (Promega) или pCR2.1TOPO (Invitrogen) согласно инструкции производителя. Как минимум три колонии с ожидаемым размером вставки были отобраны и использованы для выделения плазмиды и последующего секвенирования. Секвенирование проводили на секвенаторе ABI 3130 XL (Applied Biosystem) после реакции секвенирования с Big Dye Terminator v 1.1. Cycle Sequencing Kit (Applied Biosystem). Выделенная ДНК плазмид была помечена с помощью DIG-Nick Translation Mix или Biotin-Nick-Translation Mix (Roche) согласно прилагаемой инструкции. Повтор HAT58, CAT36 и Afi были помечены с помощью ПЦР с геномной ДНК A.fistulosum и PCR labeling Mix (Roche), используя следующие праймеры (Таблица 4). Таблица 4. Праймеры на повторы Название повтора HAT58 CAT36 Afi11 Праймеры, 5‘-3‘ Номер в NCBI F:AAAAACATCTTCCAACAGCATAAA н/п* R:TGCATGAAAAGACAGCGTTT F:TCCCACCTAAATTACGGACA н/п R:AAATAGCGGCTTCTGCACTA F: AAAGGTTCATGCCTGCTTTC AB735740 R: TTTTACGGCATGCGATACCT *н/п – номер не присвоен Плазмиды pTA71 (Gerlach and Bedbrook 1979) и pSCT7 (Lawrence and Appels. 1986) были помечены с помощью Ник-трансляции и использованы для визуализации сайтов 45S и 5S рДНК. 64 Пробы на теломерный повтор Arabidopsis типа (CCCTAAA)3 и повтор HAT58 (5‘-TGCATGAAAAGACAGCGTTTAGAGTTTTTATGC-3‘) были синтезированы и помечены TAMRA на 5‘ конце компанией Синтол (Москва, Россия). 2.5 FISH и ND-FISH (nondenaturating FISH) Предобработку препаратов проводили с помощью 4% параформальдегида в 2xSSC с последующей дегидратацией в 70%, 80% и 96% спирте. Гибридизационная смесь содержала 50% (v/v) деионизированного формамида, 10% (w/v) декстрансульфата, 2 × SSC, 0.25% натрий додецил сульфата, 1.25 ng/µl ДНК пробы. Смесь денатурировали при 80°C 5 мин и быстро помещали на лед на 5 мин. 80 µl смеси раскапывали на стѐкла, накрывали покровным стеклом (24 × 56 мм), и денатурировали 5 мин. при 75°C. Жесткость гибридизации – 78%. Гибридизация проводилась в течении ночи во влажной камере при 37C. Затем стѐкла промывали в 2xSSCT (2xSSC + 0.05% Tween 20) при 37 C и 2 раза по 5 мин в 2xSSC. Затем стѐкла помещали в 0.1xSSC при 53 C на 7 мин два раза, в 2xSSC при 37 C, 5 мин. Жесткость отмывки – 81-83%. Детекцию проводили с помощью стрептавидин-Cy3 и антидиг-FITC для детекции проб, меченных биотином или дигоксигенином, соответственно. ND-FISH проводили согласно протоколу Cuadrado and Jouve (2010). 2.6 Tyramide-FISH Предобработку препаратов проводили так же, как и для FISH (Глава 2.5). 2.6.1 Прямой метод детекция Меченные биотином пробы детектировали с помощью Стрептавидин-ПХ (Streptavidin-HRP, PerkinElmer) в соответствии с прилагаемым протоколом. Стѐкла промывали в свежем TNT буфере (0.1M Tris HCl, 0.15M NaCl pH 7.5, 0.05% Tween 20) при комнатной температуре 5 мин, выдерживали в TNB буфере (0.1M Tris-HCl, 0.15M NaCl, 0.5% blocking reagent) при комнатной температуре 30 мин во влажной камере. После блокирования в TNB, слайды выдерживали в 65 Стрептавидин-ПХ при комнатной температуре 45 мин во влажной камере, затем промывали в TNT 3 раза по 5 мин при комнатной температуре. Для детектирования тирамидами готовили раствор 1:50 Тирамид–Cy3 (PerkinElmer, NEL744001KT) с добавлением 10% (w/v) декстрансульфата. 200 µl данного раствора раскапывали на стѐкла, выдерживали при RT 7-10 мин, и затем удаляли промыванием 3 раза по 5 мин в TNT при RT и проводили окрашивание хромосом с помощью DAPI (100 мкг/мл) смешанным с Vectashield (1:20, Vector Laboratories, Burlingame, USA). Морфометрию хромосом и определение относительного положения сигнала на хромосоме проводились с помощью программы MicroMeasure (Reeves, 2001). 2.6.2 Непрямой метод детекции При непрямом методе детекцию проводили с использованием Тирамид конъюгированных с биотином, растворѐнных в amplification diluent (1:25) (PerkinElmer) без добавления декстран сульфата. После отмывки в TNT буфере при комнатной температуре в течение 5 мин, стѐкла выдерживали в TNB буфере при RT 30 мин во влажной камере. После блокирования в TNB, на препараты наносили антитела стрептавидин-TexRed (1:100) и инкубировали при 37С 1час. Затем промывали в TNT 3 раза по 5 мин при комнатной температуре и проводили окрашивание хромосом с помощью DAPI (100 мкг/мл) смешанным с Vectashield (1:20, Vector Laboratories, Burlingame, USA). 2.6.3 Многоцветная Tyramide-FISH Биотин- меченые фрагменты генов PAL и MGM и дигоксигенин -меченый фрагмент гена MLO2 были использованы для многоцветной Tyramide-FISH. Детекцию проводили последовательно. Первым детектировали биотин-меченые пробы согласно описанному инактивировали 3% протоколу (Глава 2.6.2). Стрептавидин-ПХ перекисью водорода в течение 20 мин. Затем после 66 промывки препараты инкубировали со стрептавидин-Cy3 вместе с антидигоксигенин-ПХ (1:100, Roche, 11207733910). После этого препараты промывали три раза в TNT буфере при комнатной температуре 5 мин и снова проводили детекцию с помощью тирамид-биотин (1:25). Затем препараты снова промывали три раза в TNT буфере при комнатной температуре 5 мин и инкубировались в TNB буфере при RT 30 мин во влажной камере. После блокирования в TNB, на препараты наносили антитела стрептавидин-FITC (1:100, LifeTechnologies, SA100-02) и инкубировали при 37С, 1 час. Затем промывали в TNT 3 раза по 5 мин при комнатной температуре и проводили окрашивание хромосом с помощью DAPI (100 мкг/мл) смешанным с Vectashield (1:20, Vector Laboratories, Burlingame, USA). 2.7 Флуоресцентная микроскопия и анализ изображения Препараты после FISH и Tyramide-FISH были проверены на микроскопе Zeiss Axio Imager M1 (Carl Zeiss MicroImaging, Jena, Germany). Фотографии были сняты камерой Axio Cam MRm с помощью программы AxioVision 4.6. Яркось/контраст были отрегулированы в программе AxioVision 4.6 или Photoshop CS7. 2.8 Изолирование повтора HAT58 Повтор HAT58 был выделен из данных секвенирования следующего поколения, доступных для A.fistulosum в базе данных NCBI (SRX268217), с помощью программы RepeatExplorer (Novak et al. 2013). Минимальная длина перекрытий между ридами для построения контига была 40 п.н., а для кластеризации 55 п.н.. 2.9 Генетическое картирование HRM был проведен согласно Deryckere et al. (2013) с использованием 0.86 LightCycler 480 High Resolution Melting Master Mix (Roche). Для генотипирования была использована программа LightCycler 480 Gene Scanning. С помощью ПЦР с геномной ДНК растений картируемой популяции и их родительских форм были 67 амплифицированы ДНК фрагменты, содержащие однонуклеотидные различия (SNP). Паттерны расщепления для каждого маркера были добавлены к существующим данным, описанным Moghaddam et al. (2012). Для картирования были использованы следующие параметры: используя сцепление с частотой рекомбинации меньше, чем 0.49 и LOD выше, чем 1. 68 ГЛАВА 3. РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ 3.1 SteamDrop – новый универсальный метод приготовления препаратов хромосом растений 3.1.1 Процесс разброса хромосом растений Ключевым фактором, определяющим успех физического (цитогенетического) картирования, является качественные препараты хромосом. Нами впервые было проведено детальное исследование процесса разброса хромосом растений после нанесения клеточной суспензии на стекло. Процессы, происходящие с раскапанной на стекло суспензией клеток, мы наблюдали с помощью метода фазового контраста. Микроскопия клеток была проведена после нанесения на стекло спиртовой суспензии клеток и еѐ частичного высыхания с последующим добавлением капли фиксатора 3:1 (Рисунок 13). Эксперимент был проведен при комнатной температуре (23-25°С) и влажности воздуха 45-55%. Результаты данного эксперимента показали, что в период с момента добавления фиксатора и образованием цитоплазмы мениска и над клетками происходит разрушение клеточной стенки. быстрое Хромосомы исчезновение остаются не разбросанными (Рисунок 13). Рисунок 13. Процесс разброса хромосом A. fistulosum после капания фиксатора. Действие пара отсутствует. 69 В следующем эксперименте, после нанесения клеток, добавления фиксатора и образования мениска, была проведена обработка клеток на стекле паром от водяной бани, нагретой до 55°С. Эксперимент был проведен на клетках A.fistulosum и Humulus japonicus. H.japonicus был использован в качестве растения с маленьким хромосомами вместо R.wichurana, т.к. меристемы R.wichurana имеют низкий митотический индекс, что затрудняет быстрое обнаружение клеток на стадии метафазы. В противоположность результатам первого варианта (без пара), происходил быстрый разброс как крупных хромосом A.fistulsum, так и мелких хромосом H.japonicus (Рисунок 14). Рисунок 14. Процесс разброса хромосом A.fistulosum (A-H) и H.japonicus (A‘-H‘) после капания фиксатора: A,B и A‘,B‘ – образование мениска над клеткой (II – III этапы), C, D и C‘-E‘ – клетки набухают и цитоплазма становитcя прозрачной (III этап), E-H и F‘-H‘ - разброс хромосом и иммобилизация клеток к стеклу (III – IV этапы). Этапы C-H и C‘-H‘ происходили под действием пара. Линейка – 10 мкм 70 Проведенные микроскопические наблюдения позволили, условно, разбить весь процесс разброса хромосом под действием пара на четыре этапа: I) После добавления фиксатора часть клеток остаѐтся иммобилизированной к поверхности стекла, а часть перемещается дальше от центра за счѐт силы растекающегося фиксатора (продолжительность 5 – 10 сек.); II) Уровень фиксатора уменьшается, цитоплазма становится прозрачной и хромосомы частично видимыми (продолжительность – 15 – 20 сек.); III) Фиксатор продолжает испаряться и над клетками образуется мениск, который давит на клетку (продолжительность – 10-15 сек.). На этом этапе при подаче пара к поверхности стекла, клетки начинают быстро набухать, форма с прямоугольной меняется на округлую, происходит разброс хромосом и исчезновение цитоплазмы. Разброс хромосом занимает всего 1-5 сек, в зависимости от размера хромосом. IV) Фиксатор испаряется полностью, хромосомы и ядра иммобилизируются на стекло. В эту фазу при естественном испарении, перераспределение фиксатора на стекле обуславливает сильный разброс хромосом. Результаты проведенного наблюдения позволяют сделать вывод о динамичности событий, проходящих после нанесения фиксатора и образования мениска над клетками. Итог этих событий – разрушение клеточной стенки, гидролиз цитоплазмы, разброс хромосом и их прикрепление к стеклу. Интересно отметить, что Перечисленные выше этапы схожи с этапами, описанными при изучении динамики разброса хромосом человека (Deng et al. 2003). Главным практическим выводом проделанной работы стало то, что действие пара на препарат во время образования мениска значительно улучшает разброс хромосом. Чем объясняется такая динамика разброса хромосом под действием пара? Усиление гидролиза цитоплазмы под действием пара вероятно связанос повышением температуры раствора под воздествием пара, что приводит к резкому и кратковременному повышению концентрации уксусной кислоты за счет более быстрого испарения спирта (Henegariu O. et al. 2001) Но не только 71 температура является причиной усиленного гидролиза цитоплазмы, а и вода, содержащаяся в паре, смешивается с фиксатором, в результате чего высвобождается свободная энергия, которая вероятно и является движущей силой, приводящей к разбросу хромосом (Deng et al. 2003; Henegariu O. et al. 2001). Однако, нами было отмечено, что длительное действие пара (дольше 10 сек), не приводит к разбросу хромосом и гидролизу цитоплазмы. При длительном насышении водой клетки смываются к краям стекла. Действие пара до образования мениска также оказалось неэффективным. Таким образом, только совместное действие пара при температуре 55С в течение 2-3 сек и давления мениска позволяют добиться эффективного разброса хромосом и гидролиза цитоплазмы. 3.1.2 Относительная влажность воздуха и температура – критические факторы разброса хромосом растений Рядом авторов было отмечено, что кроме своевременного действия пара, существенное влияние на разброс хромосом оказывают влажность и температура воздуха. По данным этих исследований (Spurberck et al. 1996; Henegariu O. et al. 2001; Claussen et al. 2002; Deng et al. 2003) слишком высокая влажность, так же как и слишком низкая существенно снижают разброс хромосом. Для проверки взаимосвязи между влажностью воздуха и качеством разброса хромосом растений и установления оптимальных условий разброса, было проведено сравнение динамики разброса хромосом при трех вариантах относительной влажности воздуха (25–30%, 50–55% и 65–70%) при комнатной температуре. Наши исследования на растительной клетке показывают, что влажность выше 60% и температура ниже 20°С приводят к слабому разбросу хромосом и слабому гидролизу цитоплазмы. Нами были определены оптимальные условия для метода «паровой капли»: 45-55% относительной влажности воздуха и температура 2325°С. Уменьшение разброса хромосом при высокой относительной влажности, возможно, связано с перенасыщением нанесѐнного фиксатора влагой из воздуха, в 72 виду высокой гигроскопичности фиксатора. Из-за этого к моменту образования мениска концентрация уксусной кислоты слишком низка, чтобы обеспечить гидролиз цитоплазмы и разброс хромосом. Кроме этого, высокое содержание воды значительно замедляет испарение фиксатора после действия пара, обуславливая смывание клеток и высокую загрязнѐнность препарата. Приготовление препаратов при низкой относительной влажности воздуха (ниже 30%) приводило к раннему гидролизу цитоплазмы (до образования мениска) и к низкому разбросу хромосом. Проведѐнные эксперименты показывают, что разброс хромосом во многом зависит от условий (относительная влажность и температура), предшествующих действию пара. Кроме влияния на разброс хромосом некоторыми авторами было показано, что длина хромосом человека и животных, приготовленных методом капли, существенно меняется в определенных условиях влажности и температуры (Claussen et al., 2000). Для того, чтобы проверить могут ли хромосомы растений, приготовленные методом капли растягиваться, были приготовлены препараты хромосом A.cepa в условиях высокой (65-70%) и средней (45-55%) относительной влажности воздуха. Для обоих вариантов препараты были приготовлены из одной клеточной суспензии. Клетки были предобработаны гидроксимочевиной и колхицином для синхронизации клеточного цикла и остановки деления на стадии метафазы, соответственно. Результаты показали, что при средней влажности воздуха общая длина всех хромосом A.cepa составила 232±0,37 мкм, что на 30% больше, чем длина всех хромосом, приготовленных при относительной влажности 65% (163±12 мкм) (Рисунок 15). 73 Рисунок 15. Метафазные пластинки, полученные при приготовлении препаратов методом «паровой капли» при относительной влажности 50% (слева) и 65% (справа). Ранее, было проведено измерение общей длины всех хромосом A.cepa, приготовленных методом «давления» и показано, что она составляет около 140 мкм/2n (Khrustaleva and Kik, 2001), что на 60% меньше, чем в случае препаратов, приготовленных методом «паровой капли» в условиях средней относительной влажности. Возможно, данные различия обусловлены методом приготовления препаратов или способом предобработки. Данные результаты должны быть учтены при проведении исследований по кариотипированию и вычислению размера хромосом. Наши данные свидетельствуют, что хромосомы растений имеют потенциал для растяжения, что совпадает с данными, полученными на хромосомах человека (Claussen et al., 2000), а степень растяжения зависит от влажности воздуха (в случае «паровой капли»). Согласно гипотезе Claussen с соавторами (2000) при взаимодействии уксусной кислоты с белками, в том числе хромосомы, происходит реакция ацетилирования. В результате этой реакции образуется ацетат аммония, который абсорбирует воду, что и приводит к растяжению хромосомы. Если эта теория верна, то, возможно, при увеличении влажности ход реакции изменяется и ацетилирование и, соответственно, поглощение воды белками хромосомы, идут в меньшей степени. Последние 74 исследования показывают, что разброс и растяжение хромосом – это результат сложных взаимодействий между цитоплазматическим матриксом, белками хромосомы, влагой из воздуха и уксусной кислотой (Ami et al. 2014). 3.1.2 Метод «SteamDrop» («паровая капля») 3.1.2.1 Особенности и достоинства метода Изучение динамики разброса хромосом под действием пара и обнаружение ключевых факторов, влияющих на разброс хромосом, позволили создать новый метод приготовления препаратов, который был назван метод «паровой капли» (англ. steam drop method, Kirov et al. 2014b). Ключевыми особенностями и отличиями данного метода от предыдущих модификаций метода капли являются: 1. Эффективный разброс хромосом с помощью пара; 2. Возможность приготовления суспензии из одного корня; 3. Контролируемый разброс хромосом и количество цитоплазмы посредством изменения времени обработки паром и отношения кислота/спирт в фиксаторе; 4. Продолжительное время хранения спиртовой суспензии, не влияющее на качество препарата; 5. Возможность приготовления нескольких препаратов из одного корня; 6. Простой и быстрый протокол приготовления препаратов; 7. Подходит как для митотических, так и для мейотических хромосом. 3.1.2.2 Приготовление суспензии В процессе приготовления препаратов хромосом стандартным методом капли, где суспензия хромосом делается в фиксаторе (3:1 или 9:1, уксусная кислота:спирт), часто клетки начинают разрушаться под действием кислоты ещѐ до попадания на стекло. Поэтому в методе ―SteamDrop‖ вместо фиксаторов для приготовления клеточной суспензии используется 96%-100% этанол. Приготовление суспензии клеток проводится следующим образом: 1) Проводится обработка ферментами (целлюлаза и пектиназа); 2) Добавляется вода и проводится центрифугирование; 3) Остатки воды сливаются и добавляется 75 этанол; 4) Проводится центрифугирование и добавляется финальный объѐм этанола (50-100µl). Замена фиксатора на спирт позволила сохранить все клетки целостными до попадания на стекло, а так же, как показали исследования, сделала возможным сохранять клеточные суспензии в течение продолжительного времени (при -20°С) без влияние на степень разброса хромосом. Это особенно актуально, когда речь идѐт о материнских клетках пыльцы, используемых для приготовления пахитенных хромосом. Сохранение мейотических клеток на долгое время без ущерба для разброса хромосом (что происходит, если хранить цельные пыльники) делает проведение исследований с использованием мейотических хромосом (пахитенных) независимыми от условий и времени цветения. Максимальный срок хранения клеточной суспензии, без потери хромосомами способности быть разбросанными, не установлен. Известно, что суспензию клеток человека в фиксаторе можно хранить в течение нескольких лет (Henegariu et al. 2001). Возможность хранить суспензии значительно облегчает и ускоряет процесс приготовления препаратов. Хранение клеток в спирте, а не в фиксаторе, имеет менее разрушительное действие на структуру хромосомы, так как было показано, что фиксатор 3:1 может экстрагировать белки хромосомы (Dick and Johns, 1968). Экстракция гистонов нарушает структуру хромосомы и ухудшает доступность ДНК хромосомы для гибридизации ДНК пробы. Последнее особенно важно при детекции уникальных, коротких последовательностей ДНК на хромосоме (Jiang and Gill, 2006). Для уменьшения потери клеток, мы сократили число промывок до двух: одна промывка водой, для удаления остатков ферментов, буфера и продуктов гидролиза ферментами, и одна промывка спиртом для дегидратации клеток. Мы предлагаем делать обе промывки в течении 45 сек. и на максимальных оборотах (12000-13500 rpm). Данная модификация позволила преодолеть один из главных недостатков метода капли (большое количество материала, необходимое для приготовления препаратов) и, как показали испытания на ряде видов, сделала возможным приготовление нескольких препаратов (с минимальным количеством 76 метафаз с хорошим разбросом -10) из единичной корневой меристемы (Таблица 5). Таблица 5. Диаметр и длина меристем разных видов и количество препаратов, которое возможно приготовить из меристемы одного корняметодом «паровой капли». Виды Диаметр меристемы, мм 0,6-0,7 Allium виды AC x AF 1-1.5 гибрид Humulus 1-1.2 japonicus Triticale 1-1.2 Rosa 0.5-1/1.5-21 wichurana 1 – апикальная меристема Длина меристемы, мм 1.2-1.5 Препаратов на меристему 1.5-2 7-10 1.5-2 3-5 1.2-1.5 0.7-1.2/2-31 2-3 1-2/4-101 1-2 Приведѐнные расчѐты были получены на материале с митотическим индексом от 40% (Allium виды) до 5% (R. wichurana). Количество препаратов, которые могут быть получены, сильно варьирует и зависит, как от митотического индекса, так и от размера меристемы. 3.1.2.3 Условия сушки препарата после действия пара Другим важным фактором, влияющим на качество разброса хромосом, является скорость высыхания поверхности стекла после действия пара. В работе были использованы два варианта высушивания стекла после действия пара: 1) высыхания при естественных условиях (относительная влажность 45-50%, 2325°С); 2) Высушивание стекла под действием потока воздуха. В первом варианте, время полного высыхания препарата занимало 20-30 секунд. При этом часто наблюдался сильный разброс хромосом ведущий к смешиванию хромосом из разных клеток или отсутствию полного набора хромосом в клетке 77 Микроскопические наблюдения показали, это обусловлено, тем, что после разброса под действиям пара хромосомы еще не иммобилизированы к стеклу, при этом потоки жидкости (уксусная кислота + спирт + вода), образующейся после действия пара, легко их смывают. Во втором варианте время высыхания препарата занимало несколько секунд. Переразбросы хромосом были детектированы редко, а большинство метафаз имели удовлетворительную степень разброса хромосом. 3.1.2.4 Приготовление препаратов хромосом Сам процесс приготовления препаратов методом SteamDrop сводится к нескольким шагам: 1) нанесение клеточной суспензии на стекло; 2) добавление смеси с подходящим соотношением спирта и кислоты в зависимости от степени мацерации ткани меристемы ферментной смесью; 3) Действие пара после образования мениска; 4) быстрая сушка препарата. Как было сказано выше, слишком концентрированный фиксатор обуславливает преждевременный гидролиз цитоплазмы, который приводит к отсутствию разброса одновременно с частым переразбросом хромосом, вызванным вымыванием хромосом потоками смеси фиксатора и воды. Наблюдения за динамикой разброса хромосом свидетельствуют, что цитоплазма – важный элемент движения хромосом. Цитоплазма служит субстанцией, в которой хромосомы двигаются во время разброса. Поэтому отсутствие цитоплазмы не затрудняет движение хромосом и как результат приводит к плохому разбросу хромосом. На видах с мелкими хромосомами (Cannabis sativa, Humulus japonicus, Humulus lupulus, Linum. usitatissimum, Populus nigra, Brassica oleracea, Ricinus communis) число метафазных пластинок с хорошим разбросом хромосом (менее 2 перекрытий хромосом) составило 85-97%. Исключение составили только хромосомы R.wichurana, для которых процент хорошо разбросанных метафазных пластин был крайне низок, и, кроме этого, наблюдалось большое количество цитоплазмы. Этот показатель для видов с крупными хромосомами (Allium cepa, 78 Allium fistulosum, Triticale, Triticum aestivum) составил всего 15-20%. Для видов с крупными хромосомами и для R.wichurana был протестирован подход с использованием второй капли фиксатора после действия пара: фиксатор 1– пар – Фиксатор 2 – пар – высушивание. Сравнение действия на разброс различных фиксаторов и техники двойной «паровой капли» выявили существенное повышение количества метафазных хромосом с хорошим разбросом (Таблица 6) в случае двойной паровой капли. Таблица 6. Количество метафаз A.cepa с хорошим разбросом (0-3 перекрытий хромосом), полученных после действия разных по составу фиксаторов и применения двойной «паровой капли». Состав фиксатора 3:1 (уксусная кислота:96% этанол 2:1 1:1 Общее число 462 проверенных метафаз Среднее число метафаз 8.7±6.7% ± стандартное отклонение, % 320 370 3:1 – (двойная «паровая капля») 418 1:1 14.5±5.6% 12.9±3.4% 31±4.3% Фиксатор I служит преимущественно для разброса хромосом и оставляет много цитоплазмы, а Фиксатор 2 - для разброса и, главное, для гидролиза остатков цитоплазмы и органического слоя, покрывающего весь препарат после действия Фиксатора I. Фиксатор I должен содержать меньшую концентрацию уксусной кислоты, чем Фиксатора 2, чтобы действие последнего не вызвало переразброса хромосом. Подбор пары фиксатора следует делать исходя из степени гидролизованности клеточной стенки ферментами. В роли фиксатора II может выступать и 100% уксусная кислота. 79 3.1.2.5 Апробация метода “SteamDrop” ―SteamDrop‖ протокол был апробирован на других видах с целью оценки универсальности его использования в в современных цитогенетических исследованиях. Метод был применен молекулярно- на 28 видах видах растений как с мелкими, так и с крупными хромосомами, включая Allium cepa (2n = 2x =16), A. fistulosum (2n = 2x =16), A. schoenoprasum (2n = 2x =16), A. altaicum (2n = 2x=16), Linum usitatissimum (2n = 2x =16), Triticum aestivum (2n = 6x =42), Cannabis sativa (2n = 2x =20), Allium roylei (2n = 2x =16), A. wakegi (2n = 2x =16), Humulus japonicus (2n = 2x =17, для мужских особей or 2n = 2x =16 для женских особей), H. lupulus (2n = 2x =20), Rosa wichurana (2n = 2x =14), Populus nigra (2n = 2x =38), Brassica oleracea (2n = 4x=36), Ricinus communis (2n = 2x =20), Anthurium andreanum (2n = 2x =30), Monstera deliciosa (2n = 4x =60), Philodendron scandens (2n = 2x =32), Spathiphyllum wallisii (2n = 2x =30), Syngonium auritum (2n = 2x =24), Zantedeschia elliotiana (2n = 2x =32), Aloe vera (2n = 2x =14), Hippophae rhamnoides (2n = 2x =24), Festuca arundinacea (2n = 6x =42) and Lolium perenne (2n = 2x =14), Thinopyrum ponticum (2n = 10x =70), Th. elongatum (2n = 2x =14). Мелкие хромосомы легко поддавались разбросу и 85-97% полученных метафаз имели хромосомы без перекрытий. В то же время крупные хромосомы луков так же поддавались разбросу, но число метафаз с хорошим разбросом было значительно меньше (30-40%) (Рисунок 16). 80 Рисунок 16. Митотические метафазные хромосомы восьми видов растений и пахитенные хромосомы A.cepa, приготовленные методом ―SteamDrop‖. 3.1.2.6 Адаптация метода “SteamDrop” для приготовления препаратов пахитенных хромосом A.cepa и R.wichurana Несмотря на ценность препаратов пахитенных хромосом для физического картирования, для многих видов растений получения таких препаратов затруднительно (de Jong et al. 1999; Kulikova et al. 2001; De Capdeville G. et al. 2009; Iacia and Pinto-Maglio, 2013). Пахитенные хромосомы видов Rosaceae и Alliaceae никогда не были использованы в молекулярно-цитогенетических исследованиях. Метод ―SteamDrop‖ был нами успешно использован для приготовления пахитенных хромосом A.cepa (Рисунок 16). Трудности в приготовлении препаратов пахитенных хромосом связаны с жесткой клеточной стенкой материнских клеток пыльцы (Zhong et al. 1996). Для приготовления препаратов пахитенных хромосом A.cepa были использованы фиксаторы с 81 высоким содержанием кислоты (1:1 и 1:2, спирт:кислота). В результате были получены качественные препараты пахитенных хромосом с оптимальным разбросом и минимальным количеством цитоплазмы (Рисунок 16). Информации о косвенных признаках R.wichurana (например, размер бутона или гипантия), которые бы могли быть использованы для приблизительной оценки стадии мейоза в материнских клетках пыльцы, отсутствует. В связи с этим, были отобраны бутоны R.wichurana с длиной гипантия от 2 до 5 мм для определения стадии мейоза (Рисунок 17). Рисунок 17. Бутоны R.wichurana, используемые в анализе мейоза Цитологический анализ показал, что бутоны с длиной гипантия около 3 мм содержат пыльниками с материнскими клетками пыльцы в стадиях пахитены – тетрады (Рисунок 18). Рисунок 18. Стадии мейоза, обнаруженные в пыльниках из одного бутона R.wichurana: A. Пахитена, В. Метафаза I, C. Тетрады 82 В одном бутоне R.wichurana содержатся несколько десятков пыльников и стадии мейоза между отдельными пыльниками варьируют. Бутоны с данным размером гипантия были в дальнейшем использованы для приготовления препаратов. Для получения качетсвенных препаратов, были тестированы следующие условия: 1) Различное время обработки ферментами (180, 270 и 390 минут); 2) Концентрация цитогеликазы в 0.6% смеси ферментов (от 0.2% до 1%). Сравнение качества препаратов (разброс пахитенных хромосом и количество цитоплазмы) позволило установить, что оптимальное время инкубации в ферментах составило 270 мин, а увеличение времени инкубации до 390 мин не приводило к значительному повышению качетва препарата. Увеличение концентрации цитогеликазы с 0,2% до 1% не приводило к видимым изменениям качества препарата, поэтому в дальнейшем была использована смесь ферментов, содержащая 0.6% целлулазы, 0.6% пектиназы и 0.2% цитогеликазы. Препараты пахитенных хромосом R.wichurana, полученные методом «паровой кали», имели органический слой, покрывающий весь препарат. Подобное явление было ранее описано для препаратов митотических метафазных хромосом ячменя, приготовленных методом капли и используемых для сканирующей электронно микроскопии (Martin et al. 1994). Кроме этого FISH с такими препаратами и 45S рДНК приводило к слабому сигналу. Мы предположили, что органический слой затрудняет прохождение пробы к целевому фрагменту ДНК хромосомы. Для удаления органического слоя были использованы два подхода: 1) нанесение капли 45% уксусной кислоты после действия пара и накрывание препарата покровным стеклом (Martin et al. 1994); 2) нанесение капли 45% или 60% уксусной кислоты и ее перемещение по препарату с помошью препаровальной иглы в течении 15-30 секунд на нагревательном столике (42°С). Подход, описанный Martin с соавторами (1994), приводил к значительным потерям клеток и, кроме этого, создавал трудности при сколе покровного стекла. Использование второго подхода позволило получить препараты пахитенных хромосом без детектируемого 83 органического слоя, с подходящим разбросом и без цитоплазмы (Рисунок 18А, Kirov et al. 2015a). 3.1.2.7 Использование препаратов, приготовленных методом “SteamDrop”, для FISH и Tyramide-FISH Современные молекулярно-цитогенетические методы требуют препараты высокого качества с минимальным уровнем цитоплазмы, хорошим разбросом и структурой хромосом. Для ответа на вопрос о возможности использования препаратов, приготовленных методом ―SteamDrop‖, в современных молекулярноцитогенетических исследованиях, данные препараты были задействованы в протоколах FISH и Tyramide-FISH (Рисунок 19). Препараты были практически без цитоплазмы, поэтому предобработка пепсином была исключена из протокола. К тому же, применение пепсина на препаратах, приготовленных методом ―SteamDrop‖, часто обуславливало разрушение хромосом и отсутствие сигнала даже от 5S на хромосомах A.cepa. Для FISH были выбраны пробы повторяющейся ДНК (5S, 45S, (AAC)5 микросателлит, HJSR KpnI (Alexandrov et al. 2012) тандемный повтор) и уникальных последовательностей (генов аллиназ и LFS). Для проверки были использованы препараты видов с крупными (A.cepa, A.fistulosum, T.aestivum) и мелкими (H.japonicus, R.wichurana) хромосомами. Результаты FISH с 5S рДНК на хромосомах A.fistulosum (Рисунок 19В) ,45S рДНК на хромосомах R.wichurana (Рисунок 19Н), (AAC)5 микросателлитом на хромосомах T.aestivum (Рисунок 19D) и HJSR KpnI (Alexandrov et al. 2012) на хромосомах H. japonicus (Рисунок 19A) совпадали с ранее опубликованными данными (Ricroch et al. 1992; Akasaka et al. 2002; Alexandrov et al. 2012; Cuadrado et al. 2000). 84 Рисунок 19. Результат использования препаратов, приготовленных методом ―SteamDrop‖, для FISH и Tyramide-FISH. (A) FISH с Dig меченным повтором HJSR KpnI (Alexandrov et al. 2012) на хромосомах H. japonicus; FISH с 5S рРНК на метафазных хромосомах A.fistulosum (В) и пахитенных хромосомах A. cepa (С); (D) FISH на хромосомах T.aestivum c (AAC)5 микросателлитом; FISH с 45S рРНК на метафазных (H) и пахитенных хромосомах R.wichurana (G); Tyramide-FISH c Dig меченным фрагментом гена LFS (E) (550 п.н.) и аллиназы луковицы (F) (1100 п.н.) на хромосомах A.cepa. Линейка – 10 мкм. 85 Для того, чтобы выяснить подходят ли приготовленные методом ―SteamDrop‖ препараты для детекции уникальных коротких последовательностей ДНК, было проведено физическое картирование с помощью Tyramide-FISH фрагментов генов аллиназы (1100 п.н. и LFS (550 п.н.)). Ген аллиназы луковицы был локализован в дистальном регионе длинного плеча хромосомы 4 (Рисунок 16F), а ген LFS в проксимальном регионе хромосомы 5 (Рисунок 19E). Такие же результаты были нами ранее получены на препаратах, приготовленных методом распластывания (Khrustaleva et al. 2012). Особый интерес был связан с использованием пахитенных хромосом A.cepa и R.wichurana, приготовленных методом ―SteamDrop‖ в FISH экспериментах, т.к. до этого пахитеные хромосомы видов из рода Allium и из семейства Rosaceae никогда не использовались для FISH. FISH на приготовленных препаратах позволили успешно визуализировать локусы генов 5S и 45S рРНК на хромосомах A.cepa и R.wichurana, соответственно (Рисунок 19С и 19G). Визуализация сайтов 5S рРНК на пахитенных хромосомах ―SteamDrop‖ A.cepa, приготовленных методом позволила впервые приблизительно оценить разницу в разрешающей способности FISH между пахитенными хромосомами A.cepa и митотическими метафазными хромосомами. Расстояние между сайтами 5S рРНК на митотической метафазной хромосоме составляет около 4.4 мкм, в то время как на пахитенной хромосоме – около 20.4 мкм. Таким образом, разрешающая способность FISH на пахитенной хромосоме в 5 раз выше, чем на митотической метафазной хромосоме. Хотя этот показатель даѐт приблизительную оценку и может меняться в зависимости от стадии пахитены и локализации ДНК пробы (гетерохроматин или эухроматин) (Cheng et al. 2001). Итак, нами разработан новый метод приготовления препаратов митотических и мейотических хромосом растений ―SteamDrop‖. Это удалось сделать, благодаря детальному изучению динамики разброса хромосом во время приготовления препаратов и факторов, влияющих на разброс хромосом, а также благодаря накопленным на сегодня данным по использованию метода капли на 86 хромосомах человека, животных и растений. Главными достоинствами метода являются: 1) Универса льность; 2) Простота адаптации для разных видов; 3) Возможность сохранять клеточную суспензию долгое время; 4) Возможность контролировать разброс хромосом; 5) Возможность приготовить несколько препаратов из единичной меристемы; 6) Совместимость препаратов с молекулярно-цитогенетическими методами (FISH, Tyramide-FISH). К недостаткам метода можно отнести его высокую зависимость от влажности и температуры воздуха. Стоит отметить, что«SteamDrop» метод был использован в молекулярноцитогенетических исследованиях генома R.wichurana (Kirov et al., 2014a; Kirov et al. 2015a), Cannabis sativa (Divashuk et al. 2014; Alexandrov et al. 2015; Razumova et al. 2015) и A.cepa (Kiseleva et al. 2014, Romanov et al. 2015), Nicotiana (Laskowska et al. 2015), Pennisetum squamulatum (Sapkota, 2014). Хочется верить, что разработанный метод позволит решить некоторые проблемы, связанные с приготовлением препаратов хромосом растений, и существенно облегчит проведение физического картирования различных последовательностей ДНК на хромосомах растений. 3.2 Оптимизация Tyramide-FISH 3.2.1 Оптимизация системы детекции 3.2.1.1 Оптимизация прямой и непрямой систем детекции для физического картирования генов на мелких хромосомах R.wichurana Для оптимизации системы детекции Tyramide-FISH для физического картирования генов\маркеров на хромосомах розы R.wichurana был клонирован 87 фрагмент гена OOMT (о-орцинол метил трансфераза), размером 1.1 тыс.п.н. Затем полученный фрагмент был помечен биотином с помощью Ник-трансляции (Roche). Для контроля нами была использована меченая биотином плазмида, несущая 45S рРНК (pTA71) пшеницы (Gerlach and Bedbrook, 1979). TyramideFISH с полученной пробой 45S и OOMT была проведѐна на хромосомах, приготовленных методом ―SteamDrop‖. Для проверки прямой системы детекции была проведена Tyramide-FISH с меченной биотином плазмидой 45S. Прямая детекция сайтов гибридизации проводилась с помощью пероксидазы хрена , конъюгированной со стрептавидином, с последующим инкубированием препаратов с раствором тирамид, конъюгированных с флуорохромом FITC (тирамид-FITC) или Cу3 (тирамид-Cy3). Результаты эксперимента показали чѐткие и яркие сигналы в конце одной пары хромосом, несущей спутник (Рисунок 20). Рисунок 20. Tyramide-FISH с прямой системой детекции тирамид-FITC (справа) и тирамид-Cy3 (слева) сайтов локализации генов 45S рРНК на хромосомах R.wichurana. Линейка – 10 мкм. Обе прямые системы детекции (с тирамид-FITC и тирамид-Cy3) давали схожие результаты в количестве сигналов и средней интенсивности. 88 Проверив прямую ситему детекции с использованием в качестве контроля 45S рРНК гены, которые могут занимать миллионы пар нуклеотидов на хромосоме и их детекция может быть проведена и обычной FISH (Jiang and Gill, 1994) мы провели эксперименты по визуализации коротких последовательностей ДНК. На хромосомах розы нами был проведен Tyramide-FISH эксперимент с прямой детекцией, где в качестве пробы был выбран фрагмент гена OOMT размером 1.1 тыс.п.н.. Tyramide-FISH выявила высокий уровень фоновых сигналов, которые отличаются от истинных сигналов исходящих с ДНК-мишени специфическим расположением в цитоплазме над хромосомами и неповторяемостью их локализации между метафазами (Рисунок 21). Данные фоновые сигналы значительно затрудняли выявление реальных сигналов, исходящих с сайтов гибридизации OOMT пробы. Поэтому физическая локализация гена OOMT с помощью прямого метода детекции не была определена. Описанные выше результаты применения прямой системы детекции для физической локализации гена OOMT не зависили от используемых тирамид коньюгатов (тирамид-FITC или тирамид-Cy3). Рисунок 21. Tyramide-FISH с фрагментом гена OOMT на хромосомах R.wichuraiana с прямой детекцией тирамид-FITC. 89 Изменения концентрации тирамид (1:100, 1:50) и времени инкубации (5, 7, 10 мин.) при использовании прямой системы детекции незначительно влияли на интенсивность фоновых сигналов. В целях сравнения различных систем детекции тирамидами, были проведены эксперименты по оптимизации непрямой системы детекции. Система включала в себя следующие шаги: 1) Инкубация со Стрептавидином-HRP; 2) Инкубация с тирамид-биотином; 3) Инкубация со Стрептавидином-Cy3. Оптимизация затронула второй и третий шаги непрямой детекции. Нами было выявлено, что ключевым фактором, влияющим на интенсивность сигнала и фона является концентрация тирамид-биотин коньюгатов. При сравнении трѐх разбавлений (1:25, 1:50 и 1:100) тирамид-биотин в амплифицирующем буфере (PerkinElmer) и различных концентраций стрептавидин-Cy3 (1:100 и 1:300), с помощью визуальной оценки, было найдено, что разбавление 1:25 тирамидбиотин коьюгатов и 1:100 антител стрептавидин-Cy3 давали наиболее интенсивный и часто встречающийся сигнал с низким уровнем фона. Необходимо отметить, что выявленные нами оптимальные концентрации отличаются от рекомендованных фирмой производителем и ранее используемых в TyramideFISH картирование на хромосомах пшеницы (Perez et al. 2009), овса (Sanz et al. 2012) и ячменя (Stephens et al. 2004). Использование оптимальных концентраций позволило нам впервые провести физическое картирование гена с помощью Tyramide-FISH на мелких хромосомах растений (Kirov et al. 2014a, 2015) (Рисунок 22). 90 Рисунок 22. Tyramide-FISH с оптимизированной непрямой детекцией с геном OOMT на хромосомах R.wichuraiana. Отличительной особенностью хромосом розы является их маленький размер. Работы по физическому картированию коротких последовательностей (генов\маркеров) с помощью метода Tyramide-FISH были проведены лишь на растения с крупными хромосомами (Khrustaleva and Kik, 2001; Stephens et al. 2004; Perez et al. 2009; Sanz et al. 2012; Khrustaleva et al. 2012; Романов и др., 2015, Romanov et al. 2015). Проведѐнные исследования по поиску оптимальных концентраций тирамид коньюгатов и антител позволят в дальнейшем провести физическое картирование и других важных генов на хромосомах розы и, возможно, на хромосомах других видов с мелкими хромосомами. 3.2.1.2 Оптимизация прямой и непрямой систем детекции для физического картирования генов на крупных хромосомах Allium cepa В качестве пробы для использования в оптимизации Tyramide-FISH был использован фрагмент гена аллиназы луковицы (L48614), для которого с помощью прямой системы детекции ранее была показана локализация в дистальной части хромосомы 4 (Khrustaleva et al. 2012). Были подобраны праймеры, которые позволяли амплифицировать фрагмент гена аллиназы луковицы размером 1100 п.н. Данный ПЦР продукт был клонирован в pPCR- 91 TOPO вектор. Из колоний, содержащих плазмиды с ожидаемым размером вставки, была выделена плазмидная ДНК. После секвенирования были идентифицированы клоны, несущие целевой фрагмент гена аллиназы луковицы (Allbe). Плазмидная ДНК клона Allbe была помечена биотином с помощью Никтрансляции и использована в Tyramide-FISH экспериментах. Для достижения максимального значения отношения интенсивности флуоресценции сигнала к интенсивности флуоресценции фона были применены различные концентрации пробы (50 и 100 нг на стекло) и время инкубации препаратов в растворе тирамидов (6, 8 и 10 мин). Лучшие результаты были получены при применении 50 нг пробы на стекло и времени инкубации 8 мин (Рисунок 23). Для детекции аллиназы нами была впервые для хромосом лука применена непрямая система детекции сигнала с использованием биотинилированных тирамидов с последующей детекцией стрептавидин-Cy3. Tyramide-FISH с различными концентрациями тирамидов и вторичных антител (Стрептавидин-Cy3) позволили выявить оптимальные значения данных показателей, которые составили: разбавление тирамидов - 1:25 и стрептавидинCy3 - 1:100. Увеличение концентрации обоих компонентов позволило получить сигнал от сайта гибридизации Allbe высокой интенсивности (Рисунок 23). 92 Рисунок 23. Интенсивность сигнала от фрагмента гена аллиназы луковицы (1.1. тыс.п.н.) на хромосоме 4 после непрямой детекции. Сравнение прямой и непрямой систем детекции с использованием Allbe фрагмента позволило установить, что интенсивность флуоресценции на порядок выше при непрямой системе детекции (Рисунок 24). Наиболее важный параметр при детекции уникальных коротких последовательностей – это отношение интенсивности сигнала к интенсивности фона. При использовании непрямой детекции этот показатель был в 2-3 раза выше, чем в прямой детекции (Рисунок 24). 93 Рисунок 24. Профили интенсивности сигнала от аллиназы луковицы на хромосомах A.cepa после непрямой и прямой систем детекций. Красная линия показывает интенсивность флуоресценции вдоль дистальной части хромосомы 4 A.cepa. Используемые для измерения снимки были сделаны с одним временем выдержки (exposure time). 94 Такое значительное увеличение чувствительности может быть связано с повышенной концентрацией тирамид-биотин. При уменьшении концентрации тирамид-биотин до 1:50 мы наблюдали резкое снижение интенсивности флуоресценции. С коньюгированных другой стороны, увеличение концентрации тирамид, напрямую с флуорохромом, до 1:25 приводило к резкому увеличению фонового сигнала. Другое объяснение увеличения чувствительности возможно связано со структурой антител стрептавидин-Cy3. Так как стрептавидин – тетрамер, то при его мечении Cy3 большее число флуорохромов может быть коньюгировано с одной молекулой. Поэтому при использовании стрептавидин-Cy3 для детекции депонированных тирамид-биотин молекул, на одну молекулу биотина приходится несколько флуорохромов, что и обуславливает повышенную чувствительность детекции. Непрямая система детекции тирамидами может стать удобным подходом для визуализации с высокой эффективностью коротких последовательностей ДНК как на крупных, так и мелких хромосомах. Как показали результаты исследований, физическое картирование коротких последовательностей на хромосомах R.wichurana может быть проведено только с помощью непрямой детекции. В тоже время, и прямая и непрямая система детекции могут быть использованы для картирования коротких фрагментов ДНК на хромосомах Allium. 3.2.2 Химический синтез коньюгатов тирамид и их применение в TyramideFISH детекции на хромосомах Allium и R.wichurana Цена коммерческих тирамид коньюгатов достаточно высока (1213EUR/250препаратов (PerkinElmer)) и самостоятельный синтез тирамид коньюгатов может значительно удешевить процесс физического картирования генов с помощью Tyramide-FISH. и Были предложены протоколы по синтезу тирамид коньюгатов в условиях лаборатории (Bobrow et al. 1991; Adams, 1992; 95 Hopman et al. 1998; Raap et al. 1995; Chao J. et al. 1996). Синтезированные тирамиды были использованы в ряде работ на клетках млекопитающих (Bobrow et al. 1991; Adams, 1992; Hopman et al. 1998; Pernthaler and Amann, 2004; et al. 1996; Kerstens et al. 1995). В данных работах авторы использовали, в основном, достаточно протяжѐнные уникальные и повторяющиеся последовательности. На сегодня, синтезированные тирамиды ещѐ не были использованы в физическом картировании коротких уникальных последовательностей, включая гены, на хромосомах растений. Мы провели синтез тирамид, коньюгированных с биотином, и использовали их для детекции фрагментов уникальных генов Allbe на хромосомах A.cepa и ООМТ на хромосомах R.wichurana. Синтез проводили согласно протоколу Hopman с соавторами (1998) с использованием коммерческих реагентов, включая n-гидроксисукцинимид биотин и тирамин (см. материалы и методы). A B Рисунок 25. Применение синтезированных тирамид-биотин коньюгатов в Tyramide-FISH с фрагментом Allbe на хромосомах A.cepa (А) и с фрагментом гена ООМТ на хромосомах R.wichurana (B). Результаты показали, что оба гена были успешно визуализированы с помощью синтезированных тирамид-биотин конъюгатов (Рисунок 25). Цена синтезированных тирамид-биотин конъюгатов в два раза ниже коммерческих аналогов. Использование синтезированных тирамид-биотин конъюгатов позволит значительно удешевить процесс физического картирования геном с помощью 96 Tyramide-FISH. Далее при картировании генов розы были использованы только синтезированные нами тирамиды. 3.2.3 Влияние приготовления препаратов хромосом на результаты TyramideFISH Тирамиды после активации пероксидазой соединяются с «электронбогатыми» фенольными молекулами в непосредственной близости от сайта локализации пероксидазы (Bobrow et al. 1991)). Источником таких молекул на препарате могут быть гистоны хромосомы и другие белки. Ранее было показано, что процесс приготовления препаратов митотических метафазных хромосом оказывает существенное влияние на количество гистонов и структуру хромосомы (Dick and Johns, 1968; Sugiyama S. et al. 2004). В частности, авторами было показано, что обработка хромосом 45% уксусной кислотой значительно нарушает структуру хромосомы и уменьшает диаметр хромосомы, хотя и позволяет приготовить чистый препарат. Разрушительное действие на хромосому уксусной кислоты объясняется, прежде всего, существенным экстракцией гистонов из хромосомы (Dick and Johns, 1968). И структура хромосомы и содержание гистонов, имеющих сайты посадки для активизированных ПХ тирамид, играют важную роль в детекции коротких последовательностей ДНК с помощью Tyramide-FISH. Поэтому, мы предположили, что понижение концентрации уксусной кислоты при приготовлении препаратов приведет к снижению экстракции гистонов из хромосомы и позволит улучшить результаты TyramideFISH. Для проверки этой гипотезы мы провели Tyramide-FISH на препаратах хромосом, приготовленных методом ―SteamDrop‖ с использованием низкой (фиксаторы 10:1 (спирт:уксусная кислота) – первая капля и 5:1 – вторая капля) и высокой (фиксаторы 1:1 – первая капля и 100% уксусная кислота – вторая капля) концентраций уксусной кислоты в фиксаторе. Препараты были приготовлены из одной клеточной суспензии. Затем была проведена Tyramide-FISH с фрагментом гена аллиназы корня I (1100 п.н.). Картирование показало, что данный ген локализован в 5 локусах на хромосомах A.cepa: на хромосоме 2 (три локуса), 4 97 (один локус) и 6 (один локус). Для оценки был проведен подсчет частоты встречаемости сигналов (ЧВС = число хромосом с сигналом х 100%/общее число анализированных хромосом 2и 6) на 3 препаратах хромосом для каждого вариант фиксатора (n=80). Для подсчета ЧВС учитывались только сигналы на хромосомах 2 и 6 (спутничная) (Рисунок 26). Сайты гибридизации были детектированы с помощью прямой системы детекции сигнала (тирамид-Cy3). Ch2S Ch6 Ch2Cen t Ch2Tel Хромосома 6 Хромосома 2 Рисунок 26. Положение и обозначения 4 локусов аллиназы корня, учитываемых при подсчетах числа встречаемости сигналов (ЧВС) на хромосомах 2 и 6 A.cepa. Результаты показали, что 1) морфология хромосом, приготовленных с использованием разных концентраций уксусной кислоты, была различна после Tyramide-FISH и в варианте с использованием низкой концентрации уксусной кислоты, хромосомы имели ярко выраженные выпетливания хроматина (Рисунок 27А); и 2) ЧВС на препаратах, приготовленных с низкой и высокой концентрацией уксусной кислоты, существенно отличаются (Рисунок 28). 98 Рисунок 27. Влияние концентрации уксусной кислоты, используемой для приготовления препаратов, на морфологию хромосом после Tyramide-FISH. А – вариант с низкой концентрацией (15%) уксусной кислоты; Б – вариант с высокой концентрацией (100%) уксусной кислоты. Красные сигналы соответствуют местам гибридизации фрагмента гена аллиназы корня I (1100 п.н.) на хромосомах A.cepa. Зеленые сигналы – места гибридизации 45S рДНК. Стрелки показывают выпетливание хроматина. 99 100 90 80 70 60 50 40 30 20 10 0 10:1/5:1 1:1/100% Ch6 Ch2S Ch2LCent Ch2L Tel Рисунок 28. Сравнение показателя частоты встречаемости хромосом с сигналом (ЧВС) после Tyramide-FISH детекции аллиназы корня для хромосомы 2 по 3 локусам (Ch2S, Ch2Cent, Ch2Tel) и для хромосомы 6 по одному локусу (Ch6). Столбцы гистограммы представляют два варианта фиксаторов, используемых для приготовления препаратов: синий столбец – вариант с низкой концентрацией уксусной кислоты (фиксаторы 10:1 (спирт:кислота) – первая капля и 5:1 – вторая капля), красный столбец – вариант с высокой концентрацией уксусной кислоты (фиксаторы 1:1 – первая капля и 100% уксусная кислота – вторая капля). Достоверные различия ЧВС были выявлены по всем 4 локусам (Рисунок 28) аллиназы. Во всех случаях показатель ЧВС был существенно выше при использовании низкой концентрации уксусной кислоты. Данные результаты подтвердились и на хромосомах розы, где применение мягких условий (фиксаторы 3:1 (спирт:кислота) – первая капля и 1:1 – вторая капля) при приготовлении препаратов методом ―SteamDrop‖, также существенно повысило ЧВС (до 51%) после Tyramide-FISH детекции фрагмента гена MLO1 (Рисунок 29). Как и в случае с хромосомами A.cepa, морфология хромосом лучше сохранилась в варианте с высокой концентрацией уксусной кислоты. 100 Рисунок 29. Морфология хромосом и частота встречаемости сигналов от гибридизации фрагмента гена MLO с хромосомами розы, на препаратах, приготовленных с низкой (А) (фиксаторы 3:1 (спирт:кислота) – первая капля и 1:1 – вторая капля) и высокой (Б) (1:1 / 100% уксусная кислота) концентрациями уксусной кислоты. На графике показана частота встречаемости хромосом с сигналом, приготовленных с использованием фиксаторов 3:1/1:1 (левый столбец) и 1:2/100% уксусная ксилота (правый столбец). Данные, полученные на хромосомах двух видов, согласуются. Такие отличия в морфологии и ЧВС на хромосомах, приготовленными с высокой и низкой концентрациями уксусной кислоты, могут объясняться двумя причинами. Вопервых, хромосомы, приготовленные с низкой концентрацией уксусной кислоты, имеют менее поврежденную структуру до FISH и больший диаметр хромосомы (Sugiyama et al. 2004). Тем самым улучшается доступность хроматина для гибридизации, что и проявляется в повышении частоты встречаемости сигналов. Во-вторых, уксусная кислота способна экстрагировать гистоны из хромосомы, и, таким образом, снижать число «электрон-богатых» аминокислот (тирозин, триптофан, фенилаланин), необходимых для депонирования тирамид после их 101 активации пероксидазой, и, как следствие, снижать ЧВС. Стоит отметить, что авторами ранее предложенных методов приготовления препаратов хромосом растений, как правило, использовалась нагретая 45% или 60% уксусная кислота, а так же ледяная уксусная кислота (Pijnacker and Ferwerda, 1984; Kato, 1997). В отличие от этих методов, ―SteamDrop‖ позволяет приготовить препараты хромосом с низкой концентрацией уксусной кислоты. Обнаруженные закономерности влияния условий приготовления препаратов на результаты Tyramide-FISH позволили существенно увеличить эффективность физического картирования генов и фрагментов генов (от 1100 п.н.) на хромосомах розы и лука. Впервые показано прямое влияние способа приготовления препаратов на успех физического картирования с помощью Tyramide-FISH. Разработанный метод ―SteamDrop‖ позволил манипулировать концентрациями уксусной кислоты в фиксаторе и, таким образом, сделал картирование с помощью Tyramide-FISH эффективнее. 3.3 Поиск цитогенетичеких маркеров Физическое картирование хромосом с помощью FISH и Tyramide-FISH методов существенно позволяющих облегчается распознавать наличием индивидуальные цитогенетических хромосомы или маркеров, биваленты (пахитенные хромосомы). Цитогенетические маркеры необходимы для сортинга индивидуальных хромосом с помощью FISHIS (Giorgi et al. 2013), что позволяет сиквенирование отдельных хромосом и тем самы намного ускоряет процесс сборки геномов. Набор цитогенетических маркеров для видов Allium и Rosa wichurana ограничивался 5S и 45S, позволяющих идентифицировать лишь две хромосомы кариотипа. Исключение составляет геном A. fistulosum, для хромосом которого был ранее обнаружен новый видоспецифичный повтор, AfiT32 (Киселева, 2014). AfiT32 был использован, как цитогенетический маркер, для идентификации хромосом 5 и 6 у A.fistulosum. Целью данной части работы был поиск цитогенетических маркеров для хромосом Allium fistulosum и Rosa 102 wichurana для создания платформы для эффективного физического картирования с помощью Tyramide-FISH. 3.3.1 Поиск цитогенетических маркеров для хромосом Allium 3.3.1.1 Анализ данных секвенирования следующего поколения A.fistulosum Поиск ДНК последовательностей-кандидатов в цитогенетические маркеры для хромосом A.fistulosum был проведен с помощью сервера RepeatExplorer (Novak et al. 2013). Для анализа были использованы короткие геномные риды A.fistulosum, доступные в базе данных NСBI (SRX268217). Результаты анализа ридов с помощью RepeatExplorer позволили выявить один кластер ридов, Cl58, имеющий глобулярную форму, соответствующую тандемным повторам (Novak et al. 2013). С помощью программы Tandem Repeat Finder (Benson, 1999) в контигах, построенных из ридов данного кластера, был обнаружен тандемно- организованный повтор с длиной мономера 65 п.н.. Данный повтор был назван HAT58. BLASTN поиск в базе данных NСBI не выявил каких либо сиквенсов, схожих с HAT58. Согласно данным RepeatExplorer данный повтор занимает 0,09% генома A.fistulosum, что составляет около 160 000 копий/1C. ПЦР с подобранными праймерами на HAT58 и геномной ДНК A. fistulosum подтвердила тандемную организацию HAT58 в геноме A. fistulosum (Рисунок 30). ПЦР с подобранными праймерами и геномной ДНК A.cepa не дала положительных результатов. 103 Рисунок 30. Результат ПЦР с праймерами на повтор HAT58 и геномной ДНК A.fistulosum (M - 100 bp plus (Fermentas) маркер размеров) и форма кластера Cl58, соответствующего HAT58. 3.3.1.2 FISH картирование повторов на хромосомах A.cepa и A.fistulosum FISH с меченым ПЦР продуктом HAT58 и хромосомами A.fistulosum выявил четкие сигналы в хромосомах 6, 7 и 8 (рисунок 31А). Хромосома 6 имеет один локус в прицентромерной области длинного плеча. Хромосома 7 содержит один полиморфный локус в проксимальной части. Были идентифицированы растения с единичным локусом на одной из хромосом 7 (генотип Aa), с локусами на обеих гомологичных хромосомах (генотип АА) и растения, не содержащие локуса HAT58 на хромосоме 7 (генотип аа). Хромосома 8 имеет несколько сайтов локализации в проксимальной части длинного плеча и в интерстициальной части короткого плеча. Результаты FISH с повтором HAT58 свидетельствуют, что данный повтор может быть использован в качестве цитогенетического маркера для идентификации трех из 8 хромосом A.fistulosum. FISH с меченым ПЦР продуктом HAT58 и хромосомами A.cepa не выявил сигналов на хромосомах 104 данного вида. Для определения локализации локусов HAT58 относительно других цитогенетических маркеров (AfiT32, 5S, 45S) была проведена двуцветная FISH (Рисунок 31Б, В). Г Рисунок 31. FISH на хромосомах A.fistulosum: (А) с биотин меченым повтором HAT58; (Б) с биотин меченым повтором HAT58 (красный) и дигоксигенини мечеными 5S и 45S (зеленый); (В) с биотин меченым повтором CAT36 (красный) и дигоксигенини меченым HAT58 (зеленый); (Г) ND-FISH c флуорохром меченым HAT58 (TAMRA, красный). Линейка - 10 мкм. На основе данных двуцветной FISH была построена идиограмма хромосом A.fistulosum с указанным положением всех цитогенетических маркеров (Рисунок 32), включая картированные ранее AfiT32 (Киселева, 2014) и 45, 5S рДНК. 105 Рисунок 32. Локализация цитогенетических маркеров на хромосомах A.fistulosum. Звездочкой отмечен полиморфный локус HAT58. Для обеспечения быстрой идентификации хромосом A.fistulosum с помощью цитогенетических маркеров были разработаны олигонуклеотиды для HAT58 и 5S рДНК, меченые флуоресценция), TAMRA (красная соответственно. В флуоресценция) этом случае и для (зеленая FAM детекции сайтов гибридизации олигонуклеотидов HAT58 и 5S с хромосомами A.fistulosum антитела не требуются. Для уменьшения времени идентификации хромосом была проведена безденатурационная FISH (ND-FISH, Cuadrado and Jouve (2010)). NDFISH была успешно проведена с флуорохром мечеными олигонуклеотидами HAT58 на хромосомах A.fistulosum. Это позволило провести детекцию сайтов гибридизации за 3 часа. Результаты данного эксперимента свидетельствуют, что разработанный флуорохром, меченый олигонуклеотид повтора HAT58, может стать ценным инструментом в анализе гибридов с A.fisulosum и FISHопосредованном хромосомном сортинге хромосом 6, 7 и 8. Сортинг индивидуальных хромосом с помощью FISHIS (Giorgi D. et al. 2013) с флуорохром меченым олигонуклеотидом повтора HAT58 может существенно ускорить проект секвенирования генома лука батуна. В целом, имеющиеся на сегодня цитогенетические маркеры, включая HAT58, позволяют идентифицировать 4 (хромосомы 5, 6, 7 и 8) из 8 хромосом A.fistulosum. Кроме этого хромосомы 1 и 2 могут быть опознаны и отличимы благодаря их размеру. 106 Возможность идентификации хромосом A.fistulosum с помощью этих маркеров будет особенно незаменима при физическом картировании генов с помощью Tyramide-FISH на пахитенной хромосоме, предоставляющей значительно большее разрешение (de Jong et al. 1999). 3.3.2 Поиск цитогенетичесих маркеров для хромосом Rosa wichurana 3.3.2.1 FISH с 45S, 5S и теломерным повтором Arabidopsis типа В качестве ДНК проб-кандидатов в цитогенетические маркеры для хромосом R.wichurana были использованы консервативные последовательности геномов растений, а именно 45S, 5S и теломерный повтор Arabidopsis типа (GGGATTT)n. 45S и 5S – часто используемые цитогенетические маркеры для идентификации хромосом растений, включая виды Rosa (Ma et al., 1997; Fernandez-Romero et al., 2001; Akasaka et al., 2002, 2003; Lim et al. 2005; Jian et al. 2012; Kirov et al. 2014a, 2015). Теломерные повторы занимают окончания хромосом и представлены там большим числом тандемно-организованных копий (Fuchs et al., 1995; He et al., 2013). Однако, теломерные (теломер-подобные) повторы могут быть локализованы в субтеломерной, проксимальной или интерстициальной частях хромосом растений (Fuchs et al. 1995; Uchida et al. 2002; Tek and Jiang, 2004; Mlinarec et al. 2009; Mandakova et al. 2010; Gong et al., 2012; He et al., 2013; Sousa et al., 2014, 2015). В этом случае такие теломерные повторы называют интерстицальные теломерные повторы (ИТП) и могут быть детектированы с помощью FISH c пробой на теломерный повтор. Хромосомспецифичная и уникальная локализация ИТП, делают их превосходными цитогенетическими маркерами. Нами была проведена совместная локализация 45S и теломерного повтора Arabidopsis типа (GGGATTT)n на хромосомах R.wichurana с помощью FISH. 45S рДНК, как и ожидалось, локализована в спутничной части хромосомы 7 (Рисунок 33С). 107 Рисунок 33. FISH на хромосомах R.wichurana c теломерным повтором Arabidopsis типа (GGGATTT)n и 5S, 45S рДНК. A. FISH c теломерным повтором Arabidopsis типа при мягких условиях гибридизации (25ºС). B. Та жа метафаза после FISH с 5S рДНК. С. FISH c теломерным повтором Arabidopsis типа (красный) и 45S рДНК (зеленый). В то же время, теломерный повтор Arabidopsis типа (GGGATTT)n кроме сайтов локализации в теломерных частях хромосомы, локализовался в дополнительных сайтах (ИТП) в центромерном регионе одной пары хромосом (Рисунок 33С). Так как часто, ИТП имеют лишь частичное сходство с теломерным повтором, была проведена FISH c более мягкими условиями гибридизации. Для этого температура гибридизации была снижена с 37ºС до 25ºС. Данный подход позволил выявить дополнительные сайты локализации ИТП в прицентромерных областях трѐх хромосом (Рисунок 33А). Результаты показывают, что FISH с теломерным повтором Arabidopsis типа может быть более информативен для маркирования хромосом растений, если используется температура гибридизации 25ºС, вместо обычно используемой для этих целей 37 ºС (Fuchs et al. 1995; Tek and Jiang, 2004; Sousa et al., 2014, 2015). Для ответа на вопрос, колокализуются ли сайты ИТП и 5S рДНК, после гибридизации повтора Arabidopsis типа на тех же препаратах была проведена FISH с 5S рДНК. Результаты показали, что один локус ИТП и локус 5S рДНК локализованы на одной хромосоме 7. В то же время, второй локус 5S рДНК не колокализуется ни с одним из локусов ИТП. Проведение кариотипирования позволили установить принадлежность сайтов ИТП и 5S рДНК к конкретным хромосомам. Так, локусы ИТП локализованы в центромерных областях хромосом 1, 2, 5 и 7, а 5S рДНК в проксимальных частях длинного плеча хромосом 4 и 7 (Рисунок 34). 108 . Рисунок 34. Локализация 5S, 45S и теломерного повтора Arabidopsis типа на хромосомах R.wichurana. 1-сайты, которые видимы только при гибридизации при 25°С. Для идентификации всех хромосом была проведена двуцветная FISH c флуохором-меченым (TAMRA) теломерным повтором и дигоксигенин меченой 45S рДНК (Рисунок 35). 5S рДНК не была нами использована для данного эксперимента ввиду сложности детекции на хромосомах R.wichurana. Для возможности идентификации проводилась при 25ºС. большего числа хромосом гибридизация 109 Рисунок 35. FISH идентификация хромосом R.wichurana с помощью флуохороммеченого (TAMRA) теломерного повтора и дигоксигенин меченой 45S рДНК. Линейка – 5 мкм Результаты свидетельствуют, что хромосомы 1, 2, 5 и 7 R.wichurana могут быть идентифицироаны на основе FISH сигналов. Из них, хромосомы 1 и 2 могут быть различимы с помощью измерения центромерного индекса, который составляет 46.00±1.2% и 40.30±1.3%, соответственно (Kirov et al. 2014a). Хромосома 5 имеет наиболее яркий сигнал флуоресценции сайта ИТП. Среди остальных хромосом, немаркированных локусами ИТП, хромосома 4 может быть легко идентифицировна, т.к. это единственная субметацентрическая хромосома в кариотипе R.wichurana (ЦИ= 36.9±0.7, Kirov et al. 2014a). Остальные две хромосомы (3 и 6) могут быть отличимы друг от друга по центромерному индексу, который равен 44.3±1 и 41.4 ±0.7, соответственно. Таким образом, FISH c TAMRA-меченым теломерным повтором и дигоксигенин меченой 45S рДНК в сочетании с измерениями центромерного индекса позволяют идентифицировать все 7 хромосом R.wichurana, существенно облегчая дальнейшее физическое (цитогенетическое) картирование. 110 3.3.2.2 FISH идентификация хромосом 5 и 7 R.wichurana в пахитене Так как особенную ценность выявленные цитогенетические маркеры представляют для идентификации пахитенных хромосом R.wichurana, нами была проведена двуцветная FISH с использованием TAMRA-меченного теломерного повтора и дигоксигенин меченой 45S рДНК. Картина FISH по наличию четких сайтов гибридизации от теломерного повтора и 45S рДНК позволяет легко отличить пахитенные хромосомы 5 и 7 (Рисунок 36). Рисунок 36. Маркирование пахитенных хромосом 5 и 7 с помощью FISH с TAMRA-меченым теломерным повтором (красный) и дигоксигенин меченой 45S рДНК (зеленый). Центромерная область хромосомы 5 выделена и увеличена (A‘ и B‘). Кроме этого, стоить отметить, что данный эксперимент позволил установить, что сайт ИТП хромосомы 5 локализуется в центромере, а не в прицентромерном регионе данной хромосомы. Положение центромеры было 111 установлено, благодаря еѐ слабой DAPI окраске относительно других частей хромосомы. В заключение, нами были разработаны системы маркирования хромосом для R.wichurana и A.fistulosum, которые позволяют легко идентифицировать большинство (A.fistulosum) или все (R.wichurana) митотические метафазные хромосомы. 3.4 Физическое картирование генов с помощью оптимизированных протоколов и построение интегрированных генетических и физических карт 3.4.1 Allium 3.4.1.2 Физическое картирование генов на хромосомах близкородственных видов Allium 3.4.1.2.1 Ген аллиназы луковицы Благодаря специфическому вкусу и запаху, представители рода Allium играют важную роль в питании человека. На сегодня известно, что весь спектр вкусовых характеристик видов Allium определяется работой двух ферментов, аллиназы (alliin alkyl-sulfonate-lyase, alliin lyase, alkyl Cys sulfoxide lyase, Cys sulfoxide lyase, and C-S lyase) и синтазы фактора слезотечения (LFS) (Lancaster and Boland, 1990; Imai S. et al. 2002). В связи с важностью этих ферментов, их гены (ген аллиназы луковицы, ген аллиназы корня I и II, ген LFS) были выбраны для физического картирования с помощью Tyramide-FISH. Tyramide-FISH был проведен на хромосомах A. fistulosum, A.altaicum, A.vavilovii и A.schoenoprasum (Рисунок 37). Для Tyramide-FISH картирования был использован фрагмент гена аллиназы луковицы Allbe1 A.fistulosum размером 1100 п.н. (Киров и др., 2011). TyramideFISH с меченым клоном Allbe1 показал, что ген аллиназы луковицы локализован в дистальной части (72±1.5% от центромеры) короткого плеча хромосомы 4 (Рисунок 38), которая была идентифицирована по относительной длине и центромерному индексу (42±0.5%). Очень слабые сигналы с редкой 112 встречаемостью были найдены после прямого метода детекции. Применение непрямого метода детекции позволило увеличить как интенcивность, так и частоту встречаемости сигнала. Полученные результаты на хромосомах A.fistulosum не соотносятся с результатом картирования данного гена на хромосомах A.cepa. Нами было показано, что ген аллиназы луковицы локализован в дистальной части длинного плеча хромосомы 4 A.cepa (Khrustaleva et al. 2012). Таким образом, ген аллиназы луковицы распологается в разных плечах гомеологичных хромосом 4 A.cepa и A.fistulosum. События, которые могли привести к подобным различиям, это транспозиция данного участка хромосомы, несущего гены, или перицентрическая инверсия. Транспозиция большого фрагмента хромосомы, несущего ген аллиназы, из одного плеча в другое теоретически невозможна, так как множественные выпетливания, обычно сопровождающие эту аберрацию, не были обнаружены при анализе пахитены у F1 гибридов между A.cepa и A.fistulosum (Levan, 1941). Физическое картирование гена аллиназы луковицы было также проведено на хромосомах близких видов, а именно, A. vavilovii и A. altaicum, A.schoenoprasum. Результаты Tyr-FISH показали, что физическое положение гена аллиназы корня на хромосомах A.altaicum (71.4±2.1) (Рисунок 37) соответствует положению данного гена на хромосомах A.fistulosum (72±1.5) (Рисунок 37). A.vavilovii имеет два сайта гибридизации на хромосомах 4 (78.6±1.1) и 8 (38±1.3) (Рисунок 37). У A.schoenoprasum был детектирован один сайт гибридизации в дистальной части хромосомы 4 (85.5±1.3), как у A.cepa (74.4±1). 113 Рисунок 37. Кариотипы близкородственных видов Allium после TyramideFISH c геном аллиназы луковицы. Полученные данные не соотносятся с филогенетическими данными на основе полиморфизма хлоропластной ДНК (Friesen et al. 2006). Только A. fistulosum и A. altaicum с очень близким генетическим родством, имеют абсолютно одинаковое положение гена аллиназы луковицы в дистальной части хромосомы 4. Так, например, локализация гена аллиназы луковицы у A.cepa и A.schoenoprasum совпадает. Однако, лук репчатый (A.cepa) и шнитт-лук (A.schoenoprasum) не только стоят удалѐнно друг от друга в филогенетическом дереве, но и имеют существенные различия в размерах геномов: 7.7 пг для A.schoenoprasum и 16 пг для A.cepa (Friesen et al. 2006). Поэтому вероятнее всего, что реорганизации хромосомы 4, у A.cepa и A.schoenoprasum произошли независимо после расхождения от общего предка. 114 3.4.1.2 Сравнительное физическое картирование генов на хромосоме 4 A.cepa и A.fistulosum Для того чтобы пролить свет на реорганизацию, обусловившую различия в положении генов аллиназ на хромосоме 4 A.cepa и A.fistulosum, было проведено картирование гена аллиназы корня II (ALL1, Do et al. 2004) и халкон синтазы (CHS, Мasuzaki et al. 2006), которые генетически были картированы на хромосоме 4 . Однако, авторами не было установлено физическоеположение этих генов на хромосоме. Сравнительное Tyramide-FISH картирование ALL1 и CHS на хромосомах A.cepa и A.fistulosum показало, что локализация данных генов отличается между двумя видами. Оба гена локализованы в дистальной части длинного плеча хромосомы 4 у A.cepa и в дистальной части короткого плеча хромосомы 4 у A.fistulosum (Рисунок 38). Рисунок 38. Tyramide-FISH локализация генов аллиназы луковицы, All1 и CHS на хромосоме 4 A.cepa и A.fistulosum. На основе локализации четырех генов на хромосоме 4 A.cepa и A.fistulosum, можно выдвинуть предположение о перицентрической инверсии, возникшей 115 после расхождения двух видов от общего предка. Результатом инверсии стали различия в центромерном индексе между гомеологичными хромосомами 4 A.cepa и A.fistulosum (у A.cepa – 42±0.5, а у A.fistulosum - 38±0.3) и локализация изученных генов в разных плечах хромосомы. Кроме этого, в ходе эволюции возможно произошла транлокация/делеция фрагмента хромосомы 4 A.cepa, несущего ген аллиназы корня I, локализованного в коротком плече хромосомы 4 A.cepa. Результаты представляют первое прямое доказательство присутствия инверсии, отличающей геномы A.cepa и A.fistulosum. Предположение о наличии такой инверсии были высказаны более 70 лет назад Леваном (1941) на основе анализа мейоза у F1 гибридов между A.cepa и A.fistulosum. 3.4.1.2.3 Гены аллиназы корня I и LFS Фрагмент гена аллиназы корня I A.cepa, соответствующего EST клону AOB249, был клонирован и секвенирован. Размер данного фрагмента составил 1100 п.н.. Для физического картирования клон (ACR1/4.4), содержащий целевую вставку, был использован в Tyramide-FISH с хромосомами A.cepa и A.fistulosum. Результаты показывают, что ген аллиназы корня локализован в коротком плече хромосомы 2 A.fistulosum (64±1.5%) (Рисунок 39). Рисунок 39. Результат Tyramide-FISH с фрагментом гена аллиназы корня (1100 п.н.) на хромосомах A.fistulosum. В противоположность этому, данный ген был картирован в 5 локусах на 3 хромосомах A.cepa: хромосома 2, хромосома 4 и хромосома 6 (Рисунок 27А). Данные по числу копий, полученные для A.cepa, не совпадают с данными 116 генетического картирования. Согласно генетическому картированию ген, кодирующий данную изоформу аллиназы, представлен в геноме A.cepa как минимум двумя копиями (паралогами). Обе копии картированы и показана их принадлежность к были генетически группам сцепления, соответствующим хромосомам 4 и 6 (Martin et al. 2005). Для физического картирования гена LFS был проведен Tyramide-FISH на хромосомах A.cepa, A.fistulosum, A.vavilovii и A.proliferum c использованием меченого клонированного фрагмента гена LFS (550 п.н., Киров и др. 2011). Результаты Tyramide-FISH показали, что ген LFS локализован в прицентромерном регионе хромосомы 5 (Рисунок 40) у всех видов. Данные о локализации LFS на хромосомах A.cepa совпадают с ранее полученными результатами с помощью BAC-FISH (Masamura et al. 2012). Различия в положении LFS между видами не были обнаружены. Рисунок 40. Tyramide-FISH с меченым геном LFS на хромосомах A.cepa (A), A.fistulosum (Б), F1 A.cepa x A.vavilovii (В), A.proliferum (Г). 117 Ген LFS имеет консерватиную хромосомную локализацию у трѐх изучаемых видов и высокий уровнень схожести его нуклеотидной последовательности между A. cepa и A. fistulosum (Киров и др., 2011; Masamura et al. 2012). Консервативное положение гена LFS среди изученных видов может быть связано с его проксимальной локализацией. Накопившиеся на сегодня данные о распределении частоты реорганизаций вдоль хромосомы позволяют сделать вывод о неслучайности локализации реорганизаций. Как правило, реорганизации возникают чаще около теломеры и реже у центромеры. Причины этого феномена доконца не установлены, но предполагается, что это связано, прежде всего, с супрессией рекомбинации около центромеры (Akhunov et al. 2003). В результате физического in situ картирования было установлено положение на хромосомах A.cepa и A.fistulosum 5 генов, вовлеченных в образование специфических вкуса и запах лука (гены аллиназ и ген LFS), а также окраски луковицы (ген CHS). Гены были картированы в 9 и 5 локусах на хромосомах A.cepa и A.fistulosum, соответственно (Рисунок 41, Таблица 7). 118 Рисунок 41. Физическая локализация генов аллиназ, LFS и CHS, установленная с помощью Tyramide-FISH. Таблица 7. Размер проб и физическое положение генов, использованных в Tyramide-FISH, на хромосомах A.cepa и A.fistulosum Название гена Аллиназа корня I (AOB249) Размер пробы, п.н. 1100 Аллиназа луковицы 1100 Аллиназа корня II (ALL1) 1100 LFS 550 CHS 1000 Хромосомная локализация Хромосома 2: 2S 2L 2L Хромосома 4: 4S Хромосома 6: 6S Хромосома 4: 4S 4L Хромосома 4: 4L 4S Хромосома 5 Хромосома 4: 4L 4S Физическое положение, %1 A.cepa A.fistulosum 37±1.5 44±1.1 73±1.5 36±1.5 н/д н/д 28±1.5 н/д 30±1.2 н/д н/д 28±1.5 2 26±1.1 н/д 33±1 н/д 74±0.7 н/д 60±2 74±1.2 44±1.2 н/д н/д 55±3 1 - расстояние от теломеры данного плеча до сигнала х 100%/длина плеча 2 - данные заимствованы из Романов (2012) 119 н/д – не было детектировано 3.4.1.4 Интегрирование генетической и физической карт A.cepa Группы сцепления, созданной генетической карты для A.cepa, были привязаны к индивидуальным хромосомам с использованием моносомно дополненных линий (Martin et al. 2005). Благодаря тому, что из 9 локусов, картированных с помощью Tyramide-FISH на хромосомах A.cepa, 4 локуса были до этого картированы генетически (Martin et al. 2005; King et al. 1998), мы смогли провести интегрирование генетической и физической карт для хромосом 4 и 6. Ген аллиназы корня I был генетически картирован, как EST клон AOB249 в двух группах сцепления, соответствующих хромосомам 4 и 6. Интегрирование позволило установить, что нижняя часть группы сцепления хромосомы 6 соответствует короткому плечу данной хромосомы. Группа сцепления хромосомы 4 соответствует короткому плечу данной хромосомы (Рисунок 42). Рисунок 42. Интегрирование физической и генетической карт хромосом 4 и 6 A. cepa. 120 Романов (2012) провел интегрирование карт для хромосомы 4, а Masamura et al. (2012) интегрировали положение гена LFS на генетической и физической картах хромосомы 5. В итоге, проведено интегрирование 5 групп сцепления с физическими хромосомами 1, 4, 5 и 6. 3.4.2 Rosa wichurana 3.4.2.1 Физическое картирование генов на митотических хромосомах R.wichurana Для идентификации индивидуальных хромосом R.wichurana, несущих сигналы гибридизации генов, было проведено кариотипирование на основании измерения размера хромосом и центромерного индекса (Таблица 8, Рисунок 43). Таблица 8. Размер и центромерный индекс хромосом R.wichurana Номер хромосомы 1 2 3 4 5 6 7 Длина хромосомы, мкм 3.70 ± 0.30 3.20 ± 0.60 3.00 ± 0.50 2.80 ± 0.40 2.60 ± 0.40 2.50 ± 0.40 2.20 ± 0.50 Относительная длина, % 17.80 ± 0.20 17.00 ± 0.20 15.20 ± 0.20 14.00 ± 0.10 13.60 ± 0.10 12.40 ± 0.20 10.00 ± 0.10 Центромерный индекс, % 46.00 ± 1.20 40.30 ± 1.30 44.30 ± 1.00 36.90 ± 0.70 41.40 ± 0.70 41.80 ± 1.10 23.40 ± 0.90 Рисунок 43. Идиограмм хромосом R.wichurana (Kirov et al. 2014а) 121 Кариотип R.wichurana содержит 5 метацентрических хромосом, 1 субметацентрическую (хромосома 4) и 1 субтелоцентрическую хромосому (хромосома 7, спутничная). Кроме этого, проведенные измерения позволили расчитать степень конденсации хромосом R.wichurana, которая составила 28.1±2 млн.п.н./мкм (размер генома (1C = 562 Mb, (Yokoya et al. 2000))/длину всех хромосом (1n = 20±1 мкм) при компактизированы по всей длине. условии, что хромосомы одинаково Учитывая разрешающую способность флуоресцентного микроскопа (0,25мкм) и степень конденсации хромосом, разрешающая способность FISH на хромосомах R.wichurana составляет 7 млн.п.н. (28х0,25). Для физического картирования были выбраны гены, ответственные за устойчивость розы к мучнистой росе (гены MLO1, MLO2, MLO3), формирование запаха розы (ген OOMT) и устойчивость к абиотическим факторам (PAL, P5CS, Таблица 9). Таблица 9. Гены розы и их физическое положение на хромосоме, установленное с помощью Tyramide-FISH на митотической хромосоме. Ген Аббревиатура Размер пробы, Хромосома Физическое п.н. положение, %1 Орцинол-О-метил 77±3.2 OOMT 1100 1 трансераза Локус устойчивости к мучнистой росе О Пирролинe-5Карбоксилат Синтаза Локус устойчивости к мучнистой росе О Фенилаланин аммония лиаза Локус устойчивости к мучнистой росе О 1 MLO1 1750 2 57±5 P5CS 1700 4 27±3.8 MLO3 1750 4 31±2.3 PAL 1700 7 23±2.1 MLO2 1850 7 27±4 - расстояние от теломеры данного плеча до сигнала х 100%/длина плеча 122 С помощью оптимизированной непрямой детекции Tyramide-FISH (см. Главу 3.1.2.6) 6 генов были успешно картированы на хромосомах, приготовленных методом ―SteamDrop‖ (Рисунок 44). Рисунок 44. Результаты Tyramide-FISH с фрагментами (от 1100 п.н.) целевых генов. Линейка – 10 мкм Гены OOMT и MLO1 были картированы в проксимальной части хромосомы 1 (77±3.2) и интерстициальной части хромосомы 2 (57±5), соответственно. Гены PAL и MLO2 были картированы в дистальной части хромосомы 7. Однако, рассчитанное физическое положения для PAL и MLO2 (27±3.8 и 31±2.3) не позволило определить точный порядок этих генов не хромосоме 7. Гены P5CS и MLO3 были картированы в дистальной части хромосомы 4. Как и в случае с генами PAL и MLO2, порядок генов P5CS и MLO3 не возможно было установить, опираясь только на рассчитанное физическое положение каждого гена. Результаты этого эксперимента показали: 1) короткие фрагменты генов (от 1100 п.н.) могут быть успешно физически картированы на хромосомах R.wichurana с помощью оптимизированной непрямой системы детекции Tyramide-FISH; 2) препараты, приготовленные с помощью ―SteamDrop‖, обеспечивают достаточное количество метафаз высокого качества для успешного физического картирования коротких ДНК проб на хромосомах R.wichurana; 3) разрешающая способность Tyramide-FISH (7 млн.п.н.) на мелких митотических хромосомах R.wichurana недостаточна для картирования близко расположенных генов. 123 3.4.2.2 Высокоразрешающая и многоцветная Tyramide-FISH на пахитенной хромосоме R.wichurana Для увеличения разрешающей способности физического картирования генома R.wichurana, была применена Tyramide-FISH на пахитенной хромосоме (высокоразрешающая Tyramide-FISH). Модифицированный протокол ―SteamDrop‖ позволил приготовить препараты пахитенных хромосом высокого качества. Окрашивание препаратов пахитенных хромосом DAPI не выявило для большинства хромосом (бивалентов) блоков гетерохроматина, которые могли бы быть использованы в качестве цитогенетических маркеров для распознавания индивидуальных хромосом, как это было сделано для хромосом томата (Zhong et al. 1996). Только хромосомы 4 и 7 могли быть распознаны, благодаря четким паттернам гетерохроматина в коротком плече. Кроме этого, хромосома 7 имеет спутник (ядрышкообразущий регион, ЯОР) в конце короткого плеча, что также служит маркером для данной хромосомы. Для этих двух пахитенных хромосом были построены карты расположения гетерохроматина и проведены расчеты длины хромосом, ценромного индекса и содержания гетерохроматина (Рисунок 45, Таблица 10). Эти данные послужат в дальнейшем для детального физического картирования генома R.wichurana и более точного расчета частоты рекомбинации вдоль хромосомы. 124 Рисунок 45. Инвертированная фотография пахитенных хромосом 4 (А) и 7 (В) R.wichurana после DAPI окрашивания и идиограмма. Звездочками отмечено положение центромеры. Таблица 10. Характеристика пахитенных хромосом 4 и 7 R.wichurana Хромо сома Длина хромосом ы, мкм Длина короткого плеча, мкм Длина длинного плеча, мкм Гетерохр оматин короткого плеча, %z Гетерохром атин длинного плеча, %y 4 7 45.6±5.4 11.4±1.4 34.0±4.0 62.0±6.0 21.0±4.5 38.4±4.2 4.0 ±0.6 33.3±3.5 88.0±6.7 16.8±4.4 Всего гетерохро матина, %x Центромерн ый индекс 31.0±4.0 25.0±0.5 24.3±2.3 10.3±0.8 ЯОР na + 7 9 z – рассчитано по формуле: длина гетерохроматина короткого плеча x 100%/длина короткого плеча – рассчитано по формуле: длина гетерохроматина длинного плеча x 100%/длина длинного плеча x – рассчитано по формуле: (длина гетерохроматина длинного плеча + длина гетерохроматина короткого плеча) x 100%/длина хромосомы a – число пахитенных хромосом, использованных в измерениях y Общая длина всех пахитенных хромосом R.wichurana варьировала от 235 мкм до 411 мкм, что в 10-20 раз больше длины митотических хромосом (20±1 мкм, Kirov et al. 2014). Схожие значения были ранее получены для томата (Zhong et al. 1998), банана (De Capdeville et al. 2009), арабидопсиса (Fransz et al 1998) и риса (Cheng et al. 2001). Степень компактизации пахитенных хромосом R.wichurana составляет 2.4 - 1.4 млн.п.н./мкм, что соответствует разрешающей способности Tyramide-FISH равной от 270 (для более ранней пахитены) до 500 тыс. п.н. (поздняя пахитена). Различия в длине митотической и пахитенной хромосом R.wichurana представлены на Рисунке 46 на примере хромосомы 7. 125 Рисунок 46. Различия в длине хромосомы 7 на стадии пахитены и митотической метафазы (М). Красная флуоресценция – результат Tyramide-FISH с геном MGM. Приготовленные ―SteamDrop‖ препараты пахитенных хромосом R.wichurana были использованы для картирования целевых генов. К целевым генам относились 11 генов (Таблица 11), из которых: 6 генов (MLO1, MLO2, MLO3, OOMT, PAL, P5CS) были картированные на митотической хромосоме с помощью Tyramide-FISH; 5 генов (AAA-2, MDAR, MGM, AD1, Nar) были выбраны на основе знаний о локализации этих генов в секвенированном геноме Fragaria vesca (Shulaev et al. 2011, FraVesHawaii_1.0), как сцепленных с MLO2, PAL (MGM, MDAR) или MLO3, P5CS (AAA-2) или OOMT (AD1, Nar). Размер ДНК фрагментов, используемых для Tyramide-FISH в качестве пробы, составлял от 1100 до 3500 п.н. (Таблица 11). 126 Таблица 11. Гены розы и их физическое положение на хромосоме, установленное с помощью высокоразрешающей Tyramide-FISH Название гена Аббревиатура Хромосома 2 Физическое положение, %1 Размер пробы Локус устойчивости к мучнистой росе О Пирролинe-5Карбоксилат Синтаза MLO3 4L 44.0 ± 1.5 1750 P5CS 2 4L 30.0 ± 2.0 1700 АТФаза Монодегидроаскорбат редуктаза AAA-2 MDAR 4L 7L 8.0 ± 1.0 52.0 ± 1.5 3500 3000 Фенилаланин аммония PAL2 лиаза 2 Локус устойчивости к MLO2 мучнистой росе О 7L 46.6 ± 1.0 1700 7L 44.8 ± 0.5 1850 7L 18.0 ± 1.5 3000 2S 56±2 1750 1p 94±0.1 1100 1q 1q 72±1.2 41±1.5 3000 1300 Маннозил гликопротеин эндобета-маннозидаза MGM Локус устойчивости к мучнистой росе О Орцинол-О-метил трансераза MLO1 Аннексин D1 Нарингенин 2 OOMT AD1 Nar 2 1 - расстояние от теломеры плеча до сигнала х 100%/длина плеча 2 - гены, которые были картированны на митотической хромосоме Все 11 генов были успешно картированы по отдельности на пахитенных хромосомах R.wichurana (Таблица 11) и определено их физическое положение. ЧВС для генов составила в среднем 70%, что на порядок выше ЧВС, наблюдаемой после Tyramide-FISH на митотической хромосоме (Perez et al. 2009; Kirov et al. 2014). Для уточнения положения близкорасположенных генов были проведены Tyramide-FISH эксперименты с парами генов, для хромосомы 7: MDAR+PAL, PAL+MLO2, MLO2+MGM; и для хромосомы 4: P5CS+MLO3, MLO3+AAA-2; хромосома 1: AD1+Nar, Nar+OOMT (Рисунок 47B, G, H). 127 Рисунок 47. Результат высокоразрешающей Tyramide-FISH с фрагментами целевых генов (1100 – 3500 п.н.); A. Пахитена R.wichurana после FISH с 45S рДНК, используемая для последующего Tyramide-FISH картирования гена MGM, локализованного на хромосоме 7 (D); F. Двуцветная высокоразрешающая Tyramide-FISH с ML02, MGM и PAL генами; Одноцветная высокоразрешающая Tyramide-FISH с одним геном: C - AAA2, E - MDAR, I - MLO1 и двумя генами: В - MLO3/P5CS, G - Nar/AD1, H - Ad1/OOMT Стрелки показывают положение центромеры. С помощью высокоразрешающего физического картирования был установлен порядок генов P5CS+MLO3 (Рисунок 47В), что невозможно было сделать при помощи Tyramide-FISH на митотической хромосоме. Однако, 128 высокоразрешающее картирование не позволило установить порядок близколокализованных генов PAL и MLO2 на хромосоме 7. Для решения этой проблемы нами впервые был оптимизирован и применен на хромосоме растений двухцветный Tyramide-FISH. Используя всего один тип тирамид (тирамидбиотин) и два типа антител (стрептавидин-Cy3, стрептавидин-FITC), мы смогли визуализировать положение трех генов на пахитенной хромосоме 7 (Рисунок 47F). Из 17 пахитенных хромосом, несущих сигналы от PAL и MLO2, в 8 (47%) случаях сигналы от двух генов (красный и зеленый) частично перекрывались, а в 9 (47%) случаях красный и зеленый сигналы были различимы как отдельные. Это позволило установить порядок генов и их точное относительно друг друга физическое положение. На основе длины пахитенных хромосом 7 (41±1.5 мкм), несущих неперекрывающиеся сигналы, относительной длины хромосомы 7 (10.0 ± 0.1%), размера генома (562Mb/1C) и разрешающей способности микросокопа (0,25мкм), можно сделать вывод, что физическое расстояние между PAL и MLO2 составляет около 340 тыс.п.н. Результаты высокоразрешающей и двухцветной Tyramide-FISH позволили составить физическую карту 11 генов, локализованных на 4 хромосомах R.wichurana (Таблица 11, Рисунок 48). Рисунок 48. Физическая (хромосомная) карта локализауии генов, построенная с использованием Tyramide-FISH на митотической и пахитенной (высокоразрешающая и двуцветная Tyramide-FISH) хромосомах. 129 В заключение, мы рекомендуем на розе – виде с мелкими хромосомами - для физического картирования генов и последующего интегрирования физических и генетических карт использовать менее конденсированные пахитенные хромосомы. Стоит отметить, что использование препаратов пахитенных хромосом в Tyramide-FISH картировании показало, что время дополнительной обработки уксусной кислотой существенно влияет на результаты Tyramide-FISH. Так, оптимальное время было 30 секунд, в то время как, обработка в течение 15 или 60 секунд приводила к резкому снижению ЧВС. Это наблюдение хорошо согласуется с данными, полученными на митотической хромосоме лука и розы, и является дополнительным свидетельством особой значимости способа приготовления препаратов для успешной Tyramide-FISH. 3.4.2.3 Генетическое картирование генов. Интегрирование генетической и физической карт R.wichurana Ни один из физически картированных нами генов не был картирован генетически. Нами было проведено генетическое картирование 3 генов, а именно OOMT, P5CS и PAL, распределенных по 3 хромосомам (1, 4 и 7). Для генетического картирования была использована популяция F1 (90 растений), полученаая от скрещивания R.wichurana и R. x “Yesterday” и ранее использованная для конструирования генетических карт R.wichurana (Moghaddam et al. (2012). Были подобраны праймеры, позволяющие амплифицировать фрагменты этих генов размером 200-300 п.н. Последовательность ДНК гена для подбора праймеров выбирали различий, выявленных при на основе присутствия однонуклеотидных помощи R.wichurana и R.x “Yesterday” (Таблица 12). выравнивания последовательностей 130 Таблица 12. Праймеры для амплификации фрагментов генов для метода высокоразрешающего плавления (HRM), используемого в генетическом картировании генов, и информация об ампликоне и числе сайтов с однонуклеотидным полиморфизмом (SNP). Ген PAL Праймеры (5’-3’) TTGGAGGTTCAAGGAATTTACC CCAAGAAGCGAAAAAGCTCA OOMT GTTTGAGGCAGTTCCTCCTG GGTCTTGGTCCAGATCGAGT P5CS GTGCTTGCAAACATGGAAGA TGGTGCTCTAGTTGGCAAAA z Нет информации для Rosa wichurana Ампликон (п.н.) Число интронов 227 1 Число ожидаемых SNP /z 223 1 1 204 1 1 Для детекции SNP были использованы HRM (высокоразрешающее плавление) маркеры (Croxford et al. 2008). HRM маркеры позволили в течении 3 часов в 384 луночном планшете провести картирование всех трех генов (Kirov et al. 2014). HRM профайлы кривых плавления у родителей для генов OOMT и PAL были различны, а у растений F1 соответствовали одному из родителей. Согласно JoinMap 4.0 (Van Ooijen and Voorrips 2004) в этом случае генотипы родителей по генам OOMT и PAL соответсвовали <lm x ll>. HRM профайлы кривых плавления у родителей для гена P5CS были различны, а у растений F1 наблюдалось 4 типа кривых, два из которых были идентичны типам кривых родителей (Рисунок 49). Рисунок 49. HRM кривые плавления для ампликонов гена P5CS, полученных с ДНК R.wichurana (часть кластера зеленых кривых), R.x “Yesterday”( часть кластера красных кривых) и ратений F1. Кривые плавления для растений F1 131 входят в часть кластеров зеленых и красных кривых и включают все розовые и голубые кривые. Согласно JoinMap 4.0 (Van Ooijen and Voorrips 2004) генотипы родителей по гену P5CS соответсвовали <ef x eg>. Паттерны расщепления для каждого гена, полученные на популяции F1, позволили установить положение трех генов на генетической карте (Moghaddam et al. (2012)) (Рисунок 47). Гены OOMT, P5CS и PAL были картированы в группах сцепления 2 (RwLG-B2), 1 (RwLG-B1) и 3 (RwLG-B3), соответственно. Генетическое картирование позволило установить тесное сцепление (3сМ) между геном PAL и локусом размера цветка (Flower_size). Таким образом, данные генетического и физического картирования согласуются, т.к. три гена, картированные в трех хромосомах, локализованы в 3 группах сцепления (Рисунок 50, Kirov et al. 2014а). Рисунок 50. Интегрирование генетической карты (RwLG), полученной с помощью Joinmap 4.0, и физической карты для хромосом 1 (RwChr1), 4 (RwChr4) и 7 (RwChr7) R.wichurana и псевдохромосом 6 (FvChr6) и 7 (FvChr7) F.vesca (FraVesHawaii_1.0, NCBI). Генетические карты сконструированны ранее (Moghaddam et al. (2012)). R.wichurana были 132 Приведенные исследования позволяют сделать вывод, что группы сцепления RwLG-B2, RwLG-B1 и RwLG-B3 относятся к хромосомам 1, 7 и 4, соответственно. Кроме этого, интегрирование карт позволяет сделать предположение о локализации гена OOMT в регионе с низкой частотой рекомбинации, о чем говорит его проксимальное физическое положение на хромосоме 1 и более высокая плотность маркеров на генетической карте в районе его локализации (Рисунок 50). Так как Fragaria vesca является близким видом для R.wichurana (Gar et al. (2011)) и геном этого вида секвенирован, мы провели сравнение физического положения трех генетически картированных генов на псевдохросомах F.vesca и R.wichurana. Результаты сравнения показали, что FwChr6 содержит ортологичные OOMT и PAL гены, локализованные на двух разных хромосомах R.wichurana (Рисунок 50). Ранее Gar et al. (2011) провели генетическое картирование маркеров ортологичных с F.vesca на тетраплоидной розе. Результаты этого исследования выявили 4 транслокации и 6 инверсий, отличающих геномы двух видов. Одна из обнаруженных хромосомных перестроек затрагивала псевдохромосому FvChr6 F.vesca, которая, как было показано, несет маркеры, принадлежащие двум группам сцепления R.wichurana (Gar et al. (2011)). Таким образом, наши результаты физического картирования генов на хромосомах R.wichurana свидетельствуют о наличии данной реорганизации и в геноме R.wichurana. Дальнейшее картирование большего числа генов и сравнение интегрированной карты R.wichurana и физической карты F.vesca позволит установить регионы с высокой синтенией и коллинеарностью. Знания синтении и и коллинеарности между геномами R.wichurana и F.vesca позволят использовать геном F.vesca для клонирования генов R.wichurana, контролирующих важные признаки и отвечающие за генетически картированные QTL. В заключение, наши результаты демонстрируют, что Tyramide-FISH является ценным инструментом для физического картирования генома R.wichurana. Оптимизированные протоколы для Tyramide-FISH на митотической 133 и пахитенной хромосомах позволяют проводить картирование с разным уровнем разрешения. Используя возможности оптимизированной Tyramide-FISH и высокопроизводительного HRM маркирования, мы смогли интегрировать 3 группы сцепления с соответствующими физическими хромосомами. Полученные данные могут быть использованы в будущем для ускоренного клонирования генов, а также для сборки генома розы. ЗАКЛЮЧЕНИЕ Физическое картирование отдельных генов и их фрагментов на хромосоме растений остается сложной задачей для большинства видов. Это обусловлено рядом причин: плотной компактизацией митотической хромосомы растений, наличием клеточной стенки и сложностью приготовления препаратов хромосом, отсутствием оптимизированных систем детекции и хорошо разработанных систем цитогенетического маркирования. В данной работе мы провели разработку и оптимизацию ключевых этапов физического картирования генов на хромосомах растений. Результаты работы показывают, что ключевой фактор в картировании коротких фрагментов ДНК на хромосоме является способ приготовления хромосом. Как на митотической, так и на пахитенной хромосоме (для R.wichurana) изменение условий приготовление препарата обуславливало значительные изменения в частоте детекции сигнала Tyramide-FISH. Кроме этого, нами была проведена оптимизация прямого и непрямого методов детекции. Оптимизированные протоколы позволили добиться оптимального соотношения интенсивность фона/сингала и провести картирование генов. Использование сравнительного Tyramide-FISH картирования генов семейства аллиназ на разных видах Allium позволило впервые выявить хромосомные различия внутри рода. Нами показана возможная хромосомная инверсия, возникшая после расхождения A.cepa и A.fistulosum от общего предка. Предположение о наличии такой инверсии были высказаны более 70 лет назад Леваном (1941) на основе анализа мейоза у F1 гибридов между A.cepa и A.fistulosum. 134 Обнаруженный высококопийный повтор HAT58 – может быть использован для сортинга отдельных хромосом (FISHIS, Giorgi et al. 2013) для последующего секвенирования. Данные результаты свидетельствуют, что Tyramide-FISH – ценный инструмент для сравнительной геномики растений. Так как геномы растений богаты повторяющимися элементами ДНК, то использование отдельных генов для сравнительного картирования позволяет преодолеть целый ряд трудностей, связанных с неспецифической гибридизацией. Физическое картирование коротких фрагментов на хромосомах R.wichurana с помощью оптимизированной высокопроизводительным HRM Tyramide-FISH маркированием совместно сделали с возможным интегрирование физической и генетической карт R.wichurana (Kirov et al. 2014, Kirov et al. 2015). Увеличение числа генетически и физически картированных маркеров для R.wichurana позволит в дальнейшем пролить свет на распределение плотности рекомбинации вдоль хромосомы и уровень синтении с другими видами семейства Rosaceae, чьи геномы были секвенированы (Fragaria vesca, Malus x domestica, Prunus persica, Pyrus communis). В связи с продолжающимися проектами по секвенированию геномов A.cepa (http://www.oniongenome.net/) и Rosa (Foucher et al. 2013) результаты данной работы представляют особую значимость для сборки геномов. Разработанные платформы для физического картирования генов на хромосомах исследуемых видов могут быть использованы на других видах растений. 135 ВЫВОДЫ 1. Оптимизированные протоколы ―SteamDrop‖ и Tyramide-FISH позволяют успешно физически картировать короткие фрагменты генов (от 550 п.н.) и ДНК повторов на мелких (R.wichurana) и крупных (виды Allium) хромосомах. 2. Разработанный протокол ―SteamDrop‖ позволил приготовить высококачественные препараты митотических и пахитенных хромосом для видов с мелкими и крупными хромосомами за короткое время и высоким выходом препаратов с одной меристемы. 3. Установлено, что разброс хромосом зависит от влажности (оптимальная 4555%) и температуры воздуха (оптимальная 23-26°С). 4. Использование препаратов, приготовленных ―SteamDrop‖ с пониженной концентрацией уксусной кислоты (15% для хромосом Allium и 50% для хромосом Rosa wichurana) существенно (в 2-3 раза) повышает частоту встречаемости хромосом с сигналом после Tyramide-FISH детекции. 5. Оптимизированная непрямая система детекции с использованием повышенной концентрации тирамид-биотин (1:25 вместо 1:50) и антител (1:100 вместо 1:250) позволяет физически картировать фрагменты генов (от 1100 п.н.) на хромосомах R.wichurana с частотой встречаемости хромосом с сигналом до 51%. 6. Выявлен новый цитогенетический маркер - высококопийный (160.000 копий/1C), видоспецифичный, тандемный повтор A.fisulosum HAT58, для идентификации хромосом 6, 7 и 8. 7. Установлено наличие наряду с ожидаемой теломерной локализацией теломерного повтора Arabidopsis типа (GGGATTT)n дополнительные сайты его гибридизации в центромерном регионе хромосом 1, 2, 5 и 7, что в сочетании с показателями центромерного индекса позволяет идентифицировать все митотические метафазные хромосомы R.wichurana. 8. Выявлено, что гены аллиназы луковицы, алиназы корня II (ALL1) и халкон синтазы (CHS) локализованы в коротком плече хромосомы 4 у A.cepa, а у 136 A.fistulosum в гомеологичной хромосоме 4 эти же гены расположены в длинном плече, что указывает на события инверсии и делеции/транслокации участка хромосомы после дивергенции этих видов от общего предка. 9. Успешно картированы 11 одно-локусных генов на пахитенных хромосомах R.wichurana с помощью оптимизированной высокоразрешающей и многоцветной Tyramide-FISH. 10. С помощью HRM маркирования было проведено генетическое картирование генов P5CS, PAL и OOMT на популяции F1 от скрещивания R.wichurana и R.x ’Yesterday’ , что позволило поместить эти гены в существующую генетическую карту R.wichurana, созданную на той же популяции. 11. Получены интегрированные генетические и физические карты для хромосом 1,4 и 7 R.wichurana и 4, 6 A.cepa. 137 СПИСОК ЛИТЕРАТУРЫ Кокорева В. А., Титова И. В.: Лук, чеснок и декоративные луки, НиолаПресс, ЮНИОН-паблик, М: 2007 Романов Д. В., Киселева А. В., Хрусталева Л. И. Физическое картирование генов на хромосомах лука репчатого (Allium cepa l.) С использованием ESTклонов и Tyramide-FISH // Известия ТСХА. – 2014.-№1. – С. 105 - 114. Романов Д.В.: Особенности хромосомной организации протеин-кодирующих генов у Allium cepa и Allium fistulosum. Диссертация на соискание ученой степени кандидата биологических наук, Москва, 2012. Abbasi F. M., Ahmad H., Perveen F., Inamullah M., Sajid M., Brar D.S. Assessment of genomic relationship between Oryza sativa and Oryza australinesis //African Journal of Biotechnology. – 2012. – Т. 9. – №. 9. Abbo S., Dunford R. P., Miller T. E., Reader S. M., King I. P. Primer-mediated in situ detection of the B-hordein gene cluster on barley chromosome 1H //Proceedings of the National Academy of Sciences. – 1993. – Т. 90. – №. 24. – С. 1182111824. Acevedo-Garcia J., Collins N.C., Ahmadinejad N., Ma L., Houben A., Bednarek P., Benjdia M., Freialdenhoven A., Altmüller J., Nürnberg P., Reinhardt R., Schulze-Lefert P, Panstruga R. Fine mapping and chromosome walking towards the Ror1 locus in barley (Hordeum vulgare L.) //Theoretical and applied genetics. – 2013. – Т. 126. – №. 12. – С. 2969-2982. Adams J. C. Biotin amplification of biotin and horseradish peroxidase signals in histochemical stains //Journal of Histochemistry & Cytochemistry. – 1992. – Т. 40. – №. 10. – С. 1457-1463. Akasaka M., Ueda Y., Koba T. Karyotype analyses of five wild rose species belonging to septet A by fluorescence in situ hybridization //Chromosome science. – 2002. – Т. 6. – №. 1. – С. 17-26. 138 Akasaka M., Ueda Y., Koba T. Karyotype analysis of wild rose species belonging to septets B, C, and D by molecular cytogenetic method //Breeding Science. – 2003. – Т. 53. – №. 2. – С. 177-182. Akkara J. A., Senecal K. J., Kaplan D. L. Synthesis and characterization of polymers produced by horseradish peroxidase in dioxane //Journal of Polymer Science Part A: Polymer Chemistry. – 1991. – Т. 29. – №. 11. – С. 1561-1574. Alexandrov O. S., Divashuk M.G., Yakovin N.A., Karlov G.I. Sex chromosome differentiation in Humulus japonicus Siebold & Zuccarini, 1846 (Cannabaceae) revealed by fluorescence in situ hybridization of subtelomeric repeat //Comparative cytogenetics. – 2012. – Т. 6. – №. 3. – С. 239. Ami D., Di Segni M., Forcella M., Meraviglia V., Baccarin M., Doglia S.M., Terzoli G. Role of water in chromosome spreading and swelling induced by acetic acid treatment: a FTIR spectroscopy study //European journal of histochemistry: EJH. – 2014. – Т. 58. – №. 1. Ahn S., Tanksley S. D. Comparative linkage maps of the rice and maize genomes //Proceedings of the National Academy of Sciences. – 1993. – Т. 90. – №. 17. – С. 7980-7984. Ambros P. F., Matzke M. A., Matzke A. J. M. Detection of a 17 kb unique sequence (TDNA) in plant chromosomes by in situ hybridization //Chromosoma. – 1986. – Т. 94. – №. 1. – С. 11-18. Anamthawat-Jónsson K. Preparation of chromosomes from plant leaf meristems for karyotype analysis and in situ hybridization //Methods in Cell Science. – 2004. – Т. 25. – №. 3-4. – С. 91-95. Andras S. C., Hartman T.P.V., Marshall J.A., Marchant R., Power J.B., Cocking E.C., Davey M.R. A drop-spreading technique to produce cytoplasm-free mitotic preparations from plants with small chromosomes //Chromosome Research. – 1999. – Т. 7. – №. 8. – С. 641-647. Arabidopsis Genome Initiative et al. Analysis of the genome sequence of the flowering plant Arabidopsis thaliana //Nature. – 2000. – Т. 408. – №. 6814. – С. 796. 139 Argyris J. M., Ruiz-Herrera A., Madriz-Masis P., Walter Sanseverino W., Morata J., Pujol M., Ramos-Onsins S.E., Garcia-Mas J. Use of targeted SNP selection for an improved anchoring of the melon (Cucumis melo L.) scaffold genome assembly //BMC Genomics. – 2015. – Т. 16. – №. 1. – С. 4. Ariyadasa R., Mascher M., Nussbaumer T., Schulte D., Frenkel Z., Poursarebani N., Zhou R., Steuernagel B., Gundlach H., Taudien S., Felder M., Platzer M., Himmelbach A., Schmutzer T., Hedley P.E., Muehlbauer G.J., Scholz U., Korol A., . Mayer K.F.X., Waugh R., Langridge P., Graner A., Stein N. A sequence-ready physical map of barley anchored genetically by two million single-nucleotide polymorphisms //Plant physiology. – 2014. – Т. 164. – №. 1. – С. 412-423. Badaeva E. D., Shkutina F.M., Bogdevich I.N., Badaev N.S. Comparative study of Triticum aestivum and T. timopheevi genomes using C-banding technique //Plant systematics and evolution. – 1986. – Т. 154. – №. 3-4. – С. 183-194. Baldwin S., Revanna R., Pither-Joyce M., Shaw M., Wright K., Thomson S., Moya L., Lee R., Macknight R., McCallum J. Genetic analyses of bolting in bulb onion (Allium cepa L.) //Theoretical and Applied Genetics. – 2014. – Т. 127. – №. 3. – С. 535-547. Benson G. Tandem repeats finder: a program to analyze DNA sequences //Nucleic acids research. – 1999. – Т. 27. – №. 2. – С. 573. Barch MJ, Knusten T, Spurbeck JL. The AGT cytogenetics laboratory manual. 3rd ed. New York: Raven Press; 1997. p 25–50. Bhattacharya D., Bhattacharya R., Dhar T. K. A novel signal amplification technology for ELISA based on catalyzed reporter deposition. Demonstration of its applicability for measuring aflatoxin B 1 //Journal of immunological methods. – 1999. – Т. 230. – №. 1. – С. 71-86. Blenda A., Fang D.D., Rami J.-F., Garsmeur O., Luo F., Lacape J.-M. A high density consensus genetic map of tetraploid cotton that integrates multiple component 140 maps through molecular marker redundancy check //PLoS One. – 2012. – Т. 7. – №. 9. – С. e45739. Bobrow M. N., Shaughnessy K. J., Litt G. J. Catalyzed reporter deposition, a novel method of signal amplification: II. Application to membrane immunoassays //Journal of immunological methods. – 1991. – Т. 137. – №. 1. – С. 103-112. Bolger M. E., Weisshaar B., Scholz U., Stein N., Usadel B. , Mayer K.F.X. Plant genome sequencing—applications for crop improvement //Current opinion in biotechnology. – 2014. – Т. 26. – С. 31-37. Bolsheva N. L, Dyachenko O.V., Samatadze T.E., Rachinskaya O.A., Zakharchenko N.S., Shevchuk T.V., Amosova A.V., Muravenko O.V., Zelenin A.V. Karyotype of Mesembryanthemum crystallinum (Aizoaceae) studied by chromosome banding, FISH with rDNA probes and immunofluorescence detection of DNA methylation //Plant Biosystems-An International Journal Dealing with all Aspects of Plant Biology. – 2014. – №. just-accepted. – С. 1-11. Brown A. H. D. Isozymes, plant population genetic structure and genetic conservation //Theoretical and Applied Genetics. – 1978. – Т. 52. – №. 4. – С. 145-157. Busch W., Martin R., Herrmann R. G. Sensitivity enhancement of fluorescencein situ hybridization on plant chromosomes //Chromosome Research. – 1994. – Т. 2. – №. 1. – С. 15-20. Cevik V., King G. J. Resolving the aphid resistance locus Sd-1 on a BAC contig within a sub-telomeric region of Malus linkage group 7 //Genome. – 2002. – Т. 45. – №. 5. – С. 939-945. Chao J., DeBiasio R., Zhu Z., Giuliano K.A., Schmidt B.F. Immunofluorescence signal amplification by the enzyme‐catalyzed deposition of a fluorescent reporter substrate (CARD) //Cytometry. – 1996. – Т. 23. – №. 1. – С. 48-53. Cheng B. F., Heneen W. K., Chen B. Y. Mitotic karyotypes of Brassica campestris and Brassica alboglabra and identification of the B. alboglabra chromosome in an addition line //Genome. – 1995. – Т. 38. – №. 2. – С. 313-319. 141 Cheng Z., Buell C.R., Wing R.A., Gu M., Toward J.J. A cytological characterization of the rice genome //Genome research. – 2001. – Т. 11. – №. 12. – С. 2133-2141. Clark M., Karp A., Archer S. Physical mapping of the B-hordein loci on barley chromosome 5 by in situ hybridization //Genome. – 1989. – Т. 32. – №. 5. – С. 925-929. Claussen U., Michel S., Mühlig P., Westermann M., Grummt U.-W., Kromeyer- Hauschild K., Liehr T. Demystifying chromosome preparation and the implications for the concept of chromosome condensation during mitosis //Cytogenetic and genome research. – 2002. – Т. 98. – №. 2-3. – С. 136-146. Cuadrado Á., Jouve N. Chromosomal detection of simple sequence repeats (SSRs) using nondenaturing FISH (ND-FISH) //Chromosoma. – 2010. – Т. 119. – №. 5. – С. 495-503. Cuadrado A., Schwarzacher T., Jouve N. Identification of different chromatin classes in wheat using in situ hybridization with simple sequence repeat oligonucleotides //Theoretical and Applied Genetics. – 2000. – Т. 101. – №. 5-6. – С. 711-717. Cui X., Liu F., Liu Y., Zhou Z., Zhao Y., Wang C., Wang X., Cai X., Wang Y., Meng F., Peng R., Wang K. Construction of cytogenetic map of Gossypium herbaceum chromosome 1 and its integration with genetic maps //Molecular Cytogenetics. – 2015. – Т. 8. – №. 1. – С. 2. Danilova T. V., Birchler J. A. Integrated cytogenetic map of mitotic metaphase chromosome 9 of maize: resolution, sensitivity, and banding paint development //Chromosoma. – 2008. – Т. 117. – №. 4. – С. 345-356. Danilova T. V., Friebe B., Gill B. S. Single-copy gene fluorescence in situ hybridization and genome analysis: Acc-2 loci mark evolutionary chromosomal rearrangements in wheat //Chromosoma. – 2012. – Т. 121. – №. 6. – С. 597-611. Danilova T. V., Friebe B., Gill B. S. Development of a wheat single gene FISH map for analyzing homoeologous relationship and chromosomal rearrangements within the Triticeae //Theoretical and applied genetics. – 2014. – Т. 127. – №. 3. – С. 715-730. 142 Darlington C. D., Wylie A. P. Chromosome atlas of flowering plants //Chromosome atlas of flowering plants. – 1956. De Capdeville G., Souza M.T.J., Szinay D., Eugênio L., Diniz C., Wijnker E., Swennen R., Kema G.H.J., De Jong H. The potential of high-resolution BAC-FISH in banana breeding //Euphytica. – 2009. – Т. 166. – №. 3. – С. 431443. De Vries D. P., Dubois L. A. M. Rose breeding: past, present, prospects //II International Rose Symposium 424. – 1995. – С. 241-248. Delaney D. E., Nasuda S., Endo T.R., Gill B.S., Hulbert S.H. Cytologically based physical maps of the group 3 chromosomes of wheat// Theoretical and Applied Genetics. – 1995. – Т. 91. – №. 5. – С. 780-782. Deng W., Tsao S.W., Lucas J.N., Leung C.S., Annie L. M. Cheung A new method for improving metaphase chromosome spreading //Cytometry Part A. – 2003. – Т. 51. – №. 1. – С. 46-51. Deokar A. A., Ramsay L., Sharpe A.G., Diapari M., Sindhu A., Bett K., Warkentin T.D., Tar‘an B. Genome wide SNP identification in chickpea for use in development of a high density genetic map and improvement of chickpea reference genome assembly //BMC genomics. – 2014. – Т. 15. – №. 1. – С. 708. Deryckere D., De Keyser E., Eeckhaut T., Van Huylenbroeck J., Van Bockstaele E. High resolution melting analysis as a rapid and highly sensitive method for Cichorium plasmotype characterization //Plant Molecular Biology Reporter. – 2013. – Т. 31. – №. 3. – С. 731-740. Dick C., Johns E. W. The effect of two acetic acid containing fixatives on the histone content of calf thymus deoxyribonucleoprotein and calf thymus tissue //Experimental cell research. – 1968. – Т. 51. – №. 2. – С. 626-632. Divashuk M. G., Alexandrov O.S., Razumova O.V., Kirov I.V., Karlov G.I. Molecular cytogenetic characterization of the dioecious Cannabis sativa with an XY chromosome sex determination system //PloS one. – 2014. – Т. 9. – №. 1. 143 Doerge R. W. Mapping and analysis of quantitative trait loci in experimental populations //Nature Reviews Genetics. – 2002. – Т. 3. – №. 1. – С. 43-52. Dohm J. C., Minoche A.E., Holtgräwe D., Capella-Gutiérrez S., Zakrzewski F., Tafer H., Rupp O., Sörensen T.R., Stracke R., Reinhardt R., Goesmann A., Kraft T., Schulz B., Stadler P.F., Schmidt T., Gabaldón T., Lehrach H., Weisshaar B., Himmelbauer H. The genome of the recently domesticated crop plant sugar beet (Beta vulgaris) //Nature. – 2014. – Т. 505. – №. 7484. – С. 546549. Dong F., Song J., Naess S.K., Helgeson J.P., Gebhardt C., Jiang J. Development and applications of a set of chromosome-specific cytogenetic DNA markers in potato //Theoretical and Applied Genetics. – 2000. – Т. 101. – №. 7. – С. 1001-1007. Duangjit J., Bohanec B., Chan A.P., Town C.D., Havey M.J. Transcriptome sequencing to produce SNP-based genetic maps of onion //Theoretical and Applied Genetics. – 2013. – Т. 126. – №. 8. – С. 2093-2101. Dufey I., Draye X., Lutts S., Lorieux M., Martinez C., Bertin P. Novel QTLs in an interspecific backcross Oryza sativa× Oryza glaberrima for resistance to iron toxicity in rice //Euphytica. – 2015. – С. 1-17. Dugo M. L., Satovic Z., Millán T., Cubero J.I., Rubiales D., Cabrera A., Torres A.M. Genetic mapping of QTLs controlling horticultural traits in diploid roses //Theoretical and applied genetics. – 2005. – Т. 111. – №. 3. – С. 511-520. Emsweller S. L., Jones H. A. Further studies on the chiasmata of the Allium cepa x A. fistulosum hybrid and its derivatives //American Journal of Botany. – 1945. – С. 370-379. Emsweller S. L., Jones H. A. A gene for control of interstitial localization of chiasmata in Allium fistulosum L. – American Association for the Advancement of Science, 1935. Endo T. R. Induction of chromosomal structural changes by a chromosome of Aegilops cylindrica L. in common wheat //Journal of Heredity. – 1988. – Т. 79. – №. 5. – С. 366-370. 144 Endo T. R., Gill B. S. The deletion stocks of common wheat //Journal of Heredity. – 1996. – Т. 87. – №. 4. – С. 295-307. Erayman M., Sanduh D., Sidhu D., Dilbirligi M., Baenziger1and P.S., Gill K.S.Demarcating the gene-rich regions of the wheat genome //Nucleic acids research. – 2004. – Т. 32. – №. 12. – С. 3546-3565. Feuillet C., Leach J.E., Rogers J., Schnable P.S., Eversole K. Crop genome sequencing: lessons and rationales //Trends in plant science. – 2011. – Т. 16. – №. 2. – С. 7788. Febrer M., Goicoechea J.L., Wright J., McKenzie N., Song X., Lin J., Collura K., Wissotski M., Yu Y., Ammiraju J.S.S., Wolny E., Idziak D., Betekhtin A., Kudrna D., Hasterok R., Wing R.A., Bevan M.W. An integrated physical, genetic and cytogenetic map of Brachypodium distachyon, a model system for grass research //PLoS One. – 2010. – Т. 5. – №. 10. – С. e13461. Fernández-Romero M. D., Torres A. M., Millán T., Cubero J. I., Cabrera, A. Physical mapping of ribosomal DNA on several species of the subgenus Rosa //Theoretical and Applied Genetics. – 2001. – Т. 103. – №. 6-7. – С. 835-838. Figueroa D. M., Bass H. W. Development of pachytene FISH maps for six maize chromosomes and their integration with other maize maps for insights into genome structure variation //Chromosome research. – 2012. – Т. 20. – №. 4. – С. 363-380. Fonsêca A., Ferreira J., Barros dos Santos T.R., Mosiolek M., Bellucci E., Kami J., Gepts P., Geffroy V., Schweizer D., Barros dos Santos K.G., PedrosaHarand A. Cytogenetic map of common bean (Phaseolus vulgaris L.) //Chromosome Research. – 2010. – Т. 18. – №. 4. – С. 487-502. Foucher F., Hibrand-Saint Oyant L., Hamama L., Sakr S., Nybom H., Baudino S., Caissard J.P., Byrne D.M., Smulder J.M.S., Desnoyé B., Debener T., Bruneau A., De Riek J., Matsumoto S., Torres A., Millan T., Amaya I., Yamada K., Wincker P., Zamir D., Gouzy J., Sargent D., Bendahmane M., Raymond O., Vergne P., Dubois A., Just J. Towards the rose genome sequence and its use in research and 145 breeding //VI International Symposium on Rose Research and Cultivation 1064. – 2013. – С. 167-175. Fransz F. P., Alonso-Blanco C., Liharska T.B., Peeters A.J.M., Zabel P., de Jong J.H. High‐resolution physical mapping in Arabidopsis thaliana and tomato by fluorescence in situ hybridization to extended DNA fibres //The Plant Journal. – 1996. – Т. 9. – №. 3. – С. 421-430. Fransz P., Armstrong S., Alonso-Blanco C., Fischer T.C., Torres-Ruiz R.A., Jones G. Cytogenetics for the model system Arabidopsis thaliana //The Plant Journal. – 1998. – Т. 13. – №. 6. – С. 867-876. Friesen N., Pollner S., Bachmann K., Blattner F.R. RAPDs and noncoding chloroplast DNA reveal a single origin of the cultivated Allium fistulosum from A. altaicum (Alliaceae) //American Journal of Botany. – 1999. – Т. 86. – №. 4. – С. 554-562. Friesen N., Fritsch R. M., Blattner F. R. Phylogeny and new intrageneric classification of Allium (Alliaceae) based on nuclear ribosomal DNA ITS sequences //Aliso. – 2006. – Т. 22. – №. 1. – С. 372-95. Fuchs J., Brandes A., Schubert I. Telomere sequence localization and karyotype evolution in higher plants //Plant Systematics and Evolution. – 1995. – Т. 196. – №. 3-4. – С. 227-241. Gadaleta A. , Giancaspro A., Nigro D., Giove S.L., Incerti O., Simeone R., Piarulli L., Colasuonno P., Valè G., Cattivelli L., Blanco A. A new genetic and deletion map of wheat chromosome 5A to detect candidate genes for quantitative traits //Molecular Breeding. – 2014. – Т. 34. – №. 4. – С. 1599-1611. Galván G. A., Koning-Boucoiran C.F.S., Koopman W.J.M., Burger-Meijer K., González P.H., Waalwijk C., Kik C., Scholten O.E. Genetic variation among Fusarium isolates from onion, and resistance to Fusarium basal rot in related Allium species //European Journal of Plant Pathology. – 2008. – Т. 121. – №. 4. – С. 499-512. Gar O., Sargent D.J., Tsai C.-J., Pleban T., Shalev G., Byrne D.H., Zamir D. An autotetraploid linkage map of rose (Rosa hybrida) validated using the strawberry 146 (Fragaria vesca) genome sequence //PLoS One. – 2011. – Т. 6. – №. 5. – С. e20463. Geber G., Schweizer D. Cytochemical heterochromatin differentiation in Sinapis alba (Cruciferae) using a simple air-drying technique for producing chromosome spreads //Plant systematics and evolution. – 1988. – Т. 158. – №. 2-4. – С. 97106. Gerlach W. L., Bedbrook J. R. Cloning and characterization of ribosomal RNA genes from wheat and barley //Nucleic Acids Research. – 1979. – Т. 7. – №. 7. – С. 1869-1885. Gill B. S., Friebe B., Endo T. R. Standard karyotype and nomenclature system for description of chromosome bands and structural aberrations in wheat (Triticum aestivum) //Genome. – 1991. – Т. 34. – №. 5. – С. 830-839. Giorgi D., Farina A., Grosso V., Gennaro A., Ceoloni C., Lucretti S. FISHIS: fluorescence in situ hybridization in suspension and chromosome flow sorting made easy //PloS one. – 2013. – Т. 8. – №. 2. – С. e57994. Goff S. A., Ricke D., Lan T.-H. , Presting G., Wang R., Dunn M., Glazebrook J., Sessions A., Oeller P., Varma H., Hadley D., Hutchison D., Martin C., Katagiri F., Lange B.M., Moughamer T., Xia Y., Budworth P., Zhong J., Migue T., Paszkowski U., Zhang S., Colbert M., Sun W., Chen L., Cooper B., Park S.,Wood T.C., Mao L., Quail P., Wing R., Dean R., Yu Y., Zharkikh A., Shen R., Sahasrabudhe S., Thomas A., Cannings R., Gutin A., Pruss D., Reid J., Tavtigian S., Mitchell J., Eldredge G., Scholl T., Miller R.M., Bhatnagar S., Adey N., Rubano T., Tusneem N., Robinson R., Feldhaus J., Macalma T., Oliphant A., Briggs S. A draft sequence of the rice genome (Oryza sativa L. ssp. japonica) //Science. – 2002. – Т. 296. – №. 5565. – С. 92-100. Gong Z., Wu Y., Koblížková A., Torres G.A., Wang K., Iovene M., Neumann P., Zhang W., Novák P., Buell C.R., Macas J., Jiang J. Repeatless and repeatbased centromeres in potato: implications for centromere evolution //The Plant Cell Online. – 2012. – Т. 24. – №. 9. – С. 3559-3574. 147 Gill K. S., Gill B. S., Endo T. R. A chromosome region-specific mapping strategy reveals gene-rich telomeric ends in wheat //Chromosoma. – 1993. – Т. 102. – №. 6. – С. 374-381. Gill K. S., Gill B.S., Endo T.R., Boyko E.V. Identification and high-density mapping of gene-rich regions in chromosome group 5 of wheat //Genetics. – 1996. – Т. 143. – №. 2. – С. 1001-1012. Gross R. A., Kumar A., Kalra B. Polymer synthesis by in vitro enzyme catalysis //Chemical Reviews. – 2001. – Т. 101. – №. 7. – С. 2097-2124. Gudin S. Rose breeding technologies //III International Symposium on Rose Research and Cultivation 547. – 2000. – С. 23-33. Han Y., Zhang Z., Huang S., Jin W. An integrated molecular cytogenetic map of Cucumis sativus L. chromosome 2 //BMC genetics. – 2011. – Т. 12. – №. 1. – С. 18. Hasterok R., Maluszynska J. Cytogenetic markers of Brassica napus L. chromosomes //Journal of Applied Genetics. – 2000. – Т. 41. – №. 1. – С. 1-10. Havey M. J. Restriction enzyme analysis of the chloroplast and nuclear 45s ribosomal DNA ofAllium sectionsCepa andPhyllodolon (Alliaceae) //Plant systematics and evolution. – 1992. – Т. 183. – №. 1-2. – С. 17-31. Havey M., McCallum J.J., Town C.D., Jakse J., Shigyo M. The potential impact of genomes for Allium crop improvement //XXVII International Horticultural Congress-IHC2006: International Symposium on Cultivation and Utilization of Asian, 770. – 2006. – С. 139-146. He L., Liu J., Torres G.A., Zhang H., Jiang J., Xie C. Interstitial telomeric repeats are enriched in the centromeres of chromosomes in Solanum species //Chromosome research. – 2013. – Т. 21. – №. 1. – С. 5-13. Henegariu O., Heerema N.A., Lowe Wright L., Bray-Ward P., Ward D.C., Vance G.H. Improvements in cytogenetic slide preparation: controlled chromosome spreading, chemical aging and gradual denaturing //Cytometry. – 2001. – Т. 43. – №. 2. – С. 101-109. 148 Hizume M., Shibata F., Matsusaki Y., Garajova Z. Chromosome identification and comparative karyotypic analyses of four Pinus species //Theoretical and Applied Genetics. – 2002. – Т. 105. – №. 4. – С. 491-497. Hliscs R., Mühlig P., Claussen U. The spreading of metaphases is a slow process which leads to a stretching of chromosomes //Cytogenetic and Genome Research. – 1997. – Т. 76. – №. 3-4. – С. 167-171. Hopman A. H. N., Ramaekers F. C. S., Speel E. J. M. Rapid synthesis of biotin-, digoxigenin-, trinitrophenyl-, and fluorochrome-labeled tyramides and their application for In situ hybridization using CARD amplification //Journal of Histochemistry & Cytochemistry. – 1998. – Т. 46. – №. 6. – С. 771-777. Hohmann U., Endo T.R., Gill K.S., Gill B.S. Comparison of genetic and physical maps of group 7 chromosomes from Triticum aestivum L //Molecular and General Genetics MGG. – 1994. – Т. 245. – №. 5. – С. 644-653. Huang S., Li R., Zhang Z., Li L., Gu X. et al. The genome of the cucumber, Cucumis sativus L //Nature genetics. – 2009. – Т. 41. – №. 12. – С. 1275-1281. Huang L., Brooks S.A., Li W., Fellers J.P., Trick H.N., Gill B.S. Huang L. Map-based cloning of leaf rust resistance gene Lr21 from the large and polyploid genome of bread wheat //Genetics. – 2003. – Т. 164. – №. 2. – С. 655-664. Huang X., Wang J., Liu H., Lan T., Ren J. Quantum dot-based FRET for sensitive determination of hydrogen peroxide and glucose using tyramide reaction //Talanta. – 2013. – Т. 106. – С. 79-84. Hurst, C.C. 1925. Chromosomes and characters in Rosa and their significance in the origin of species. Experiments in Genetics 37: 534-550. Hurst, C.C. 1927. Differential polyploidy in the genus Rosa L. Verhandiungen des V.internationalen Kongresses fur Vererbungswissenschaft. 867-906. Iacia A. A. S., Pinto-Maglio C. A. F. Mapping pachytene chromosomes of coffee using a modified protocol for fluorescence in situ hybridization //AoB plants. – 2013. – Т. 5. – С. plt040. 149 Islam-Faridi M. N., Childs K. L., Klein P. E., Hodnett G., Menz M.A., Klein R.R., Rooney W.L., Mullet J.E., Stelly D.M., Pric H.J. A molecular cytogenetic map of sorghum chromosome 1: fluorescence in situ hybridization analysis with mapped bacterial artificial chromosomes //Genetics. – 2002. – Т. 161. – №. 1. – С. 345-353. Jaillon O. , Aury J.M., Noel B. , Policriti A. , Clepet C. et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla //Nature. – 2007. – Т. 449. – №. 7161. – С. 463-467. Jian H., Zhang H., Tang K., Li S., Wang Q., Zhang T., Qi, X., Yan H. Decaploidy in Rosa praelucens Byhouwer (Rosaceae) endemic to zhongdian plateau, Yunnan, China //Caryologia. – 2010. – Т. 63. – №. 2. – С. 162-167. Jian H.Y., Min T., Ting Z., Li S.B., Zhang H., Tang K.X. Chromosome variation from sect. Chinenses (Rosa L.) through Chinese old garden roses to modern rose cultivars //International Conference on Germplasm of Ornamentals 977. – 2012. – С. 157-166. Jian H.Y., Zhang T., Wang Q.G., Li S.B., Zhang H., Tang K.X. Karyological diversity of wild Rosa in Yunnan, southwestern China //Genetic resources and crop evolution. – 2013. – Т. 60. – №. 1. – С. 115-127. Jiang J., Gill B. S. Current status and the future of fluorescence in situ hybridization (FISH) in plant genome research //Genome. – 2006. – Т. 49. – №. 9. – С. 1057-1068. Jiang J., Gill B.S., Wangt G.-L., Ronald P.C., Wardt D.C. Metaphase and interphase fluorescence in situ hybridization mapping of the rice genome with bacterial artificial chromosomes //Proceedings of the National Academy of Sciences. – 1995. – Т. 92. – №. 10. – С. 4487-4491. Jones H. A., Mann L.K. Onions and their allies //Soil Science. – 1964. – Т. 98. – №. 1. – С. 68. Jung S., Staton M., Lee T., Blenda A., Syancara R. GDR (Genome Database for Rosaceae): integrated web-database for Rosaceae genomics and genetics data //Nucleic acids research. – 2008. – Т. 36. – №. suppl 1. – С. D1034-D1040. 150 Kalavacharla V., Hossain K., Gu Y., Riera-Lizarazu O., Vales M.I., Bhamidimarri S., Gonzalez-Hernandez J.L. , Maan S.S., Kianian S.F. High-resolution radiation hybrid map of wheat chromosome 1D //Genetics. –2006. – Т. 173. – №. 2. – С. 1089-1099. Kang W.H., Hoang N.H., Yang H.B., Kwon J.K., Jo S.H., Seo J.K., Kim K.H., Choi D., Kang .BC. Molecular mapping and characterization of a single dominant gene controlling CMV resistance in peppers (Capsicum annuum L.) //Theoretical and applied genetics. – 2010. – Т. 120. – №. 8. – С. 1587-1596. Kaczmarek A., Naganowska B., Wolko B. Prins and C-PRINS: Promising tools for the physical mapping of the lupin genome //Cellular and Molecular Biology Letters. – 2007. – Т. 12. – №. 1. – С. 16-24. Karp A. Preparation of chromosome spreads by root-tip meristem dissection for in situ hybridization with biotin-labeled probes //Protocols for Nucleic Acid Analysis by Nonradioactive Probes. – Humana Press, 1994. – С. 149-151. Kato A. An improved method for chromosome counting in maize //Biotechnic & histochemistry. – 1997. – Т. 72. – №. 5. – С. 249-252. Kato A. Air drying method using nitrous oxide for chromosome counting in maize //Biotechnic & histochemistry. – 1999. – Т. 74. – №. 3. – С. 160-166. Kato A., Kato A., Albert P.S., Vega J.M., Kato A., Albert P.S., Vega J.M., Birchler J.A. Sensitive fluorescence in situ hybridization signal detection in maize using directly labeled probes produced by high concentration DNA polymerase nick translation //Biotechnic & Histochemistry. – 2006. – Т. 81. – №. 2-3. – С. 71-78. Kato A., Lamb J. C., Birchler J. A. Chromosome painting using repetitive DNA sequences as probes for somatic chromosome identification in maize //Proceedings of the National Academy of Sciences of the United States of America. – 2004. – Т. 101. – №. 37. – С. 13554-13559. Kato A., Vega J.M., Han F., Lamb J.C., Birchler J.A. Advances in plant chromosome identification and cytogenetic techniques //Current opinion in plant biology. – 2005. – Т. 8. – №. 2. – С. 148-154. 151 Kerstens H. M., Poddighe P. J., Hanselaar A. G. A novel in situ hybridization signal amplification method based on the deposition of biotinylated tyramine //Journal of Histochemistry & Cytochemistry. – 1995. – Т. 43. – №. 4. – С. 347-352. Karafiátová M., Bartoš J., Kopecký D., Ma L., Sato K., Houben A., Stein N., Doležel J. Mapping nonrecombining regions in barley using multicolor FISH //Chromosome Research. – 2013. – Т. 21. – №. 8. – С. 739-751. Khrustaleva L. I., de Melo P.E., van Heusden A.W., Kik C. The integration of recombination and physical maps in a large-genome monocot using haploid genome analysis in a trihybrid Allium population //Genetics. – 2005. – Т. 169. – №. 3. – С. 1673-1685. Khrustaleva L.I., Kik C. Localization of single‐copy T‐DNA insertion in transgenic shallots (Allium cepa) by using ultra‐sensitive FISH with tyramide signal amplification //The Plant Journal. – 2001. – Т. 25. – №. 6. – С. 699-707. Khrustaleva L., Kirov I., Romanov D., Budylin M., Lapitskaya I., Kiseleva A., Fesenko I., Karlov G. The chromosome organization of genes and some types of extragenic DNA in Allium //Acta Hort. – 2012. – Т. 969. – С. 43-51. King J. J., Bradeen J. M., Bark O., McCallum J. A., Havey M. J. A low-density genetic map of onion reveals a role for tandem duplication in the evolution of an extremely large diploid genome //Theoretical and Applied Genetics. – 1998. – Т. 96. – №. 1. – С. 52-62. Kirov I., Van Laere K. , De Riek J., De Keyser E., Van Roy N., Khrustaleva L. Anchoring linkage groups of the Rosa genetic map to physical chromosomes with Tyramide-FISH and EST-SNP markers //PloS one. – 2014a. – Т. 9. – №. 4. – С. e95793. Kirov I., Divashuk M., Van Laere K., Soloviev A., Khrustaleva L. An easy ―SteamDrop‖ method for high quality plant chromosome preparation //Molecular cytogenetics. – 2014b. – Т. 7. – №. 1. – С. 21. Kirov I. et al. High Resolution Physical Mapping of single gene fragments on pachytene chromosome 4 and 7 of Rosa. BMC Genetics– 2015a – Т. 16. – №. 1. – С. 74. 152 Kirov I. V. et al. Molecular Cytogenetics in the Genus Rosa: Current Status and Future Perspectives //Acta horticulturae. -1087. – 2015b. – С. 41-48. Kiseleva A. V., Kirov I. V., Khrustaleva L. I. Chromosomal organization of centromeric Ty3/gypsy retrotransposons in Allium cepa L. and Allium fistulosum L //Russian journal of genetics. – 2014. – Т. 50. – №. 6. – С. 586-592. Kobayashi S., Higashimura H. Oxidative polymerization of phenols revisited //Progress in Polymer Science. – 2003. – Т. 28. – №. 6. – С. 1015-1048. Kobayashi F. et al. A high-resolution physical map integrating an anchored chromosome with the BAC physical maps of wheat chromosome 6B //BMC genomics. – 2015. – Т. 16. – №. 1. – С. 595. Koch J.E., Kolvraa S., Petyersen K.B., Gregersen N., Bolund L. Oligonucleotidepriming methods for the chromosome-specific labelling of alpha satellite DNA in situ //Chromosoma. – 1989. – Т. 98. – №. 4. – С. 259-265. Komuro S., Endo R., Shikata K., Kato A. Genomic and chromosomal distribution patterns of various repeated DNA sequences in wheat revealed by a fluorescence in situ hybridization procedure //Genome. – 2013. – Т. 56. – №. 3. – С. 131-137. Koo D. H., Choi H.-W., Cho J., Hur Y., Bang J.-W. A high-resolution karyotype of cucumber (Cucumis sativus L.'Winter Long') revealed by C-banding, pachytene analysis, and RAPD-aided fluorescence in situ hybridization //Genome. – 2005. – Т. 48. – №. 3. – С. 534-540. Koumbaris G.L., Bass H.W. A new single‐locus cytogenetic mapping system for maize (Zea mays L.): overcoming FISH detection limits with marker‐selected sorghum (S. propinquum L.) BAC clones //The Plant Journal. – 2003. – Т. 35. – №. 5. – С. 647-659. Kubalakova M., Macas, J. and Dolezel, J.: Mapping of repeated DNA sequences in plant chromosomes by PRINS and C-PRINS //Theoretical and applied genetics. – 1997. – Т. 94. – №. 6-7. – С. 758-763. 153 Kubaláková M., Vrána J., Cíhalíková J., Lysák M.A., Dolezel J. Localisation of DNA sequences on plant chromosomes using PRINS and C-PRINS //Chromosome Painting. – Springer Netherlands, 2001. – С. 71-82. Kulikova O., Gualtieri G., Geurts R., Kim D.J., Cook D., Huguet T., Jong J.H. de, Fransz P.F., Bisseling T. Integration of the FISH pachytene and genetic maps of Medicago truncatula //The Plant Journal. – 2001. – Т. 27. – №. 1. – С. 49-58. Kumar A., Bassin F.M., de Jimenez M.K.M., Ghavami F., Mazaheri M., Simons K., Iqbal M.J., Mergoum M., Kianian S.F., Kianian P.M.A.Radiation Hybrids: A valuable Tool for Genetic, Genomic and Functional Analysis of Plant Genomes //Genomics of Plant Genetic Resources. – Springer Netherlands, 2014. – С. 285318. Künzel G., Korzun L., Meister A. Cytologically integrated physical restriction fragment length polymorphism maps for the barley genome based on translocation breakpoints //Genetics. – 2000. – Т. 154. – №. 1. – С. 397-412. Lamb J. C., Danilova T. , Bauer M.J., Meyer J.M., Holland J.J., Jensen M.D., Birchler J.A. Single-gene detection and karyotyping using small-target fluorescence in situ hybridization on maize somatic chromosomes //Genetics. – 2007. – Т. 175. – №. 3. – С. 1047-1058. Laskowska D., Berbeć A., Van Laere K., Kirov I., Czubacka A., Trojak-Goluch A. Cytology and fertility of amphidiploid hybrids between Nicotiana wuttkei Clarkson et Symon and N. tabacum L //Euphytica. – С. 1-12. Lavania U. C., Yamamoto M., Mukai Y. Extended chromatin and DNA fibers from active plant nuclei for high-resolution FISH //Journal of Histochemistry & Cytochemistry. – 2003. – Т. 51. – №. 10. – С. 1249-1253. Lengerova M., Kejnovsky E., Hobza R., Macas J., Grant S. R., Vyskot B. Multicolor FISH mapping of the dioecious model plant, Silene latifolia //Theoretical and applied genetics. – 2004. – Т. 108. – №. 7. – С. 1193-1199. 154 Leus L., Moghaddam H., Van Huylenbroeck J. Pathotype-Specific Powdery Mildew Resistance in Roses //V International Symposium on Rose Research and Cultivation 870. – 2009. – С. 99-102. Levan A. The cytology of the species hybrid Allium cepa× fistulosum and its polyploid derivatives //Hereditas. – 1941. – Т. 27. – №. 3‐4. – С. 253-272. Li F., Fan G., Wang K., Sun F., Yuan Y., Song G. et al.. Genome sequence of the cultivated cotton Gossypium arboreum //Nature genetics. – 2014. – Т. 46. – №. 6. – С. 567-572. Lim K. Y., Werlemark G., Matyasek R., Bringloe J. B., Sieber V., El Mokadem H., Roberts A. V. Evolutionary implications of permanent odd polyploidy in the stable sexual, pentaploid of Rosa canina L //Heredity. – 2005. – Т. 94. – №. 5. – С. 501-506. Ling H. Q., , Zhao S., Liu D., Wang J., Sun H. et al.. Draft genome of the wheat Agenome progenitor Triticum urartu //Nature. – 2013. – Т. 496. – №. 7443. – С. 87-90. Liu D.H., Li M.X. 1985. A study on karyotypes of some flowers of Rosa in China. J Wuhan Bot Res (Wuhan zhiwuxue Yanjou) 3: 403–408. Liu C.Y., Wang G.L., Xie Q.L., Jin J., Liu G.N. An study on the chromosome karyomorphology of 6 species in Rosa [J] //Journal of Jiangsu Forestry Science & Technology. – 2008. – Т. 6. – С. 001. Lou Q., He Y., Cheng C., Zhang Z., Li J., Huang S., Chen J. Integration of highresolution physical and genetic map reveals differential recombination frequency between chromosomes and the genome assembling quality in cucumber //PloS one. – 2013. – Т. 8. – №. 5. – С. e62676. Ma Y., Islam-Faridi M.N., Crane C.F., Stelly D.M., Price H.J., Byrne D.H. A new procedure to prepare slides of metaphase chromosomes of roses //HortScience. – 1996. – Т. 31. – №. 5. – С. 855-857. 155 Ma L., Vu G.T.H., Schubert V., Watanabe K., Stein N., Houben A., Schubert I. Synteny between Brachypodium distachyon and Hordeum vulgare as revealed by FISH //Chromosome research. – 2010. – Т. 18. – №. 7. – С. 841-850. Ma Y., Chen J. Y. Chromosome studies of seven roses //J Fujian Coll For. – 1991. – Т. 11. – С. 215-218. Ma Y., Chen J. Y. Chromosome studies of six species of Rosa in China //Guihaia. – 1992. – Т. 12. – С. 333-336. Ma Y., Crane C. F., Byrne D. H. Karyotypic relationships among some Rosa species //Caryologia. – 1997. – Т. 50. – №. 3-4. – С. 317-326. Ma Y., Islam-Faridi M. N., Crane C. F., Ji Y., Stelly D. M., Price H. J., Byrne, D. H. In situ hybridization of ribosomal DNA to rose chromosomes //Journal of Heredity. – 1997. – Т. 88. – №. 2. – С. 158-161. Macas J., Navrátilová A., Mészáros T. Sequence subfamilies of satellite repeats related to rDNA intergenic spacer are differentially amplified on Vicia sativa chromosomes //Chromosoma. – 2003. – Т. 112. – №. 3. – С. 152-158. Maeda T. Chiasma studies in Allium fistulosum, Allium cepa, and their F1, F2 and backcross hybrids //遺伝學雑誌. – 1937. – Т. 13. – №. 3. – С. 146-159. Mandáková T., Joly S., Krzywinski M., Mummenhoff K., Lysak M.A. Fast diploidization in close mesopolyploid relatives of Arabidopsis //The Plant Cell Online. – 2010. – Т. 22. – №. 7. – С. 2277-2290. Marks G. E. A rapid HCl/toluidine blue squash technic for plant chromosomes //Biotechnic & Histochemistry. – 1973. – Т. 48. – №. 5. – С. 229-231. Martin R., Busch W., Herrmann R.G., Wanner G. Efficient preparation of plant chromosomes for high-resolution scanning electron microscopy //Chromosome Research. – 1994. – Т. 2. – №. 5. – С. 411-415. Masamura N., McCallum J., Khrustaleva L., Kenel F., Pither-Joyce M., Shono J., Suzuki G., Mukai Y., Yamauchi N.,Shigyo M. Chromosomal Organization and Sequence Diversity of Genes Encoding Lachrymatory Factor Synthase in Allium cepa L //G3: Genes| Genomes| Genetics. – 2012. – Т. 2. – №. 6. – С. 643-651. 156 Masuzaki S., Shigyo M., Yamauchi N. Complete assignment of structural genes involved in flavonoid biosynthesis influencing bulb color to individual chromosomes of the shallot (Allium cepa L.) //Genes & genetic systems. – 2006. – Т. 81. – №. 4. – С. 255-263. Mayer K.F., Martis M., Hedley P.E., Simková H., Liu H. et al. Unlocking the barley genome by chromosomal and comparative genomics //The Plant Cell Online. – 2011. – Т. 23. – №. 4. – С. 1249-1263. Martin W.J., McCallum J., Shigyo M., Jakse J., Kuhl J.C., Yamane N., Pither-Joyce M., Gokce A.F., Sink K.C., Town C.D.,Havey M.J. Genetic mapping of expressed sequences in onion and in silico comparisons with rice show scant colinearity //Molecular Genetics and Genomics. – 2005. – Т. 274. – №. 3. – С. 197-204. Menke M., Fuchs J., Schubert I. A comparison of sequence resolution on plant chromosomes: PRINS versus FISH //Theoretical and applied genetics. – 1998. – Т. 97. – №. 8. – С. 1314-1320. Mickelson-Young L., Endo T. R., Gill B. S. A cytogenetic ladder-map of the wheat homoeologous group-4 chromosomes //Theoretical and Applied Genetics. – 1995. – Т. 90. – №. 7-8. – С. 1007-1011. Mirzaghaderi G. A simple metaphase chromosome preparation from meristematic root tip cells of wheat for karyotyping or in situ hybridization //African Journal of Biotechnology. – 2010. – Т. 9. – №. 3. Mlinarec J., Chester M., Siljak-Yakovlev S., Papes D., Leitch A.R., Besendorfer V. Molecular structure and chromosome distribution of three repetitive DNA families in Anemone hortensis L.(Ranunculaceae) //Chromosome research. – 2009. – Т. 17. – №. 3. – С. 331-346. Moghaddam H. H., Leus L., Riek J., Huylenbroeck J., Bockstaele E. Construction of a genetic linkage map with SSR, AFLP and morphological markers to locate QTLs controlling pathotype-specific powdery mildew resistance in diploid roses //Euphytica. – 2012. – Т. 184. – №. 3. – С. 413-427. 157 Mohan M., Nair S., Bhagwat A., Krishna T.G., Yano M. Genome mapping, molecular markers and marker-assisted selection in crop plants //Molecular breeding. – 1997. – Т. 3. – №. 2. – С. 87-103 Mouras A., Salesses G., Lutz A. Sur l'utilisation des protoplastes en cytologie: amélioration d'une méthode récente en vue de l'identification des chromosomes mitotiques des genres Nicotiana et Prunus //Caryologia. – 1978. – Т. 31. – №. 1. – С. 117-127. Murata M. Staining air dried protoplasts for study of plant chromosomes //Biotechnic & Histochemistry. – 1983. – Т. 58. – №. 2. – С. 101-106. Muravenko O.V., Yurkevich O.Y., Bolsheva N.L., Samatadze T.E., Nosova I.V., Zelenina D.A., Volkov A.A., Popov K.V.,Zelenin A.V. Comparison of genomes of eight species of sections Linum and Adenolinum from the genus Linum based on chromosome banding, molecular markers and RAPD analysis //Genetica. – 2009. – Т. 135. – №. 2. – С. 245-255. Nakao S., Matsunaga S., Sakai A., Kuroiwa T., Kawano S. RAPD isolation of a Y chromosome specific ORF in a dioecious plant, Silene latifolia //Genome. – 2002. – Т. 45. – №. 2. – С. 413-420. Navratilova A., Neumann P., MACAS J. Karyotype analysis of four Vicia species using in situ hybridization with repetitive sequences //Annals of botany. – 2003. – Т. 91. – №. 7. – С. 921-926. Novák P., Neumann P., Macas J. Graph-based clustering and characterization of repetitive sequences in next-generation sequencing data //BMC bioinformatics. – 2010. – Т. 11. – №. 1. – С. 378. Novák P., Neumann .P, Pech J., Steinhaisl J., Macas J. RepeatExplorer: a Galaxy-based web server for genome-wide characterization of eukaryotic repetitive elements from next-generation sequence reads //Bioinformatics. – 2013. – Т. 29. – №. 6. – С. 792-793. 158 Ohmido N., Ishimaru A., Kato .S, Sato S., Tabata .S, Fukui K. Integration of cytogenetic and genetic linkage maps of Lotus japonicus, a model plant for legumes //Chromosome research. – 2010. – Т. 18. – №. 2. – С. 287-299. Ohara T., Song Y.-S., Tsukazaki H., Wako T., Nunome T., Kojima A.Genetic mapping of AFLP markers in Japanese bunching onion (Allium fistulosum) //Euphytica. – 2005. – Т. 144. – №. 3. – С. 255-263. Olin-Fatih M., Heneen W. K. C-banded karyotypes of Brassica campestris, B. oleracea, and B. napus //Genome. – 1992. – Т. 35. – №. 4. – С. 583-589. Ohmido N., Akiyama Y., Fukui K. Physical mapping of unique nucleotide sequences on identified rice chromosomes //Plant molecular biology. – 1998. – Т. 38. – №. 6. – С. 1043-1052. Paesold S., Borchardt D., Schmidt T., Dechyeva D. A sugar beet (Beta vulgaris L.) reference FISH karyotype for chromosome and chromosome‐arm identification, integration of genetic linkage groups and analysis of major repeat family distribution //The Plant Journal. – 2012. – Т. 72. – №. 4. – С. 600-611. Pal A., Dhar T. K. An analytical device for on-site immunoassay. Demonstration of its applicability in semiquantitative detection of aflatoxin B1 in a batch of samples with ultrahigh sensitivity //Analytical chemistry. – 2004. – Т. 76. – №. 1. – С. 98104. Paterson A.H., Bowers J.E., Burow M.D., Draye X., Elsik C.G., Jiang C.-X., Katsar C.S., Lan T.H., Lin Y.R., Ming R., Wright R.J. Comparative genomics of plant chromosomes //The Plant Cell. – 2000. – Т. 12. – №. 9. – С. 1523-1539. Paterson A.H., Bowers J.E., Bruggmann R., Dubchak I., Grimwood J. et al. The Sorghum bicolor genome and the diversification of grasses //Nature. – 2009. – Т. 457. – №. 7229. – С. 551-556. Pedrosa A., Sandal N., Stougaard J., Schweizer D., Bachmair A. Chromosomal map of the model legume Lotus japonicus //Genetics. – 2002. – Т. 161. – №. 4. – С. 1661-1672. 159 Peffley, E.B. (1986). Evidence for chromosomal differentiation of A. fistulosum and A. cepa. J Am Soc Hort Sci, Vol. 111, pp. 126-129 Pérez R., de Bustos A,. Jouve N., Cuadrado Á. Localization of Rad50, a single-copy gene, on group 5 chromosomes of wheat, using a FISH protocol employing tyramide for signal amplification (Tyr-FISH) //Cytogenet Genome Res. – 2009. – Т. 125. – С. 321-328. Pernthaler A., Amann R. Simultaneous fluorescence in situ hybridization of mRNA and rRNA in environmental bacteria //Applied and Environmental Microbiology. – 2004. – Т. 70. – №. 9. – С. 5426-5433. Pinkel D., Landegent J., Collins C., Fuscoe J., Segraves R., Lucas J., Gray J. Fluorescence in situ hybridization with human chromosome-specific libraries: detection of trisomy 21 and translocations of chromosome 4 //Proceedings of the National Academy of Sciences. – 1988. – Т. 85. – №. 23. – С. 9138-9142. Poczai P., Varga I., Laos M., Cseh A., Bell N., Valkonen J.P., Hyvönen J. Advances in plant gene-targeted and functional markers: a review //Plant Methods. – 2013. – Т. 9. – №. 1. – С. 6. Poursarebani N., Nussbaumer T., Simková H., Safář J., Witsenboer H., van Oeveren J., Doležel J., Mayer K.F., Stein N., Schnurbusch T. Whole‐genome profiling and shotgun sequencing delivers an anchored, gene‐decorated, physical map assembly of bread wheat chromosome 6A //The Plant Journal. – 2014. – Т. 79. – №. 2. – С. 334-347. Porter D. R., Jones H. A. Resistance of some of the cultivated species of Allium to pink root (Phoma terrestris) //Phytopathology. – 1933. – Т. 23. – С. 290-298. Potato Genome Sequencing Consortium et al. Genome sequence and analysis of the tuber crop potato //Nature. – 2011. – Т. 475. – №. 7355. – С. 189-195. Price L., Short K. C., Roberts A. V. Poor resolution of C-bands and the presence of Bchromosomes in Rosa rugosa ‗Scabrosa‘ //Caryologia. – 1981. – Т. 34. – №. 1. – С. 69-72. 160 Primer доступа: 3Plus-режим http://www.bioinformatics.nl/cgi- bin/primer3plus/primer3plus.cgi Raap A.K., van de Corput M.P., Vervenne R.A., van Gijlswijk R.P., Tanke H.J., Wiegant J. Ultra-sensitive FISH using peroxidase-mediated deposition of biotin-or fluorochrome tyramides //Human Molecular Genetics. – 1995. – Т. 4. – №. 4. – С. 529-534. Rattanachaikunsopon P., Phumkhachorn P. Diallyl sulfide content and antimicrobial activity against food-borne pathogenic bacteria of chives (Allium schoenoprasum) //Bioscience, biotechnology, and biochemistry. – 2008. – Т. 72. – №. 11. – С. 2987-2991. Rayburn A. L., Gold J. R. A procedure for obtaining mitotic chromosomes from maize //Maydica (Italy). – 1982. Razavi F., De Keyser E., De Riek J., Van Labeke M.C. A method for testing drought tolerance in Fragaria based on fast screening for water deficit response and use of associated AFLP and EST candidate gene markers //Euphytica. – 2011. – Т. 180. – №. 3. – С. 385-409. Reeves A. MicroMeasure: a new computer program for the collection and analysis of cytogenetic data //Genome. – 2001. – Т. 44. – №. 3. – С. 439-443. Reynders-Aloisi S., Bollereau P. Characterisation of genetic diversity in genus Rosa by randomly amplified polymorphic DNA //II International Rose Symposium 424. – 1995. – С. 253-260. Rice Chromosome 10 Sequencing Consortium et al. In-depth view of structure, activity, and evolution of rice chromosome 10 //Science. – 2003. – Т. 300. – №. 5625. – С. 1566-1569. Ricroch A., Peffley E. B., Baker R. J. Chromosomal location of rDNA in Allium: in situ hybridization using biotin-and fluorescein-labelled probe //Theoretical and Applied Genetics. – 1992. – Т. 83. – №. 4. – С. 413-418. 161 Riera-Lizarazu O., Vales M.I., Ananiev E.V., Rines H.W., Phillips R.L. Production and characterization of maize chromosome 9 radiation hybrids derived from an oatmaize addition line //Genetics. – 2000. – Т. 156. – №. 1. – С. 327-339. Roberts A. V., Gladis T., Brumme H. DNA amounts of roses (Rosa L.) and their use in attributing ploidy levels //Plant cell reports. – 2009. – Т. 28. – №. 1. – С. 61-71. Rogers S. O., Bendich A. J. Extraction of DNA from milligram amounts of fresh, herbarium and mummified plant tissues //Plant molecular biology. – 1985. – Т. 5. – №. 2. – С. 69-76. Romanov D. et al. Tyramide-FISH mapping of single genes for development of an integrated recombination and cytogenetic map of chromosome 5 of Allium cepa //Genome. – 2015. – Т. 58. – №. 3. – С. 111-119. Sanz M.J., Loarce Y., Ferrer E., Fominaya A. Use of tyramide-fluorescence in situ hybridization and chromosome microdissection for ascertaining homology relationships and chromosome linkage group associations in oats //Cytogenetic and genome research. – 2011. – Т. 136. – №. 2. – С. 145-156. Sapkota S. K. Physical and genetic mapping of the ASGR-carrier chromosome in Pennisetum squamulatum : Master thesis. – uga, 2014. Schmidt B.F., Chao J., Zhu Z., DeBiasio R.L., Fisher G. Signal amplification in the detection of single-copy DNA and RNA by enzyme-catalyzed deposition (CARD) of the novel fluorescent reporter substrate Cy3. 29-tyramide //Journal of Histochemistry & Cytochemistry. – 1997. – Т. 45. – №. 3. – С. 365-373. Schmidt R., West J., Love K., Lenehan Z., Lister C. Physical map and organization of Arabidopsis thaliana chromosome 4 //Science. – 1995. – Т. 270. – №. 5235. – С. 480-483. Schmidt T., Heslop-Harrison J. S. Genomes, genes and junk: the large-scale organization of plant chromosomes //Trends in Plant Science. – 1998. – Т. 3. – №. 5. – С. 195-199. 162 Schmutzer T. et al. Kmasker-a tool for in silico prediction of single-copy FISH probes for the large-genome species Hordeum vulgare //Cytogenetic and genome research. – 2014. – Т. 142. – №. 1. – С. 66-78. Schnable P.S., Ware D., Fulton R.S., Stein J.C., Wei F., et al: The B73 maize genome: complexity, diversity, and dynamics //science. – 2009. – Т. 326. – №. 5956. – С. 1112-1115. Schriml L.M., Padilla-Nash H.M., Coleman A., Moen P., Nash W.G., Menninger J., Jones G., Ried T., Dean M. Tyramide Signal Amplification (TSA)--FISH Applied to Mapping PCR-Labeled Probes //Biotechniques. – 1999. – Т. 27. – С. 613. Schubert I., Wobus U. In situ hybridization confirms jumping nucleolus organizing regions in Allium //Chromosoma. – 1985. – Т. 92. – №. 2. – С. 143-148. Schubert I. Mobile nucleolus organizing regions (NORs) inAllium (Liliaceae s. lat.)?— Inferences from the specifity of silver staining //Plant systematics and evolution. – 1984. – Т. 144. – №. 3-4. – С. 291-305. Schwarzacher T , Leitch A. R., Bennett M. D, Heslop-Harrison J. S. In situ localization of parental genomes in a wide hybrid //Annals of Botany. – 1989. – Т. 64. – №. 3. – С. 315-324. Schwarzacher T., Leitch A. R. Enzymatic treatment of plant material to spread chromosomes for in situ hybridization //Protocols for Nucleic Acid Analysis by Nonradioactive Probes. – Humana Press, 1994. – С. 153-160. Sears E. R. et al. Aneuploids of common wheat. – 1954. Sears E. R. et al. The transfer of leaf-rust resistance from Aegilops umbellulata to wheat //Genetics in plant breeding. Brook-haven Symposia in Biology 1956. – 1956. – С. 1-22. Sears E. R. Nullisomic-tetrasomic combinations in hexaploid wheat //Chromosome manipulations and plant genetics. – Springer US, 1966. – С. 29-45. 163 Shulaev V., Sargent D.J., Crowhurst R.N., Mockler T.C., Folkerts O. et al: The genome of woodland strawberry (Fragaria vesca) //Nature genetics. – 2011. – Т. 43. – №. 2. – С. 109-116. She C.W., Jiang X.H., Ou L.J., Liu J., Long K.L., Zhang L.H., Duan W.T., Zhao W., Hu J.C. Molecular cytogenetic characterisation and phylogenetic analysis of the seven cultivated Vigna species (Fabaceae) //Plant Biology. – 2015. – Т. 17. – №. 1. – С. 268-280. Shearer L.A., Anderson L.K., de Jong H., Smit S., Goicoechea J.L., Roe B.A., Hua A., Giovannoni J.J., Stack S.M. Fluorescence in situ hybridization and optical mapping to correct scaffold arrangement in the tomato genome //G3: Genes| Genomes| Genetics. – 2014. – Т. 4. – №. 8. – С. 1395-1405. Shibata F., Hizume M. Evolution of 5S rDNA units and their chromosomal localization in Allium cepa and Allium schoenoprasum revealed by microdissection and FISH //Theoretical and Applied Genetics. – 2002. – Т. 105. – №. 2-3. – С. 167-172. Shigyo M., Tashiro Y., Miyazaki S. Chromosomal locations of glutamate oxaloacetate transaminase gene loci in Japanese bunching onion (Allium fistulosum L.) and shallot (A. cepa L. Aggregatum group) //Jpn. J. Genet. – 1994. – Т. 69. – С. 417424. Shigyo M., Tashiro Y., Isshiki S., Miyazaki S. Establishment of a series of alien monosomic addition lines of Japanese bunching onion (Allium fistulosum L.) with extra chromosomes from shallot (A. cepa L. Aggregatum group) //Genes and Genetic Systems. – 1996. – Т. 71. – С. 363-372. Shimizu M., Pu Z.J., Kawanabe T., Kitashiba H., Matsumoto S., Ebe Y., Sano M., Funaki T., Fukai E., Fujimoto R.,Okazaki K. Map-based cloning of a candidate gene conferring Fusarium yellows resistance in Brassica oleracea //Theoretical and Applied Genetics. – 2015. – Т. 128. – №. 1. – С. 119-130. Snowdon R. J. Molecular cytogenetic localization and characterization of 5S and 25S rDNA loci for chromosome identification in oilseed rape (Brassica napus L.) //Annals of Botany. – 2000. – Т. 86. – №. 1. – С. 201-204. 164 Song Y. S. Development of microsatellite markers in bunching onion (Allium fistulosum L.) //Breeding science. – 2004. – Т. 54. – №. 4. – С. 361-365. Sousa A., Cusimano N., Renner S. S. Combining FISH and model-based predictions to understand chromosome evolution in Typhonium (Araceae) //Annals of botany. – 2014. – Т. 113. – №. 4. – С. 669-680. Sousa A., Renner S. S. Interstitial telomere‐like repeats in the monocot family Araceae //Botanical Journal of the Linnean Society. – 2015. – Т. 177. – №. 1. – С. 15-26. Spurbeck J.L., Zinsmeister A.R., Meyer K.J., Jalal S.M. Dynamics of chromosome spreading //American journal of medical genetics. – 1996. – Т. 61. – №. 4. – С. 387-393. Stephens J.L., Brown S.E., Lapitan N.L., Knudson D.L. Physical mapping of barley genes using an ultrasensitive fluorescence in situ hybridization technique //Genome. – 2004. – Т. 47. – №. 1. – С. 179-189. Stevenson M., Armstrong S.J., Ford-Lloyd B.V., Jones G.H. Comparative analysis of crossover exchanges and chiasmata in Allium cepa× fistulosum after genomic in situ hybridization (GISH) //Chromosome Research. – 1998. – Т. 6. – №. 7. – С. 567-574. Sturtevant A. The linear arrangement of six sex-linked factors in Drosophila as shown by their mode of association //Molecular and General Genetics MGG. – 1913. – Т. 10. – №. 1. – С. 293-294. Sturtevant A. H. A History of Genetics. – New York, Harper and Row, 1965. Subramanian D. Cytotaxonomic studies of South Indian Rosaceae //Cytologia. – 1987. – Т. 52. – №. 3. – С. 395-403. Sugiyama S., Yoshino T., Kanahara H., Shichiri M., Fukushi D., Ohtani T. Effects of acetic acid treatment on plant chromosome structures analyzed by atomic force microscopy //Analytical biochemistry. – 2004. – Т. 324. – №. 1. – С. 39-44. Sun J.Y., Zhang Z.H., Zong X., Huang S.W., LI Z.Y., Han Y.H. A high-resolution cucumber cytogenetic map integrated with the genome assembly //BMC genomics. – 2013. – Т. 14. – №. 1. – С. 461. 165 Suzuki G., Ura A., Saito N., Do G.S., Seo B.B., Yamamoto M., Mukai Y. BAC FISH analysis in Allium cepa //Genes & genetic systems. – 2001. – Т. 76. – №. 4. – С. 251-255. Tanksley S.D., Ganal M.W., Prince J.P., de Vicente M.C., Bonierbale M.W., Broun P., Fulton T.M., Giovannoni J.J.,Grandillo S., Martin G.B. High density molecular linkage maps of the tomato and potato genomes //Genetics. – 1992. – Т. 132. – №. 4. – С. 1141-1160. Tek A. L., Jiang J. The centromeric regions of potato chromosomes contain megabasesized tandem arrays of telomere-similar sequence //Chromosoma. – 2004. – Т. 113. – №. 2. – С. 77-83. Tomato Genome Consortium et al. The tomato genome sequence provides insights into fleshy fruit evolution //Nature. – 2012. – Т. 485. – №. 7400. – С. 635-641. Tonami H. Peroxidase‐catalyzed oxidative polymerization of m‐substituted phenol derivatives //Macromolecular Chemistry and Physics. – 1999. – Т. 200. – №. 10. – С. 2365-2371. Triebe B., Mukai Y., Dhaliwal H.S., Martin T.J., Gill B.S. Identification of alien chromatin specifying resistance to wheat streak mosaic and greenbug in wheat germ plasm by C-banding and in situ hybridization //Theoretical and applied genetics. – 1991. – Т. 81. – №. 3. – С. 381-389. Tsujimoto H., Mukai Y., Akagawa K., Nagaki K., Fujigaki J., Yamamoto M., Sasakuma T. Identification of individual barley chromosomes based on repetitive sequences: conservative distribution of Afa-family repetitive sequences on the chromosomes of barley and wheat //Genes & genetic systems. – 1997. – Т. 72. – №. 5. – С. 303-309. Tsukazaki H., Yamashita K., Yaguchi S., Masuzaki J., Kanamori H., Kono I., Hang T.T.,Shigyo S., Fukuoka H., Yonemaru M., Kojima A., Wako T. Construction of SSR-based chromosome map in bunching onion (Allium fistulosum) //Theoretical and applied genetics. – 2008. – Т. 117. – №. 8. – С. 1213-1223. 166 Turac E., Sahmetlioglu E. Oxidative polymerization of 4-[(4-phenylazo-phenyimino)methyl]-phenol catalyzed by horseradish peroxidase //Synthetic Metals. – 2010. – Т. 160. – №. 1. – С. 169-172. Uchida W., Matsunaga S., Sugiyama R., Kawano S. Interstitial telomere-like repeats in the Arabidopsis thaliana genome //Genes & genetic systems. – 2002. – Т. 77. – №. 1. – С. 63-67. Valárik M, Bartos J, Kovárová P, Kubaláková M, de Jong J.H., Dolezel J. High‐resolution FISH on super‐stretched flow‐sorted plant chromosomes //The Plant Journal. – 2004. – Т. 37. – №. 6. – С. 940-950. Valárik M., Bartos J., Kovárová P., Kubaláková M., de Jong J.H., Dolezel J. Horseradish peroxidase-labeled oligonucleotides and fluorescent tyramides for rapid detection of chromosome-specific repeat sequences //Cytogenetic and Genome Research. – 1996. – Т. 75. – №. 4. – С. 258-262. Van Heusden A. W. AFLP linkage group assignment to the chromosomes of Allium cepa L. via monosomic addition lines //Theoretical and applied genetics. – 2000. – Т. 100. – №. 3-4. – С. 480-486. Van der Knaap E., Sanyal A., Jackson S. A., Tanksley S. D. , Van der Knaap E. Highresolution fine mapping and fluorescence in situ hybridization analysis of sun, a locus controlling tomato fruit shape, reveals a region of the tomato genome prone to DNA rearrangements //Genetics. – 2004. – Т. 168. – №. 4. – С. 2127-2140. Van der Meer Q. P. Improving the onion crop (Allium cepa L.) by transfer of characters from Allium fistulosum L //Biuletyn warzywniczy.= Bulletin of vegetable crops research work.= Biulleten'po ovoshchevodstvu. – 1978. Van de Rijke F.M., Florijn R.J., Tanke H.J., Raap A.K. DNA fiber-FISH staining mechanism //Journal of Histochemistry & Cytochemistry. – 2000. – Т. 48. – №. 6. – С. 743-745. Vamosi J. C., Dickinson T. A. Polyploidy and diversification: a phylogenetic investigation in Rosaceae //International Journal of Plant Sciences. – 2006. – Т. 167. – №. 2. – С. 349-358. 167 Vischi M., Jurman I., Bianchi G., Morgante M. Karyotype of Norway spruce by multicolor FISH //Theoretical and Applied Genetics. – 2003. – Т. 107. – №. 4. – С. 591-597. Wang C. J. R., Harper L., Cande W. Z. High-resolution single-copy gene fluorescence in situ hybridization and its use in the construction of a cytogenetic map of maize chromosome 9 //The Plant Cell Online. – 2006. – Т. 18. – №. 3. – С. 529-544. Wang K., Guo W., Yang Z., Hu Y., Zhang W., Zhou B., Stelly D.M., Chen Z.J., Zhang T. Structure and size variations between 12A and 12D homoeologous chromosomes based on high-resolution cytogenetic map in allotetraploid cotton //Chromosoma. – 2010. – Т. 119. – №. 3. – С. 255-266. Wang K., Wang Z., Li F., Ye W., Wang J. et al. The draft genome of a diploid cotton Gossypium raimondii //Nature genetics. – 2012. – Т. 44. – №. 10. – С. 10981103. Watabe S., Sakamoto Y., Morikawa M., Okada R., Miura T., Ito E Highly sensitive determination of hydrogen peroxide and glucose by fluorescence correlation spectroscopy //PloS one. – 2011. – Т. 6. – №. 8. – С. e22955. Wai C.M., Moore P.H., Paull R.E., Ming R., Yu Q. An integrated cytogenetic and physical map reveals unevenly distributed recombination spots along the papaya sex chromosomes //Chromosome research. – 2012. – Т. 20. – №. 6. – С. 753-767. Weber D., Helentjaris T. Mapping RFLP loci in maize using BA translocations //Genetics. – 1989. – Т. 121. – №. 3. – С. 583-590. Werner J. E., Endo T. R., Gill B. S. Toward a cytogenetically based physical map of the wheat genome //Proceedings of the National Academy of Sciences. – 1992. – Т. 89. – №. 23. – С. 11307-11311. Wissemann V. Conventional taxonomy of wild roses //Encyclopedia of rose science. – 2003. – Т. 1. – С. 111-117. Wu P., Zhou C., Cheng S., Wu Z., Lu W., Han J. et al. Integrated genome sequence and linkage map of physic nut (Jatropha curcas L.), a biodiesel plant //The Plant Journal. – 2015. 168 Wylie, A.P. 1954. The history of garden roses. Journal of the Royal Horticultural Society (London) 79: 555-571. Xu Z., Kohel R.J., Song G., Cho J., Yu J., Yu S., Tomkins J., Yu J.Z. An integrated genetic and physical map of homoeologous chromosomes 12 and 26 in Upland cotton (G. hirsutum L.) //BMC genomics. – 2008. – Т. 9. – №. 1. – С. 108. Xiao J. Identification of QTLs affecting traits of agronomic importance in a recombinant inbred population derived from a subspecific rice cross //Theoretical and Applied Genetics. – 1996. – Т. 92. – №. 2. – С. 230-244. Xiong Z., Kim J. S., Pires J. C. Integration of genetic, physical, and cytogenetic maps for Brassica rapa chromosome A7 //Cytogenetic and genome research. – 2010. – Т. 129. – №. 1-3. – С. 190-198. Yang L., Koo D.H., Li Y., Zhang X., Luan F., Havey M.J., Jiang J., Weng Y. Chromosome rearrangements during domestication of cucumber as revealed by high-density genetic mapping and draft genome assembly //The Plant Journal. – 2012. – Т. 71. – №. 6. – С. 895-906. Yim Y.S., Davis G.L., Duru N.A., Musket T.A., Linton E.W., Messing J.W. Characterization of three maize bacterial artificial chromosome libraries toward anchoring of the physical map to the genetic map using high-density bacterial artificial chromosome filter hybridization //Plant Physiology. – 2002. – Т. 130. – №. 4. – С. 1686-1696. Yohannes T. Marker-assisted introgression improves Striga resistance in an Eritrean Farmer-Preferred Sorghum Variety //Field Crops Research. – 2015. – Т. 173. – С. 22-29. Yaguchi S., Hang T.T., Tsukazaki H., Hoa V.Q., Masuzaki S., Wako T., Masamura N., Onodera S., Shiomi N.,Yamauchi N., Shigyo M. Molecular and biochemical identification of alien chromosome additions in shallot (Allium cepa L. Aggregatum group) carrying extra chromosome (s) of bunching onion (A. fistulosum L.) //Genes & genetic systems. – 2009. – Т. 84. – №. 1. – С. 43-55. 169 Yamaoka T., Tabata Y., Ikada Y. Fate of water‐soluble polymers administered via different routes //Journal of pharmaceutical sciences. – 1995. – Т. 84. – №. 3. – С. 349-354. Zhang L. Enzymatic polymerization of phenol catalyzed by horseradish peroxidase in aqueous micelle system //European Polymer Journal. – 2012. – Т. 48. – №. 3. – С. 580-585. Zhang P., Li W., Fellers J., Friebe B., Gill B.S. BAC-FISH in wheat identifies chromosome landmarks consisting of different types of transposable elements //Chromosoma. – 2004. – Т. 112. – №. 6. – С. 288-299. Zhang W., Wai C., Ming R., Yu Q., Jiang J. Integration of genetic and cytological maps and development of a pachytene chromosome-based karyotype in papaya //Tropical plant biology. – 2010. – Т. 3. – №. 3. – С. 166-170. Zhang B., Tang D., Goryacheva I.Y., Niessner R., Knopp D. Anodic‐Stripping Voltammetric Immunoassay for Ultrasensitive Detection of Low‐Abundance Proteins Using Quantum Dot Aggregated Hollow Microspheres //Chemistry-A European Journal. – 2013. – Т. 19. – №. 7. – С. 2496-2503. Zhang D., Li P., Zhang Q., Zhang W. Ultrasensitive nanogold probe-based immunochromatographic assay for simultaneous detection of total aflatoxins in peanuts //Biosensors and Bioelectronics. – 2011. – Т. 26. – №. 6. – С. 2877-2882. Zhong X. B., de Jong J. H., Zabel P. Preparation of tomato meiotic pachytene and mitotic metaphase chromosomes suitable for fluorescence in situ hybridization (FISH) //Chromosome Research. – 1996. – Т. 4. – №. 1. – С. 24-28. Zhong X.-B., J. Bodeau, P. F. Fransz, V. M. Williamson, A. van Kammen J. H. de Jong, P. Zabel FISH to meiotic pachytene chromosomes of tomato locates the rootknot nematode resistance gene Mi-1 and the acid phosphatase gene Aps-1 near the junction of euchromatin and pericentromeric heterochromatin of chromosome arms 6S and 6L, respectively //Theoretical and Applied Genetics. – 1999. – Т. 98. – №. 3-4. – С. 365-370. 170 Zhou H., Yang D., Qiu X., Wu X., Li Y. A novel and efficient polymerization of lignosulfonates by horseradish peroxidase/H2O2 incubation //Applied microbiology and biotechnology. – 2013. – Т. 97. – №. 24. – С. 10309-10320. Zhu T., Shi L., Keim P. Detection byin-situ fluorescence of short, single-copy sequences of chromosomal DNA //Plant Molecular Biology Reporter. – 1995. – Т. 13. – №. 3. – С. 270-277.