Описание орнитинкарбамоилтрансферазы (Ornithine carbamoyltransferase) Escherichia coli, strain K12
advertisement

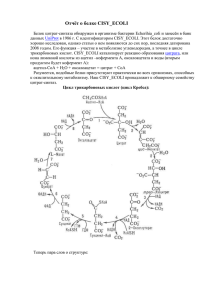
Лапашина Анна Описание орнитинкарбамоилтрансферазы (Ornithine carbamoyltransferase) Escherichia coli, strain K12 Орнитинкарбамоилтрансфераза бактерии Escherichia coli (кишечная палочка) – это фермент, относящийся к классу трансфераз. Альтернативное название этого белка – ОТС-аза-1. Он относится к семейству ОТС-аз/АТС-аз и участвует в биосинтезе аминокислот, а именно аргинина, катализируя реакцию образования L-аргинина из L-орнитина и карбамоилфосфата (стадия 1/3). На рисунке показана эта реакция, в результате которой образуются L-цитруллин и неорганический фосфат: Белок полностью секвенирован. Он является тримером и состоит из трех идентичных или неидентичных друг другу полипептидных цепей. Эти цепи кодируются двумя дуплицированными генами (argI и argF), находящимися в разных локусах. Полипептидные цепи содержат 334 аминокислотных остатка каждая. Но первый остаток (метионин) является инициирующим при синтезе и позже отсекается. Таким образом, фактически каждая цепь состоит из 333 остатков. Молекулярная масса одной цепи равна 36907. Приблизительный объем одной молекулы всего трипептида равен 267946,67 кубических ангстрем. Полная аминокислотная последовательность белка выглядит так: MSGFYHKHFLKLLDFTPAELNSLLQLAAKLKADKKSGKEEAKLTGKNIALIFEKDSTRTRCSFEVAAYDQGARVTYLGPSGSQIGHKESIKDTARV LGRMYDGIQYRGYGQEIVETLAEYASVPVWNGLTNEFHPTQLLADLLTMQEHLPGKAFNEMTLVYAGDARNNMGNSMLEAAALTGLDLRL VAPQACWPEAALVTECRALAQQNGGNITLTEDVAKGVEGADFIYTDVWVSMGEAKEKWAERIALLREYQVNSKMMQLTGNPEVKFLHCL PAFHDDQTTLGKKMAEEFGLHGGMEVTDEVFESAASIVFDQAENRMHTIKAVMVATLSK Рассмотрим участки связывания и важные аминокислотные остатки на примере цепи I: Участки Участок 56-60 Связывание с карбамоилфосфатом Участок 273-276 Связывание с орнитином Сайты Центр связывания 107 Карбамоилфосфат Центр связывания 134 Карбамоилфосфат Сайт 31 Важен для структурной целостности Сайт 147 Важен для структурной целостности Распределение типов вторичной структуры полипептидной цепи I белка показано на рисунке ниже: Голубым цветом выделены альфа-спирали, зеленым – бета-тяжи, желтым – бета-повороты. Неструктурированные участки показаны белым. Как видим, во вторичной структуре белка преобладает альфа-спираль. Орнитинкарбамоилтрансфераза взаимодействует с различными лигандами. Белок контактирует с лигандом MPD, который представляет собой (4S)-2-метил-2,4-пентадиол и имеет брутто-формулу С6Н14О2 . Кроме того, каждая цепь может взаимодействовать с лигандом PSQ (N-дельта-(N'сульфодаминофосфинил)-L-орнитин), брутто-формула которого С5Н15N4O6PS. Это взаимодействие рассматривается в статье «Механизм инактивации орнитинтранскарбамоилазы N-дельта-(N’сульфодиаминофосфинил)-L-орнитином …» (авторы Langley DB, Templeton MD, Fields BA, Mitchell RE, Collyer CA, опубликована в J Biol Chem. 30 июня 2000 г.;275(26):200129, PubMed ID 10747936). Также тример связывается с 735 молекулами воды. Белок синтезируется бактерией Escherichia coli (штамм К12) и находится в цитоплазме. Также в клетке существует орнитинкарбамоилтрансферазный комплекс. Рассмотрим таксономию кишечной палочки. E. coli относится к роду Escherichia, семейству Enterobacteriaceae, порядку Enterobacteriales, классу Gammaproteobacteria, царству Proteobacteria, надцарству Bacteria. Интересно, что в организме человека тоже синтезируется орнитинкарбамоилтрансфераза, которая является митохондриальным белком. Эти ферменты кишечной палочки и человека схожи: имеют похожую структуру (тримеры) и катализируют схожие реакции. Для орнитинкарбамоилтрансферазы описана третичная структура (изображена на рисунке). Разными цветами показаны полипептидные цепи. Ссылки на белок в различных базах данных: База данных UniProt: http://www.uniprot.org/uniprot/P04391 Банк данных PDB: http://www.rcsb.org/pdb/explore.do?structureId=1DUV http://www.rcsb.org/pdb/explore/explore.do?structureId=1AKM http://www.rcsb.org/pdb/explore/explore.do?structureId=2OTC Список статей о белке: 1) Kuo LC, Miller AW, Lee S, Kozuma C., Site-directed mutagenesis of Escherichia coli ornithine transcarbamoylase: role of arginine-57 in substrate binding and catalysis. (Сайт-специфичный мутагенез орнитинтранскарбамоилазы Escherichia coli: роль аргинина-57 в связывании с субстратом и катализе). Подтверждено, что в реакции переноса карбамоила, катализируемой орнитинкарбамоилтрансферазой, остаток аргинина в активном центре фермента Escherishia coli связывается с фосфатным концом субстрата карбамоилфосфата. С применением сайт-специфичного мутагенеза наиболее вероятный из трех кандидатов на центр связывания с карбамоилфосфатом, аргинин57, был замещен на глицин. Получившийся мутантный фермент с глицином-57 абсолютно неэффективен в катализе. При синтезе L-цитруллина из карбамоилфосфата и L-орнитина с образованием неорганического фосфата, производительность мутанта оказалась в 21000 раз ниже, чем у дикого типа. Тем не менее, мутация аргинина-57 всего лишь умеренно влияет на связывание карбамоилфосфата; константа диссоциации этого субстрата, измеренная в устойчивом обновленном состоянии, при мутации возросла с 0,046 до 3,2 mМ. С другой стороны, установлено, что на связывание орнитина значительно влияет изменение константы диссоциации его аналога L-норвалина. Константа диссоциации L-норвалина возрастает примерно в 500 раз от 54 mcМ у дикого типа до 25 mМ у мутанта. Так как предполагается, что аргинин-57 удален от сайта орнитина и аминокислота (как орнитин, так и норвалин) связывается только после карбамоилфосфата в реакции дикого типа, слабое сродство мутанта к норвалину подтверждает, что аргинин-57 участвует в необходимых для продуктивного синтеза аминокислоты. Такое объяснение подтверждается разницей в спектре ультрафиолетового поглощения, которая показывает, что изменения конформации, происходящие у дикого типа при связывании с карбамоилфосфатом , отсутствуют у мутанта. Таким образом, присутствие карбамоилфосфата в активном центре мутанта перестает быть необходимым для связывания с орнитином. В температурной области 5-50 градусов по Цельсию перенос карбамоила и у дикого типа, и у мутанта имеет практически одинаковые энтальпии активации в уравнении Аррениуса, 11 и 10 ккал/моль соответственно. Энтропия активации для реакции дикого типа равна -5,5, а для реакции мутанта -29, учитывая потерю 6-7 ккал/моль на определяющей скорость стадии ферментативной реакции. Biochemistry. 29 ноября 1988;27(24):8823-32. Доступ к статье закрыт, ссылка на аннотацию: http://pubs.acs.org/doi/abs/10.1021/bi00424a021 PubMed ID: 3072022 2) Bencini,D.A., Houghton,J.E., Hoover,T.A., Foltermann,K.F., Wild,J.R., O'Donovan,G.A., The DNA sequence of argI from Escherichia coli K12. (ДНК-секвенирование гена argI Escherichia coli K12). Секвенирован ген argI бактерии E. coli. Он содержит открытую рамку считывания из 1002 оснований, которые кодируют пептид из 334 аминокислот. Три таких полипептида формируют функциональный каталитический триммер (с3) орнитинтранскарбамоилазы (ОТС-азы-1, ЕС 2.1.3.3). Молекулярная масса готового тримера, рассчитанная по аминокислотной последовательности, равна 114,465 дальтонам. Измененная форма argI была получена путем субклонирования фрагмента DdeI, состоящего из 1600 оснований, в сайт HincII плазмиды pUC8, продлевающий открытую рамку считывания на 20 дополнительных нуклеотидов. Ранее было показано, что N-концы сравниваемых полипептидов E. coli argI, argF и pyrB обладают значительной гомологией. По сравнению с этим, гомологичные промотор/оператор argI и argF не проявляют никакой гомологии с pyrB. Тем не менее, ближайшее рассмотрение непосредственно предшествующей аттенюатору pyrBI нуклеотидной последовательности выявило заметное сходство с контрольными участками argI и argF. Nucleic Acids Res. 10 декабря 1983;11(23):8509-18 Доступ к статье закрыт, ссылка на аннотацию: http://nar.oxfordjournals.org/cgi/content/abstract/11/23/8509 PubMed ID: 6369246 3) Burland V, Plunkett G 3rd, Sofia HJ, Daniels DL, Blattner FR Analysis of the Escherichia coli genome VI: DNA sequence of the region from 92.8 through 100 minutes. The 338.5 kb of the Escherichia coli genome described here together with previously described segments bring the total of contiguous finished sequence of this genome to > 1 Mb. Of 319 open reading frames (ORFs) found in this 338.5 kb segment, 147 (46%) are potential new genes. The positions of several genes which had been previously located here by mapping or partial sequencing have been confirmed. Several ORFs have functions suggested by similarities to other characterised genes but cannot be assigned with certainty. Fifteen of the ORFs of unknown function had been previously sequenced. Eight transfer RNAs are encoded in the region and there are two grey holes in which no features were found. The attachment site for phage P4 and three insertion sequences were located. The region was also analysed for chi sites, bend sites, REP elements and other repeats. A computer search identified potential promoters and tentative transcription units were assigned. The occurrence of the rare tetramer CTAG was analysed in 1.6 Mb of contiguous E.coli sequence. Hypotheses addressing the rarity and distribution of CTAG are discussed. Nucleic Acids Res. 25 июня 1995;23(12):2105-19. Доступ к статье закрыт, ссылка на аннотацию: http://nar.oxfordjournals.org/cgi/content/abstract/23/12/2105 PubMed ID: 7610040 4) Blattner FR, Plunkett G 3rd, Bloch CA, Perna NT, Burland V, Riley M, Collado-Vides J, Glasner JD, Rode CK,Mayhew GF, Gregor J, Davis NW, Kirkpatrick HA, Goeden MA, Rose DJ, Mau B, Shao Y. The complete genome sequence of Escherichia coli K-12. The 4,639,221-base pair sequence of Escherichia coli K-12 is presented. Of 4288 protein-coding genes annotated, 38 percent have no attributed function. Comparison with five other sequenced microbes reveals ubiquitous as well as narrowly distributed gene families; many families of similar genes within E. coli are also evident. The largest family of paralogous proteins contains 80 ABC transporters. The genome as a whole is strikingly organized with respect to the local direction of replication; guanines, oligonucleotides possibly related to replication and recombination, and most genes are so oriented. The genome also contains insertion sequence (IS) elements, phage remnants, and many other patches of unusual composition indicating genome plasticity through horizontal transfer. Science. 5 сентября 1997;277(5331):1453-74. Статью возможно открыть в формате PDF: http://www.ebi.ac.uk/~sp/intern/projects/pdf_archive/pdfpumped/3/9278503.pdf PubMed ID: 9278503