Определение эволюционного расстояния между
advertisement
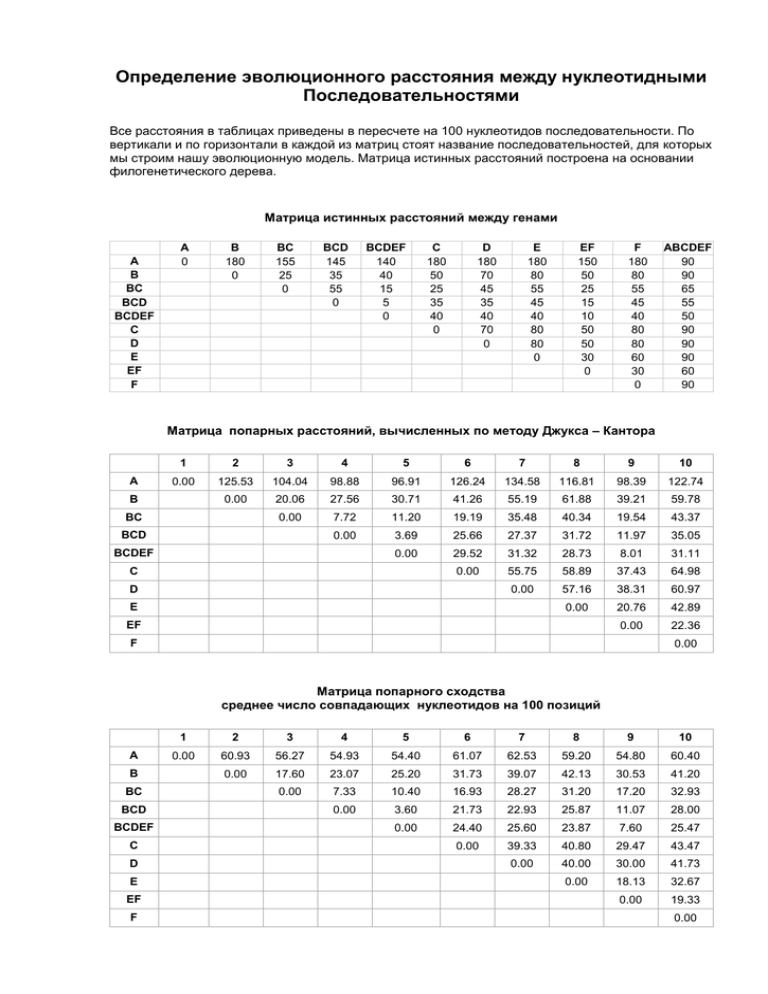
Определение эволюционного расстояния между нуклеотидными Последовательностями Все расстояния в таблицах приведены в пересчете на 100 нуклеотидов последовательности. По вертикали и по горизонтали в каждой из матриц стоят название последовательностей, для которых мы строим нашу эволюционную модель. Матрица истинных расстояний построена на основании филогенетического дерева. Матрица истинных расстояний между генами A B BC BCD BCDEF C D E EF F A 0 B 180 0 BC 155 25 0 BCD 145 35 55 0 BCDEF 140 40 15 5 0 C 180 50 25 35 40 0 D 180 70 45 35 40 70 0 E 180 80 55 45 40 80 80 0 EF 150 50 25 15 10 50 50 30 0 F 180 80 55 45 40 80 80 60 30 0 ABCDEF 90 90 65 55 50 90 90 90 60 90 Матрица попарных расстояний, вычисленных по методу Джукса – Кантора A 1 2 3 4 5 6 7 8 9 10 0.00 125.53 104.04 98.88 96.91 126.24 134.58 116.81 98.39 122.74 0.00 20.06 27.56 30.71 41.26 55.19 61.88 39.21 59.78 0.00 7.72 11.20 19.19 35.48 40.34 19.54 43.37 0.00 3.69 25.66 27.37 31.72 11.97 35.05 0.00 29.52 31.32 28.73 8.01 31.11 0.00 55.75 58.89 37.43 64.98 0.00 57.16 38.31 60.97 0.00 20.76 42.89 0.00 22.36 B BC BCD BCDEF C D E EF F 0.00 Матрица попарного сходства среднее число совпадающих нуклеотидов на 100 позиций A B BC BCD BCDEF C D E EF F 1 2 3 4 5 6 7 8 9 10 0.00 60.93 56.27 54.93 54.40 61.07 62.53 59.20 54.80 60.40 0.00 17.60 23.07 25.20 31.73 39.07 42.13 30.53 41.20 0.00 7.33 10.40 16.93 28.27 31.20 17.20 32.93 0.00 3.60 21.73 22.93 25.87 11.07 28.00 0.00 24.40 25.60 23.87 7.60 25.47 0.00 39.33 40.80 29.47 43.47 0.00 40.00 30.00 41.73 0.00 18.13 32.67 0.00 19.33 0.00 Сравнение методов подсчета эволюционного расстояния Для сравнения разных методов подсчета эволюционного расстояния были построены диаграммы. Диаграмма 1: По оси X отложено количество замен, по оси Y отложены пары, между которыми измерено эволюционное расстояние. 210 180 150 120 90 60 30 C D -A -A BA FA EBC A -A EF B BC C D A AB D E -A C F AB D E - A AB CD F- A C E AB D E F- B C FAB D E C C FAB D E D CD FEF E -F FC EB FD FB EC ED D AB -C CD D EF -B -B AB C CD F EF - E -E F- F BC AB B ECD C D BC EF -B -B C C D C -B EF AB -B EF CD EF E - D - B FC C D E D F F- -B C BC E D D - BC -B D F- CD B EF BC C D D EF C EF -B B E- CD BC EF D BC EF D D -B -B C CD -B CD FE EF F BC E EF - B -B C C E BC F- B C D BC EF EF D BC - B - B D CD C EF E -B F C D AA B B BC B C - B C D D BC EF -B - B CD C DE F C -C D -D E EF - E -E F FF 0 Истинное растояние 'Попарное сходств о' Джукс-Кантор Диаграмма 2. Зависимость полученных эволюционных расстояний от истинного расстояния. По оси X отложены истинное количество замен, а по оси Y количество замен, полученное в моделях. 160 140 120 100 80 60 40 20 0 200 200 170 160 140 100 100 100 80 Джукс-Кантор 80 80 60 60 60 40 40 30 20 10 'Попарное сходств о' Построенные диаграммы показывают, что для описания данной модели эволюции лучше подходит алгоритм Джукса-Кантора, т.к. в ней есть последовательности, в которых произошло более 100% замен. Алгоритм "Попарного сходства" применим только в тех случаях, когда организмы практически не разошлись в ходе эволюции, а для далеких последовательностей он не применим. Как видно из диаграммы 2, для качественного определения, того факта, что гены разошлись в ходе эволюции подходят и тот, и другой методы, однако в случае использования алгоритма Джукса-Кантора кривая позволяет точнее определять расхождение генов в ходе эволюции.







