Cравнение аминокислотных последовательностей
advertisement

Cравнение аминокислотных последовательностей белков и нуклеотидных
последовательностей соответствующих генов
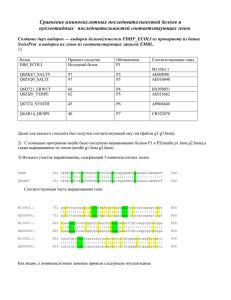
Я создал выборку из 6 аминокислотных последовательностей гомологов моего белка, и найденных в
банке данных Uniprot + последовательность моего белка. Аналогичная выборка была создана для их CDS.
В выборке оказались белки с Identity 99% 96% 76% 51% 43% к моему белку.
Я построил полные выравнивания аминокислотных и нуклеотидных последовательностей моего белка
с ближайшим гомологом (c Identity 99%).
В выравнивании нуклеотидных последовательностей были обнаружены 22 синонимичные замены.
Среди них 18 произошли в третьих позициях кодонов, одна замена во второй (tga на taa (стоп кодон)),
и две замены в первой позиции (tgg на ggg (кодируют глицин), сtg на ttg (кодируют лейцин)).
В выравнивании аминокислотных последовательностей были обнаружены 2 замены: глицин на
триптофан и глицин на серин. Эти замены были вызваны 2 мутациями в нуклеотидной
последовательности, а именно замена кодона tgg на ggg повлекшая замену триптофана на глицин, замена
ggc на agt повлекшая замену глицина на серин. Про последний случай следует сказать, что замена в
третьей позиции кодона является синонимичной, так как триплеты кодирующие глицин и серин
полностью вырождены по третьей позиции. Поэтому замена тимина на гуанин в третьей позиции не
может повлечь замену глицина на серин.
Примечание: так как невозможно определить какая из последовательностей предковая, то под фразой:
«произошла замена X на Y» можно подразумевать также, что произошла замена Y на X.
Соотношение синонимичных и несинонимичных замен нуклеотидов составляет 11 (22/2)
Далее я составил матрицу замен нуклеотидов:
a
a
t
g
c
t
g
1
c
8
3
0
8
1
Из анализа матрицы можно сделать вывод, что пурин чаще заменяется тоже на пурин, тоже самое
можно сказать про пиримидины.
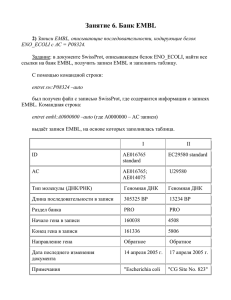
Я определил проценты идентичности аминокислотных и нуклеотидных послследовательностей для
каждой пары белков, построив попарные выравнивания с помощью программы needle. Это было сделанно
с помощью нижеприведенного скрипта:
y=1
until test $y -eq 6 ; do
x=6
until test $x -eq $y ; do
p=`needle p_$x.fasta p_$y.fasta -auto stdout | awk '($2=="Identity:"){print $4}'`
embl=`needle embl_$x.fasta embl_$y.fasta -auto stdout | awk '($2=="Identity:") {print
$4}'`
echo "$x/vs$y" $p $embl|tr -d "()%/"|tr . ,
((x--))
done
let "y= y + 1"
done
результат:
6vs1 42,0 50,7
5vs1 51,1 57,0
4vs1 75,0 69,3
3vs1 96,7 94,0
2vs1 99,3 97,3
6vs2 41,7 48,8
5vs2 50,7 56,9
4vs2 75,0 69,7
3vs2 96,7 94,5
6vs3 42,0 53,3
5vs3 50,4 57,2
4vs3 73,6 69,0
6vs4 40,9 50,5
5vs4 52,9 55,7
6vs5 40,6 49,0
На основе полученных данных я построил график, отображающий связь процента совпадения
нуклеотидных последовательностей с процентом совпадения аминокислотных для моего белка. И сравнил
его с графиком для белка-предшественника гемагглютинина у разных штаммов вируса гриппа.
100
100
90
80
80
70
70
Gene % identity
90
60
50
40
60
50
40
30
30
20
20
10
10
0
0
0
10
20
30
40
50
60
70
80
90
100
0
10
20
30
Prot ID %
40
50
60
70
80
Protein % identity
Сравнение линий трендов данных графиков
100
90
80
70
Gene ID %
Gene ID %
Haem agglutinin
белок Ttaud_Ecoi
60
50
40
30
20
10
0
0
10
20
30
40
50
60
70
80
90
100
Prot ID %
Голубой – линия тренда для TAUD_ECOLI
Черный – линия тренда для белка-предшественника гемагглютинина
Так как линии трендов графиков полностью совпадают, можно предположить, что белки выполняющие
функцию моего белка также консервативны, как и предшественники гемагглютинина у вируса гриппа.
Хотя верится в это с трудом. Ведь вышеопределенное соотношение синонимичных замен к
несинонимичным для двух ближайших гомологов составляет 22/2, что свидетельствует о высокой
консервативности (по сравнению с белком-предшественником гемагглютинина).
90
100