Изучение возможной роли антисенс
advertisement

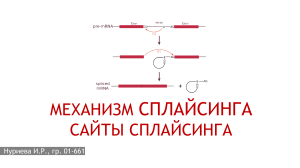
Изучение возможной роли антисенс-РНК в регуляции альтернативного сплайсинга Климова Евгения1, Первушин Дмитрий2 1 Московский Государственный Университет им. М.В. Ломоносова, 2 Факультет биоинформатики и биоинженерии, Московский Государственный Университет им. М.В. Ломоносова jane1390@rambler.ru Аннотация 1. Введение Альтернативный сплайсинг - это процесс, наблюдаемый у эукариот, в результате которого экзоны пре-мРНК, образованные после транскрипции, могут сшиваться не одним, а несколькими различными способами. До недавнего времени было известно о существовании транс-регуляции этого процесса различными белковыми факторами. Но в последние годы были найдены экспериментальные подтверждения трансрегуляции альтернативного сплайсинга молекулами некодирующих РНК. Так, малая ядрышковая РНК HBII-52 принимает участие в регуляции сплайсинга серотонинового рецептора, а некодирующая 17А РНК мешает правильному сплайсингу GABA B2 рецептора. Таким образом, антисенс-транскрибируемые молекулы РНК могут мешать сплайсингу, скрывая от сплайсеосомы сайты сплайсинга, играющие в нѐм ключевую роль. В данной работе мы использовали данные CAGE для поиска точек инициации транскрипции на противоположной цепи ДНК от сайтов сплайсинга. Мы изучили распределение точек инициации транскрипции в окрестности альтернативный сайтов сплайсинга и получили статистические свидетельства возможности регуляции сплайсинга молекулами РНК, используя в качестве контроля конститутивные интроны и экзоны. Мы выяснили, что в интронной части окрестности альтернативных акцепторных и донорных сайтов сплайсинга частота антисенстранскриптов больше по сравнению с окрестностями конститутивных сайтов сплайсинга. Также, наблюдаемая частота встречаемости антисенс-транскриптов в интронной части окрестностей донорных и акцепторных сайтов меньше, чем соответствующая частота в контроле серединных частях конститутивных экзонов. Но больше, чем соответствующая частота в серединных частях конститутивных интронов. Сплайсинг – это процесс модификации РНК после транскрипции, заключающийся в вырезании интронов и сшивании экзонов. Сплайсинг осуществляется большим РНКбелковым комплексом, называемым сплайсеосомой. Правильному распознаванию интронов сплайсеосомой способствуют высоко консервативные участки генома, такие как 5’сайт сплайсинга, точка ветвления, полипиримидиновый тракт и 3’-сайт спласинга, которые являются вспомогательными структурами при осуществлении сплайсинга. [1] Альтернативный сплайсинг - это процесс, наблюдаемый у эукариот, в результате которого экзоны пре-мРНК, образованные после транскрипции, могут сшиваться не одним, а несколькими различными способами. Полученные альтернативным сплайсингом мРНК могут транслироваться в разные белки, таким образом, один ген кодирует несколько различных белков.[2] У человека, приблизительно 95% генов, состоящих из нескольких экзонов, сплайсируются альтернативно. Неправильно сплайсированные мРНК являются причиной многих заболеваний. [3] Существуют различные способы регуляции сплайснига. Наиболее изученной в настоящее время является транс-регуляция сплайсинга белковыми факторами. Например, в регуляции могут участвовать богатые аргинином белки [4], белки, которые своим связыванием с пре-мРНК влияют на еѐ вторичную структуру и тем самым на сам процесс сплайсинга [5]. В последние же годы были описаны случаи транс-регуляции альтернативного сплайсинга некодирующими короткими молекулами РНК. В 2010 году была обнаружена новая некодирующая РНК, названная 17А, транскрибируемая РНК-полимеразой III. Эта РНК была картирована на противоположную цепь интрона 3 гена GPR51, который кодирует GABA B2 рецептор. Интрон 3 содержит два предполагаемых консенсуса, ассоциированных со сплайсингом: полипиримидиновый тракт и точку 101 ветвления. Одновременный синтез GPR51 преmRNA и 17A транскрипта приводит к образованию РНК-РНК дуплекса, в результате эти сайты (полипиримидиновый тракт и точка ветвления), расположенные в области комплементарной 17А РНК оказываются заблокированы. Это и служит причиной, вызывающей события альтернативного сплайсинга.[6] Экспрессия 17А РНК в нейробластоме вызывает синтез альтернативно сплайсированной изоформы, которая разрушает внутриклеточные сигнальные процессы, например, накопление сАМФ и активацию К+ каналов. Также это усиливает секрецию амилоидного β пептида, что вызывает болезнь Альцгеймера.[7] Другая, недавно обнаруженная РНК, способная регулировать сплайсинг, относится к малым ядрышковым РНК. Малые ядрышковые РНК (snoRNAs) – это некодирующие РНК длиной от 60 до 300 нуклеотидов, которые осуществляют 2’О-метилирование и псевдоуридинизацию рибосомных РНК(rRNAs), малых ядерных РНК(snRNAs) и транспортных РНК. HBII-52 –это специфичная для мозга C/D box малая ядрышковая РНК. Еѐ антисенсэлемент комлементарен альтернативно сплайсируемому экзону Vb серотонинового рецептора 5-HT2CR. Серотониновый рецептор располагается на Х хромосоме и состоит из семи трансмембранных сегментов. Экзон V данного рецептора содержит как минимум два альтернативных 5’-сайтов сплайсинга, благодаря чему после сплайсинга образуются разные изоформы, несущие в себе экзон Va либо Vb. Экзон Vb кодирует участок рецептора, отвечающий за связывание с G-белком. Было показано, что HBII-52 влияет на альтернативный сплайсинг 5-HT2CR пре-мРНК. Эта малая ядрышковая РНК блокирует сайленсер, расположенный в комплементарном ей участке экзона Vb. Также было обнаружено, что у больных синдромом Прадера-Вилли не экспрессируется HBII-52. В следствие этого, их изоформа 5-HT2CR мРНК отличается от изоформы здоровых людей.[8] Анализ экспрессии генов с применением антител к кэпированному 5’-концу РНК (Capanalysis gene expression, CAGE) – это метод, разработанный в рамках международных проектов FANTOM, которые были инициированы институтом RIKEN (Omics Science Center). Метод CAGE позволяет определять точки инициации транскрипции с точностью до нескольких нуклеотидов, это позволило подробно изучить структуру и свойства промоторов млекопитающих. Эти открытия мотивировали данную работу, целью которой стал поиск статистических доказательств регуляции альтернативного сплайсинга молекулами РНК, транскрибируемыми с противоположной цепи ДНК, используя данные CAGE (Cap-analysis gene expression) Этот метод, разработанный в рамках международных проектов FANTOM позволяет определять точки инициации транскрипции с точностью до нескольких нуклеотидов. Суть метода: Со случайного праймера на мРНК синтезируется кДНК; КЭП биотинилируется и затем, с использованием антител к биотинилированному КЭПУ, извлекается гибрид мРНК-кДНК; К 5’-концу пришивается линкер, несущий в своѐм составе сайты для XhoI, I-CeuI, XmaJI, или MmeI рестриктаз; После действия рестриктазы получают CAGE-тэги длиной приблизительно 20 п.о.; Затем кДНК клонируют в плазмиде и секвенируют в клонотеках; Полученные CAGE-теги картируются на геном и объединяются в кластеры. [9] 2. Данные и методы Исследование проводилось на человеческом геноме версии hg18. Сайты сплайсинга были взяты из базы данных RefSeq (http://www.ncbi.nlm.nih.gov/RefSeq/). Для поиска транскрипции с противоположной сайтам сплайсинга цепи ДНК были использованы кластеры CAGE-тегов, полученные в рамках проекта FANTOM4 (http://fantom.gsc.riken.jp/4/). Для статистического анализа полученных нами результатов был вычислен Z-score = (X-µ)/σ, позволяющий понять на сколько стандартных отклонений наблюдаемое значение случайной величины больше или меньше среднего генеральной совокупности. Среднее значение (µ) и стандартное отклонение (σ) были определены по случайным выборкам. В данной работе мы имеем дело с малыми величинами порядка 10-2-10-3, значит, вычисление Z-score для статистической оценки наших результатов является не совсем корректным. Поэтому была выбрана вероятностная оценка случайной величины, распределѐнной по Пуассону. Распределение 102 Пуассона моделирует случайную величину, представляющую собой число редких событий в единицу времени или длины при условии, что данные события происходят с некоторой фиксированной средней интенсивностью и независимо друг от друга. При этом вероятность того, что редкое событие произойдѐт k раз, вычисляется по формуле Значиения p-value для альтернативных сайтов сплайсинга и интронов показывают значимое преобладание частоты антисенстранскриптов в окрестности сайтов. В случае же с экзонами, мы видим, что частота кластеров CAGE-тегов в экзоне значимо больше, чем в интронной окрестности альтернативных акцепторных сайтов, и не отличается от частоты антисенс-транскриптов в окрестности альтернативных донорных. Где λ – ожидаемая интенсивность данного события. 4. Выводы 1. В интронной части окрестности альтернативных акцепторных и донорных сайтов сплайсинга частота антисенс-транскриптов больше по сравнению с окрестностями конститутивных сайтов сплайсинга. 3. Результаты и обсуждение Для исследования регуляции альтернативного сплайсинга мы искали точки инициации транскрипции на противоположной цепи ДНК от гена, т.к. антисенс- транскрипты могут маскировать сайты, участвующие в сплайсинге. Мы исследовали наличие точек инициации транскрипции в интронной окрестности радиуса 250 п.о. альтернативных донорных и акцепторных сайтов сплайсинга. В качестве контроля мы выбрали конститутивные сайты сплайсинга и середины конститутивных экзонов и инторонов длиной более 500 п.о. Для сравнения альтернативных сайтов сплайсинга с контролем мы использовали вероятностную оценку для Пуассоновской величины. Используя распределение Пуассона мы получили, что вероятность наблюдать имеющуюся (или большую) частоту кластеров CAGE-тегов в интронной окрестности альтенативных акцепторных сайтов в сравнении с конститутивными акцепторными сайтами очень мала (0.007). Это позволяет нам заключить на уровне значимости 5%, что антисенстранскрипты встречаются в окрестности альтернативных акцепторных сайтов чаще, чем в окрестности конститутивных. Эти антисенстранскрипты могли бы играть роль в регуляции альтернативного сплайсинга. Например, в случае с акцепторными сайтами, транскрибируемые с противоположной цепи некодирующие РНК могли бы маскировать полипиримидиновый тракт и точку ветвления, это бы препятствовало связыванию сплайсеосомы и, следовательно, прохождению сплайсинга. На 5% уровне значимости у нас не достаточно оснований полагаться, что частота встречаемости антисенс-транскриптов в интронной окрестности альтернативных донорных сайтов отличается от таковой в интронной окрестности конститутивных донорных сайтов. 2. Наблюдаемая частота встречаемости антисенс-транскриптов в интронной части окрестностей донорных и акцепторных сайтов меньше, чем соответствующая частота в контроле - серединных частях конститутивных экзонов. 3. Наблюдаемая частота встречаемости антисенс-транскриптов в интронной части окрестностей донорных и акцепторных сайтов больше, чем соответствующая частота в контроле - серединных частях конститутивных интронов. 4. Сенс-транскрипция окрестности сайтов преобладает над транскрипцией. в сплайсинга антисенс- 5. Благодарности Данная работа выполнена совместно с Д.Д. Первушиным и А.А. Мироновым. 6. Список литературы [1] Clancy S, (2008). RNA Splicing: Introns, Exons and Spliceosome. Nature Education 1 (1). [2] Black, Douglas L. (2003). Mechanisms of alternative pre-messenger RNA splicing. Annual Reviews of Biochemistry 72 (1): 291–336. [3] Pan Q; Shai O, Lee LJ, Frey BJ, Blencowe BJ. (Dec 2008). Deep surveying of alternative splicing complexity in the human transcriptome by high- 103 throughput sequencing. Nature Genetics 40 (12): 1413–1415 [4] Qian W, Liang H, Shi J, Jin N, Grundke-Iqbal I, Iqbal K, Gong CX, Liu F. (2011). Regulation of the alternative splicing of tau exon 10 by SC35 and Dyrk1A. Nucleic Acids Res. [5] Warf MB, Berglund JA. (2010). Role of RNA structure in regulating pre-mRNA splicing. Trends Biochem Sci. 35(3):169-78. [6] Massone S, Vassallo I, Fiorino G, Castelnuovo M, Barbieri F, Borghi R, Tabaton M, Robello M, Gatta E, Russo C, Florio T, Dieci G, Cancedda R, Pagano A. (2010). 17A, a novel non-coding RNA, regulates GABA B alternative splicing and signaling in response to inflammatory stimuli and in Alzheimer disease. Neurobiol Dis. 41(2):308-17. [7] Selkoe, D.J., (1990). Deciphering Alzheimer's disease: the amyloid precursor protein yields new clues. Science 248, 1058–1060. [8] Kishore S, Stamm S. (2006). The snoRNA HBII52 regulates alternative splicing of the serotonin receptor 2C. Science 311(5758):230-2. [9] Shiraki T, Kondo S, Katayama S, Waki K, Kasukawa T, Kawaji H, Kodzius R, Watahiki A, Nakamura M, Arakawa T, Fukuda S, Sasaki D, Podhajska A, Harbers M, Kawai J, Carninci P, Hayashizaki Y. (2003). Cap analysis gene expression for high-throughput analysis of transcriptional starting point and identification of promoter usage. Proc Natl Acad Sci U S A 100(26):15776-81 104