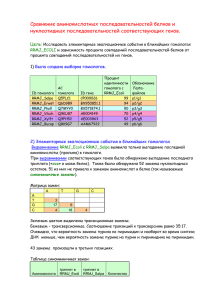
Приложение № 1 - Казанский (Приволжский) федеральный
advertisement

Министерство образования и науки Российской Федерации Федеральное государственное автономное образовательное учреждение высшего профессионального образования «Казанский (Приволжский) федеральный университет» УДК 53.082.722.56, 53.083.2 № госрегистрации 01201060140 Инв.№ 1.1.2.06.2.20/87/11 УТВЕРЖДАЮ Проректор по научной деятельности д-р геол.- минерал. наук, профессор ______________ Д.К.Нургалиев «___»_________ ______ г. ОТЧЕТ О НАУЧНО-ИССЛЕДОВАТЕЛЬСКОЙ РАБОТЕ В рамках федеральной целевой программы «Научные и научно-педагогические кадры инновационной России» на 2009-2013 годы по теме: ПРОСТРАНСТВЕННОЕ СТРОЕНИЕ АB-ПЕПТИДОВ И ФУНДАМЕНТАЛЬНЫЕ СВОЙСТВА ЛИПИДНЫХ МЕМБРАН В УСЛОВИЯХ ИХ ВЗАИМОДЕЙСТВИЯ. КОМПЛЕКСНОЕ ИССЛЕДОВАНИЕ МЕТОДАМИ ЯМР Шифр заявки «2010-1.1-142-043-024» (промежуточный, этап № 4) Наименование этапа: «Межпротонные расстояния в исследованных пептидах и в комплексах пептид - поверхность липидных мембран» Руководитель проекта: ___________________ (д.х.н., профессор, А.В.Аганов) подпись, дата Казань 2011 СПИСОК ИСПОЛНИТЕЛЕЙ Научный руководитель, д.х.н., профессор Аганов А.В. подпись, дата (разделы 1, 2, 3, 4) Исполнители В.н.с., д.х.н., профессор Клочков В.В. подпись, дата Г.н.с., д.ф-м.н., профессор (разделы 1, 2, 3, 4) Скирда В.Д. подпись, дата В.н.с., д.ф.-м.н. (разделы 1, 4) Филиппов А.В. подпись, дата Ведущий инженер, к.ф.-м.н. (разделы 2, 3 ) Клочков А.В. подпись, дата Ассистент, к.ф.-м.н. (разделы 1, 3) Юльметов А.Р. подпись, дата С.н.с., к.ф.-м.н. (разделы 1, 4) Васильев Г.И. подпись, дата Ассистент, к.ф.-м.н (разделы 1, 2) Рудакова М.А.. подпись, дата Инженер (разделы 2, 3 ) Халуиллина А.В.. подпись, дата Аспирант (разделы 2,3 ) Мунавиров Б.В. подпись, дата Аспирант (разделы 2, 3) Ефимов С.В. подпись, дата Аспирант (разделы 1, 2) Усачев К.С. подпись, дата Аспирант (разделы 1,2,3,4) Блохин Д.С. подпись, дата Аспирант (разделы 1,2) Николаева Т.Н. подпись, дата 2 (разделы 1,4) Техник Попова Г.К. подпись, дата Нормоконтролер (разделы 1,2) Рыбасова В.И. подпись, дата 3 РЕФЕРАТ Отчёт – 59 страниц, 15 рисунков, 4 таблицы, библиография - 32 источника, 3 приложения ОЛИГОПЕПТИДЫ, А - ПЕПТИДЫ, ЯМР, СПЕКТРОСКОПИЯ, ДВУМЕРНАЯ СПЕКТРОСКОПИЯ, NOESY, COSY, HSQC, TOCSY, HSQC-TOCSY МОДИФИКАЦИИ На данном этапе целью выполнения Проекта “Пространственное строение Аb-пептидов и фундаментальные свойства липидных мембран в условиях их взаимодействия. Комплексное исследование методами ЯМР” планировалось проведение исследований по разделу: “Межпротонные расстояния в исследованных пептидах и в комплексах пептид - поверхность липидных мембран” и получение следующих результатов: - проведение двумерных ЯМР NOESY экспериментов для синтетического А(1-40) пептида нативного типа и его активного фрагмента А(16-22) в растворе; обработка результатов экспериментов и определение количественной информации о межпротонных расстояниях в исследованных пептидах; - проведение двумерных ЯМР NOESY экспериментов для исследуемых комплексов: пептид - поверхность липидных мембран в растворе; обработка результатов двумерных ЯМР NOESY экспериментов и определение количественной информации о межпротонных расстояниях в этих комплексах; - описание строения комплексов: пептид - поверхность липидных мембран в растворе на основании количественной информации о межпротонных расстояниях в этих комплексах. Объектами исследований на данном этапе Проекта были: синтетический А(140) пептид нативного типа (H-Asp-Ala-Glu-Phe-Arg-His-Asp-Ser-Gly-Try--Glu-Val-His- His-Gln-Lys-Leu-Val-Phe-Phe-Ala-Glu-Asp-Val-Gly-Ser-Asn-Lys-Gly-Ala-Ile-Ile-GlyLeu-Met-Val-Gly-Gly-Val-Val-OH), его активный фрагмент А(16-22) (NAc-Lys-LeuVal-Phe-Phe-Ala-Glu-NH2). В результате проведены двумерные ЯМР NOESY эксперименты для синтетического А(1-40) пептида нативного типа и его активного фрагмента А(16-22) в растворе; показано, что применение двумерной NOESY ЯМР спектроскопии к исследованию олигопептидов, подпадающих под условие быстрого движения неэффективно; в этом случае не наблюдаются, либо наблюдаются слабые по 4 интенсивности кросс-пики в спектрах ЯМР NOESY, что затрудняет получение количественной информации о межпротонных расстояниях; предложено информацию о пространственном строении синтетического А(1-40) пептида и его активного фрагмента А(16-22) в растворах извлекать из таблиц координат атомов этих пептидов (в pdb-формате) определенных в результате анализа величин остаточного диполь-дипольного взаимодействия (см. отчет по Проекту, 3-этап) Проведены двумерные ЯМР NOESY эксперименты для исследуемых комплексов: пептид - поверхность мицелл на основе додецилсульфата натрия в растворе; обработаны результаты двумерных ЯМР NOESY экспериментов и определены межпротонные расстояния в бета-амилоиде А(1-40) и в гептапептиде А(16-22) в этих комплексах. Впервые описано пространственное строение комплексов: пептид - поверхность мицелл на основе додецилсульфата натрия в растворе на основании количественной информации о межпротонных расстояниях в олигопептидах в этих комплексах и качественном рассмотрении изменений химических сдвигов протонов олигопептидов при переходе от растворов к комплексам на основе додецил сульфата натрия. Областью применения полученных результатов может быть фундаментальная наука (биофизика, биохимия, физиология клетки), фармакология, медицина. Подобная полученная структурная информация является практически значимой, поскольку позволит провести исследования биохимических свойств изучаемых пептидов в комплексах пептид-модель мембраны, выяснить роль мембраны при образовании промежуточных структур агрегации и механизмы изменения свойств мембраны (проницаемости) при взаимодействии с пептидами, что является последующими задачами Проекта. 5 ОБОЗНАЧЕНИЯ И СОКРАЩЕНИЯ ЯМР ядерный магнитный резонанс COSY Correlation spectroscopy – двумерная корреляционная спектроскопия TOCSY Total correlation spectroscopy – полная корреляционная двумерная спектроскопия HSQC Heteronuclear single-quantum correlation – двумерная гетероядерная корреляционная спектроскопия HMBC Heteronuclear multiple-quantum correlation - двумерная много-квантовая гетероядерная корреляционная спектроскопия HMQC Heteronuclear multiple-bond correlation - двумерная полная много-квантовая гетероядерная корреляционная спектроскопия CP/MAS NMR 13С Cross polarisation magic angle spinning NMR 13 С – ЯМР метод кросс-поляризации и вращение под магическим углом на ядрах 13С NOESY Nuclear Overhauser effect spectroscopy –спектроскопия ядерного эффекта Оверхаузера ХС химический сдвиг ТМС тетраметилсилан, его сигналы в спектрах ЯМР 1Н и 13 С приняты за 0 м.д. м.д. миллионная доля – единица измерения химических сдвигов в спектрах ЯМР 1Н и 13С ТФЭ трифторэтанол 6 СОДЕРЖАНИЕ ОБОЗНАЧЕНИЯ И СОКРАЩЕНИЯ 6 СОДЕРЖАНИЕ 7 ВВЕДЕНИЕ 8 1. Проведение двумерных ЯМР NOESY экспериментов для исследуемых 15 пептидов растворе; обработка результатов экспериментов и определение количественной информации о межпротонных расстояниях в исследованных пептидах. 2. Проведение двумерных ЯМР NOESY экспериментов для исследуемых 19 комплексов: пептид- поверхность липидных мембран в растворе. 3. Обработка результатов двумерных ЯМР NOESY экспериментов и 30 определение количественной информации о межпротонных расстояниях в комплексах пептид - поверхность липидных мембран. 4. На основании данных о межпротонных расстояниях описание 33 строения комплексов: пептид- поверхность липидных мембран в растворе. ЗАКЛЮЧЕНИЕ 35 Список использованных источников 36 Приложение А. Список работ, выполненных в рамках проекта, 40 опубликованных в 2011 г. Приложение Б. 42 Приложение В. 46 7 Введение Как отмечалось в предыдущих отчетах по Проекту, агрегация амилоидных пептидов сопровождает развитие таких нейродегенеративных заболеваний как болезни Альцгеймера, Хантингтона, и другие [1]. Результат агрегации наблюдается в форме амилоидных фибрилл, в которых молекулы протеина в конформации складки уложены поперек направления оси фибриллы, а также нефибриллярных агрегатов. Цитотоксичность олигомерные продукты [2]. проявляют В не настоящее сами время фибриллы, а исследованы некоторые процессы фибриллярной агрегации в протеинах, связанных с развитием болезни Альцгеймера [3, 4]. Начальные стадии агрегации пептидов исследованы мало, в частности Апептиды и лизоцим в растворе были исследованы методом ЯМР с импульсным градиентом магнитного поля [5-8]. Открытым остается вопрос - образуются ли олигомерные агрегаты при агрегации амилоидного А-пептида. Хорошо известно, что биологическая активность протеинов или белков связана с их пространственным строением, что, несомненно, делает актуальным данное исследование. Изучение конформаций олигопептидов также важно, поскольку их можно рассматривать в качестве структурных блоков протеинов и знание их строения может быть использовано для предсказания конфигурации цепей полипептидов [9,10]. Нейротоксичное действие альцгеймеровских амилоидных пептидов проявляется в результате их взаимодействия с клеточной мембраной [3]. Отсюда описание пространственного строения как самих амилоидных А-пептидов, так комплексов бета-амилоиды – мембрана, позволит подойти к фундаментальному пониманию механизмов протекающих на поверхности клеток, что может дать возможность поиска лекарственных препаратов, ингибирующих образование сенильных бляшек. Строение относительно небольших органических молекул в растворах, как правило, определяется по данным одномерной ЯМР спектроскопии, двумерных методик (NOESY, корреляционных методик COSY, HSQC) [11,12], динамического ЯМР [13-15]. Спектроскопия ядерного эффекта Оверхаузера (2D NOESY) позволяет также определять расстояния между магнитными ядрами до 5 Å, и, тем самым, устанавливать пространственную структуру растворённых соединений [11,12]. К сожалению, применение двумерной NOESY ЯМР спектроскопии к исследованию низкомолекулярных органических соединений, подпадающих под условие быстрого 8 движения ( 0 * c << 1, где c - время корреляции молекулярного движения, 0 - угловая скорость прецессии магнитных ядер) не всегда эффективно. В этом случае наблюдаются слабые по интенсивности кросс-пики в спектрах ЯМР NOESY, что затрудняет получение количественной информации о межпротонных расстояниях. Проведение двумерных ЯМР NOESY экспериментов для комплексов протеина с мицелярными системами на основе поверхностно-активных веществ, позволяет надеяться на определение расстояний между магнитными ядрами в олигопептидах методом двумерной ЯМР NOESY спектроскопии. С точки зрения ЯМР спектроскопии, комплексобразование увеличивает молекулярную массу системы, что может перевести протеин из разряда малых молекул, подпадающих под условие быстрого движения, в разряд молекул, подпадающих под условие медленного движения. Последнее обстоятельство позволит использовать спектроскопию ЯМР NOESY при решении структурных задач и для небольших по количеству аминокислотных остатков протеинов. Одной из составляющих данного Проекта является установление пространственной структуры, а именно определение конформаций и геометрических параметров (координат атомов) ряда пептидов: синтетический А(1-40) пептид нативного типа (Рисунок 1), его вариации при изменении одной из аминокислот в первичной структуре А(10-35) и активный фрагмент пептида А(1-40) - пептид А(16-22) в растворе и твердой фазе. В качестве методов исследования использованы ЯМР 1Н, 13 С спектроскопия, включая подход, основанный на анализе остаточных величин диполь-дипольного взаимодействия между магнитными ядрами использованы двумерная модификации), 13 спектроскопия ЯМР (COSY, 13 С и 1Н. Также HSQC и NOESY C CP/MAS ЯМР и HR-MAS спектроскопия и теоретическое моделирование молекулярных структур с программным обеспечением MOPAC 7.0 (PM 3). 9 Leu Val Phe Phe Ala Glu Lys Lys Asp Val Gln Gly His Ser Asn His Lys Val Gly Ala Glu Tyr Ile Ile Gly Ser Gly Asp His Leu Met Val Arg Phe Glu Gly Gly Ala Asp Val Val Рисунок 1 - Схематическое изображение бета-амилоида Aβ1-40 (H-Asp-Ala-Glu-PheArg-His-Asp-Ser-Gly-Try--Glu-Val-His-His-Gln-Lys-Leu-Val-Phe-Phe-Ala-Glu-Asp-ValGly-Ser-Asn-Lys-Gly-Ala-Ile-Ile-Gly-Leu-Met-Val-Gly-Gly-Val-Val-OH). Черным цветом выделен участок Aβ16-22, который, как предполагается, отвечает за агрегацию между амилоидами. На четвертом этапе выполнения Проекта “Пространственное строение Аbпептидов и фундаментальные свойства липидных мембран в условиях их взаимодействия. Комплексное исследование методами ЯМР” планировалось проведение исследований по разделу: “Межпротонные расстояния в исследованных пептидах и в комплексах пептид - поверхность липидных мембран” и получение следующих результатов: - проведение двумерных ЯМР NOESY экспериментов для синтетического А(1-40) пептида нативного типа и его активного фрагмента А(16-22) в растворе; обработка результатов экспериментов и определение количественной информации о межпротонных расстояниях в исследованных пептидах; - проведение двумерных ЯМР NOESY экспериментов для исследуемых комплексов: пептид - поверхность липидных мембран в растворе; обработка результатов двумерных ЯМР NOESY экспериментов и определение количественной информации о межпротонных расстояниях в этих комплексах; 10 - описание строения комплексов: пептид - поверхность липидных мембран в растворе на основании количественной информации о межпротонных расстояниях в этих комплексах. Подобные исследования являются практически значимыми, поскольку полученные результаты позволят проводить исследования биохимических свойств изучаемых пептидов в комплексах пептид-модель мембраны. Объекты исследования Методом ЯМР NOESY спектроскопии исследованы, следующие пептиды: синтетический А(1-40) пептид нативного типа (H-Asp-Ala-Glu-Phe-Arg-His-Asp-SerGly-Try--Glu-Val-His-His-Gln-Lys-Leu-Val-Phe-Phe-Ala-Glu-Asp-Val-Gly-Ser-Asn-LysGly-Ala-Ile-Ile-Gly-Leu-Met-Val-Gly-Gly-Val-Val-OH), и активный фрагмент А(16-22) (NAc-Lys-Leu-Val-Phe-Phe-Ala-Glu-NH2) пептида А(1-40) в растворах и в комплексах: пептид - поверхность липидных мембран в растворе. Подготовка образцов и условия проведения ЯМР исследований пептидов А(1622) и А(1-40) Пептиды А(16-22) и А(1-40) были синтезированы по стандартному методу твердофазного синтеза FMOC [16-18] с помощью автоматического синтезатора пептидов ABI 433A (Applied Biosystems, Foster City, CA, USA) на кафедре прикладной химии Технического Университета Лулео (Швеция) [18]. Отделение пептидов от подложки и защитных групп производилось в кислой среде на основе трифторуксусой кислоты. Очистка пептидов производилась методом высокопроизводительной жидкостной хроматографии на приборе Series 200 Perkin Elmer HPLC System (Waltham, MA, USA) в градиенте вода - ацетонитрил. Качество конечного продукта характеризовали методом MALDI-TOF (matrix-assisted laser desorption-ionization) масс-спектрометрии в университете Карлсруэ (Karlsruhe Institute of Technology, Germany). Регистрация ЯМР 1H (500 МГц) и 13C (125.69 МГц) спектров пептидов А(16-22) и А(1-40) в растворах боратного буфера (Na2B4O7·10H2O+HCL+D2O), фосфатного буфера (NaH2PO4·2H2O/ Na2HPO4·2H2O +D2O) и трифторэтанола (CF3CD2OD) и в комплексах: пептид - поверхность липидных мембран в растворе проводилась на ЯМР спектрометре высокого разрешения AVANCE-500 IITM фирмы «Bruker». 11 Спектрометр работает в режиме внутренней стабилизации поля по линии резонанса дейтерия (2H). При записи протонных спектров в изотропном растворителе использовались 900 импульсы и поднасыщение сигнала воды, либо 300 импульсы без поднасыщения растворителя. Задержки между импульсами составляли 2 сек., число накоплений – от 4 до 100, ширина спектра – 11 м.д., число точек – 26832. При записи спектров ЯМР 13 C использовались 300 импульсы и развязка от протонов (WALTZ– 16). Межимпульсные задержки равнялись 2 сек., число накоплений – 19485, ширина спектра – 236,6 м. д., число точек – 65536. При обработке спектра применялась цифровая экспоненциальная фильтрация с константой 3 Гц. Одномерные спектры регистрировались при стабилизированной температуре 200 C или 250 C. Точность определения химических сдвигов на ядрах 1H равнялась 0.01 м.д., на ядрах 13С - 0.1 м.д., констант спин-спинового взаимодействия – 0.3 Гц. Концентрации пептидов в растворах была не менее 10 % по массе. Концентрация боратного буфера: Na2B4O7·10H2O составляла 5.82% по массе и 0.1 М соляной кислоты в воде (отношение H2O к D2O - 10/1); фосфатного буфера: NaH2PO4·2H2O 28,16% по массе, Na2HPO4·2H2O 26.71% по массе, H2O – 44.13% по массе, D2O –1% по массе [19-24]. Двумерные эксперименты ЯМР NOESY (1H-1H) соответствующих спектров пептидов А(16-22) и А(1-40) буфера (Na2B4O7·10H2O+HCL+D2O), фосфатного [25,26] по записи в растворах боратного буфера (NaH2PO4·2H2O/ Na2HPO4·2H2O +D2O), трифторэтанола (CF3CD2OD) и в комплексах: пептид поверхность липидных мембран в растворе также были проведены на ЯМР спектрометре AVANCE-500 IITM фирмы «Bruker». Двумерная ЯМР спектроскопия NOESY в определении пространственного строения органических соединений Сегодня при проведении исследований строения органических и биоорганических соединений в растворах широко используются приемы и подходы двумерной ЯМР (COSY, HSQC, и NOESY модификации) спектроскопии. Идея применения двумерного (2М) преобразования Фурье в спектроскопии ЯМР впервые была предложена Джинером Дж. в 1971 году. Он предложил проводить 2М-Фурье преобразование сигнала ЯМР, полученного в виде функции двух временных переменных, вследствие чего интенсивность сигнала становится зависимой от двух частотных переменных. 12 Любой двумерный ЯМР эксперимент можно разбить на четыре временных интервала: первый - подготовительный, система спинов приходит (или приводится) в начальное состояние, обычно состояние теплового равновесия. Второй - период эволюции длительностью t1, в течение которого выведенная из равновесия спиновая система эволюционирует. Третий - период смешивания (в некоторых экспериментах может отсутствовать), в течение которого в изучаемой спиновой системе происходит перенос когерентности или поляризации. Четвёртый - период регистрации. В течение времени t2 регистрируется интенсивность сигнала ЯМР S как функция от t2. Такая процедура повторяется многократно с различной величиной t1 при неизменных остальных параметрах. Результирующий сигнал во временной области S(t1,t2) становится зависимым от двух переменных t1 и t2. Двумерное Фурьепреобразование переводит сигнал S(t1,t2) в спектр частотной области S(1,2). Идея использования двумерной ЯМР спектроскопии для изучения таких процессов как химический обмен, кросс-релаксация и спиновая диффузия была предложена Эрнстом Предложенный [11,12]. алгоритм эксперимента и соответствующая импульсная последовательность (Рисунок 2) получили название NOESY (Nuclear Overhauser Exchange Spectroscopy- спектроскопия ядерного эффекта Оверхаузера и обмена). Физическую сущность данного эксперимента можно представить, рассматривая случай взаимного обмена между двумя состояниями А и В (т.е. когда kAB = kBA) в системе невзаимодействующих спинов (JAB= 0). Примем, что времена продольной релаксации равны, т.е. Т1А=Т1В и обозначим равновесную намагниченность в позиции А как МА, а в позиции В - МВ. Рисунок 2. Импульсная последовательность в эксперименте NOESY. В результате действия первого 90- импульса намагниченность повернётся по часовой стрелке на 90 вокруг оси x и в момент t1= 0 будет направлена вдоль оси -y. Если обмен медленный, то в период t1 эволюцию намагниченностей МА и МВ можно 13 рассматривать независимо. В таком случае намагниченности будут независимо прецессировать в плоскости xy с частотами равными химическим сдвигам А и В. Благодаря поперечной релаксации намагниченность уменьшится в exp(-t1/T2) раз. Второй 90- импульс переводит намагниченности МА и МВ в плоскость xz, zкомпонента намагниченности МА оказывается пропорциональной cos At1, а МBZ(m=0)cosBt1. Модулированные таким образом компоненты z- намагниченности отличаются от равновесных. В течение m будет происходить процесс перераспределения продольной намагниченности в сторону состояния равновесия за счет химического обмена (процесс идентичен переносу намагниченности в экспериментах Форзена - Гофмана) или процессов кроссрелаксации. X-компоненты намагниченности в период смешивания не содержат полезной информации. Они уничтожаются либо импульсом, создающим неоднородности в плоскости ху, либо в серии экспериментов с чередующимися фазами импульсов. Последний 90- импульс переводит z- намагниченность в плоскость x,y, обуславливая наблюдаемую поперечную намагниченность. Часть спинов, прецессировавших в течении t1 с частотой А, в результате процессов обмена или процессов кросс-релаксации в течение m, стала в период t2 прецессировать с частотой В. Это приводит к появлению на 2М спектре так называемых кросс пиков с координатами (1,2) = (А,В). Появление таких кросс пиков в системе невзаимодействующих спинов свидетельствует о процессах обмена намагниченностями или процессов кросс-релаксации. Количественная двумерная спектроскопия ЯМР NOESY недостаточно адаптирована к исследованию структуры относительно малых молекул, попадающих под условие быстрого движения (0с <<1; с – время корреляции, 0 – резонансная частота ядер). Нами предложен способ получения количественной информации о меж-протонных расстояниях для малых органических молекул путем комбинации ЯМР NOESY эксперимента с методами обработки данных [27]. 14 1. ПРОВЕДЕНИЕ ДВУМЕРНЫХ ЯМР NOESY ЭКСПЕРИМЕНТОВ ДЛЯ ИССЛЕДУЕМЫХ ПЕПТИДОВ РАСТВОРЕ; ОБРАБОТКА РЕЗУЛЬТАТОВ ЭКСПЕРИМЕНТОВ ИНФОРМАЦИИ И О ОПРЕДЕЛЕНИЕ МЕЖПРОТОННЫХ КОЛИЧЕСТВЕННОЙ РАССТОЯНИЯХ В ИССЛЕДОВАННЫХ ПЕПТИДАХ Ранее (см. отчет по Проекту, 1-этап) были проведены исследования методами ЯМР синтетического А(1-40) пептида нативного типа и активного фрагмента А(1-40) пептида (16-22): проведена регистрация одномерных ЯМР (1Н и 13 С) спектров синтетического А(1-40) пептида нативного типа, его фрагмента А(10-35) и активного фрагмента А(16-22) пептида А(1-40) в растворах и CP/MAS ЯМР 13 С спектров исследуемых пептидов в твердой фазе; проведена запись двумерных спектров ЯМР (COSY, HSQC, TOCSY, HSQC-TOCSY модификации) исследуемых пептидов в растворах; произведено полное отнесение сигналов ЯМР на ядрах 1H и 13C. Структурная формула гептапептида Aβ(16-22) NAc-Lys-Leu-Val-Phe-Phe-AlaGlu-NH2 приведена на рисунке 1.1. Рисунок 1.1 - Структурная формула гептапептида Aβ(16-22) (NAc-Lys-Leu-Val-PhePhe-Ala-Glu-NH2). На рисунке 1.2 представлен одномерный 1 Н (500 МГц) спектр ЯМР гептапептида NAc-Lys-Leu-Val-Phe-Phe-Ala-Glu-NH2 в растворе боратного буфера (Na2B4O7·10H2O+D2O). Представленное на рисунке 1.2 отнесение сигналов в ЯМР 1Н спектре гептапептида NAc-Lys-Leu-Val-Phe-Phe-Ala-Glu-NH2 было сделано с 15 использованием двумерных гетеро-корреляционных экспериментов ЯМР (см. отчет по Проекту, 1-этап). Рисунок 1.2 - Одномерный 1Н (500 МГц) спектр ЯМР гептапептида NAc-Lys-LeuVal-Phe-Phe-Ala-Glu-NH2 в растворе боратного буфера (Na2B4O7·10H2O+D2O) при pH = 8.4. Т= 293 К. Для определения межпротонных расстояний, напрямую характеризующих пространственную геометрию гептапептида Aβ16-22 (NAc-Lys-Leu-Val-Phe-Phe-AlaGlu-NH2) в растворе, записывали двумерные ЯМР NOESY спектры (рисунок 1.3, как пример) с вариацией времени смешивания τm. Как следует из одномерного 1Н (500 МГц) спектра ЯМР гептапептида NAc-Lys-Leu-Val-Phe-Phe-Ala-Glu-NH2, в двумерном ЯМР NOESY спектре мы вправе ожидать большое число кросс-пиков, отражающих близкое пространственное расположение (до 5 Ао) между разнообразными протонами, в частности между протонами алкильных групп и NH протонами (область 1-3 м.д. – 7.5-9 м.д.). 16 Рисунок 1.3 - Двумерный 1Н-1H NOESY (τm=100 мс) спектра ЯМР гептапептида Aβ1622 в растворе боратного буфера (Na2B4O7·10H2O+D2O) при pH = 8.4. Т= 293 К. Как следует из рисунка 1.3, подобных кросс-пиков в двумерном ЯМР спектре не наблюдается, что объяснимо неэффективностью применения двумерной NOESY ЯМР спектроскопии к исследованию низкомолекулярных органических соединений, подпадающих под условие быстрого движения ( корреляции молекулярного движения, 0 * c << 1, где c - время 0 - угловая скорость прецессии магнитных ядер). В этом случае не наблюдаются, либо наблюдаются слабые по интенсивности кросс-пики в спектрах ЯМР NOESY, что затрудняет получение количественной информации о межпротонных расстояниях. На рисунке 1.4 представлен двумерный 1Н-1H NOESY (τm=100 мс) спектр ЯМР бета-амилоида Aβ1-40 в растворе H2O+D2O, в котором наблюдаются слабые по интенсивности кросс-пики (выделено). К сожалению, подобные спектры также не позволяют получить количественную информацию о межпротонных расстояниях в исследуемых молекулах, подпадающих под условие быстрого движения. 17 Рисунок 1.4 - Двумерный 1Н-1H NOESY (τm=100 мс) спектр (фрагмент) ЯМР бетаамилоида Aβ1-40 в растворе H2O+D2O при Т= 293 К. Таким образом, информацию о пространственном строении синтетического А(1-40) пептида нативного типа (H-Asp-Ala-Glu-Phe-Arg-His-Asp-Ser-Gly-Try--GluVal-His-His-Gln-Lys-Leu-Val-Phe-Phe-Ala-Glu-Asp-Val-Gly-Ser-Asn-Lys-Gly-Ala-IleIle-Gly-Leu-Met-Val-Gly-Gly-Val-Val-OH), и его активного фрагмента А(16-22) (NAcLys-Leu-Val-Phe-Phe-Ala-Glu-NH2) в растворах мы можем извлечь из таблиц координат атомов этих пептидов (в pdb-формате) определенных в результате анализа величин остаточного диполь-дипольного взаимодействия между магнитными ядрами 13 С и 1 Н, разделенных одной химической связью (1D) по программе DYNAMO (см. отчет по Проекту, 3-этап). 18 2. ПРОВЕДЕНИЕ ДВУМЕРНЫХ ЯМР NOESY ЭКСПЕРИМЕНТОВ ДЛЯ ИССЛЕДУЕМЫХ КОМПЛЕКСОВ: ПЕПТИД- ПОВЕРХНОСТЬ ЛИПИДНЫХ МЕМБРАН В РАСТВОРЕ Протеины могут взаимодействовать с мембраной клетки преимущественно двумя способами: проникая сквозь бислой (и тогда говорят об интегральных мембранных белках) или образовывать комплекс с поверхностью бислоя (периферийные или внешние мембранные белки) [28]. В настоящее время ЯМР спектроскопия широко используется для исследования структуры липидной мембраны, однако ее приложения к исследованию комплексов протеин – мембрана клетки все еще ограничены. Известно, что хорошей моделью мембранной поверхности, в том числе и для структурных исследований методом ЯМР спектроскопии [29], являются мицеллы и мицеллярные комплексы. Одной из наиболее полных работ, выполненных в этом направлении является работа [30], в которой методами спектроскопии ЯМР (TOCSY, HSQC, HMBC, NOESY) изучена структура комплекса протеин (Gly-Leu-Phe-AspLys-Leu-Lys-Ser-Leu-Val-Ser-Asp-Asp-Lys-Lys) – мицеллы. Для исследования комплексов протеин-поверхность мембраны доступны два варианта синтетической модели поверхности мембраны клетки: мицеллы на основе поверхностно-активных веществ, и небольшие фосфолипидные везикулы [29,30]. Среда, которая наиболее близко соответствует нативному бислою липида, состоит из фосфолипидных везикул, минимальный размер частиц которых составляет 250-300 Å в диаметре. Частицы такого размера имеют большое вращательное время корреляции (τc ~ 4 x 10-6 секунд). Длинные времена корреляции приводят к коротким значениям времен поперечной релаксации Т2, что, в свою очередь, приводит к уширению резонансных сигналов в спектрах ЯМР и к увеличению спиновой диффузии в 1 Н NOE экспериментах. Короткие времена поперечной релаксации Т2 приводят к уменьшению информативности двумерных ЯМР экспериментов (TOCSY, HSQC, HMBC, NOESY), необходимых как для отнесения резонансных сигналов, так и для определения пространственной структуры протеинов в комплексе [11,12]. Таким образом, частицы такого размера не подходят для двумерных экспериментов и, отсюда следует, что структурные исследования, использующие 1 Н NOE эксперименты, ограничены для молекул больших молекулярных масс, связанными с небольшими мицеллами поверхностно-активных веществ. 19 Известно, что поверхностно-активные вещества образуются амфифильными молекулами, имеющими гидрофобные и гидрофильные участки. Кроме фосфолипидов, формирующих бислои или мультибислои в водных средах, существуют и другие органические соединения, формирующие мицеллярные системы, в которых мицеллы рассредоточены по всему объему и находятся в быстром обмене с мономерными структурами. Критической концентрацией мицеллообразования поверхностно-активных веществ является концентрация ПАВ в растворе, при которой в системе образуются в заметных количествах устойчивые мицеллы. Полярная группа мицелл поверхностно-активных веществ в водной среде расположена на оболочке мицеллы, которая является гидрофильной, а центральная часть мицеллы является гидрофобной [29,30]. В водном растворе мицеллы ведут себя как глобулярные белки, содержащие от 60 мономерных молекул, при этом частицы такого размера имеют относительно небольшое вращательное время корреляции (τc ~ 5 x 10-8 секунд). Важным с точки зрения ЯМР спектроскопии, является то, что при связывании протеина с мицеллами образуется комплекс протеин-мицелла, молекулярная масса которого становится больше, чем у несвязанного протеина, что может перевести молекулу протеина из разряда малых молекул, подпадающих под условие быстрого обмена, в разряд молекул, подпадающих под условие медленного обмена [11,12]. Последнее обстоятельство позволяет использовать спектроскопию ЯМР NOESY при решении структурных задач и для небольших по количеству аминокислотных остатков протеинов. Мицеллярные системы на основе додецилсульфата натрия (ДСН) (рисунок 2.1) образуются в воде при минимальной концентрации 8.1 мМ. Рисунок 2.1 - Структурная формула додецилсульфата натрия. Мицеллы ДСН могут быть использованы для моделирования поведения протеинов на биологических мембранах для небольших гидрофобных протеинов, которые образуют комплексы, связываясь непосредственно с мицеллой ДСН. 20 Отметим, что у синтетических мицелл ДСН, подобно многим биологическим мембранам, имеется поверхностно-отрицательный заряд. От величины этого заряда зависит критическая концентрация мицеллообразования ДСН при формировании мицеллы. Определение концентрации додецилсульфата натрия в воде, при которой образуются мицеллярные системы, проводили с помощью ЯМР 1Н спектроскопии [31]. Если поместить олигопептид в раствор с мицелярными образованиями на основе додецилсульфат натрия [15,16], то можно ожидать образование комплекса протеин - мицелла, молекулярная масса которого будет существенно больше, чем у несвязанной молекулы протеина. При этом протеин из разряда малых молекул, для которого сохраняется условие быстрого движение, переходит в разряд молекул, для которых справедливо условие медленногодвижения, что дает возможность применить метод NOESY спектроскопии ЯМР для установления структуры исследуемого пептида в комплексе с мицеллой. Были исследованы следующие системы: олигопептиды (амилоидные пептиды А(16-22) и А(1-40))– водный раствор с додецил сульфатом натрия (ДСН), который использовался в качестве модели мембраны. При концентрации (ККМ) ДСН в растворе воде большей, чем 4.3 г/л, происходило образование мицелл, что контролировалось с помощью ЯМР 1Н спектроскопии. ЯМР 1Н спектр гептапептида Aβ16-22 (NAc-Lys-Leu-Val-Phe-Phe-Ala-Glu-NH2), растворенного с мицеллами на основе додецил сульфата натрия, представлен на рисунке 2.2 (химические сдвиги приведены в таблице 2.1). Большие по интенсивности сигналы на спектре принадлежат сигналам протонов от ДСН. Отнесение сигналов в спектре ЯМР 1Н гептапептида Aβ16-22 (NAc-Lys-Leu-ValPhe-Phe-Ala-Glu-NH2) в растворе Н2О+D2О с ДСН сделано на основании двумерных экспериментов ЯМР 1Н-1H TOCSY (рисунок 2.3, как пример), данных предыдущих исследований и сведений из литературы (таблица 2.1). 21 Рисунок 2.2 - ЯМР 1Н (500 МГц) спектр гептапептида NAc-Lys-Leu-Val-Phe-Phe-AlaGlu-NH2 в растворе Н2О + D2O с додецилсульфатом натрия, находящемся в мицеллярном состоянии. Т 293 К, 0 м.д. соответствует сигналу ЯМР 1Н ТМС. Рисунок 2.3 - Область двумерного 1Н-1H TOCSY спектра ЯМР (7.2-8.5 м.д.) гептапептида NAc-Lys-Leu-Val-Phe-Phe-Ala-Glu-NH2 в растворе Н2О+ D2O с додецилсульфатом натрия, находящемся в мицеллярном состоянии. Т 293 К, 0 м.д. соответствует сигналу ЯМР 1Н ТМС. 22 Таблица 2.1. ЯМР 1H химические сдвиги (Н, м.д., относительно ТМС) гептапептида NAc-Lys-Leu-Val-Phe-Phe-Ala-Glu-NH2 в растворе Н2О + D2O с додецил сульфатом натрия, находящемся в мицеллярном состоянии. Т 293 К (в скобках, приведены данные ХС в растворе боратного буфера из таблицы 2.1, отчет по Проекту, 1 этап). Остаток Химические сдвиги, м.д. NH CαH CβH CγH прочие Lys16 7.41 4.10 (4.26) 1.75 (3.00) 1.45 1.65 (δ), 2.94 (ε) Leu17 8.02 4.30 (4.36) 1.72 (1.60) 1.52 0.93(δ1), 0.86 (δ2) Val18 7.57 3.80 (4.06) 1.97 (1.94) 0.82(γ1), 0.70 (γ2) Phe19 7.64 4.23 (4.20) 2.84;2.75 (2.07;2.28) 6.86(2/6);7.1 2(3/5) Phe20 7.52 4.44 (4.20) 3.17;2.93 (2.07;2.28) 7.2 (2/6), 6.95 (3/5) Ala21 7.85 4.15 (4.15) 1.36 (1.34) Glu22 8.01 4.13 (4.35) 2.02;1.89 (2.38;2.11) 2.27 Сравнивая химические сдвиги гептапептида в растворе Н2О + D2O с додецилсульфатом натрия, находящемся в мицеллярном состоянии (таблица 2.1) и в растворе боратного буфера (Na2B4O7·10H2O+D2O), можно видеть изменения этих величин. Отличие химических сдвигов в этих растворах возможно обусловлено изменением конформации молекулы гептапептида и образованием комплекса гептапептид - мицеллы на основе додецилсульфата натрия. Кроме того, в двумерном ЯМР 1Н NOESY спектре гептапептида наблюдаются кросс-пики положительного знака, что характерно для молекул, подпадающих под условие медленного движения. Все выше приведенные факты подтверждают образование комплекса гептапептид – мицеллы на основе додецилсульфата натрия. 23 Рисунок 2.4 - Область двумерного ЯМР 1Н-1H NOESY спектра (6.4-9.0 м.д.) гептапептида NAc-Lys-Leu-Val-Phe-Phe-Ala-Glu-NH2 в растворе Н2О+ D2O с додецилсульфатом натрия, находящемся в мицеллярном состоянии. Т 293 К, 0 м.д. соответствует сигналу ЯМР 1Н ТМС. Время смешивания τm 0,4 с. Для определения межпротонных расстояний, напрямую характеризующих пространственную геометрию гептапептида Aβ16-22 (NAc-Lys-Leu-Val-Phe-Phe-AlaGlu-NH2) в комплексе в исследованном растворе, записывали двумерные ЯМР NOESY спектры (рисунок 2.4) с вариацией времени смешивания τm. Наблюдались кросс-пики в двумерных ЯМР спектрах NOESY между сигналами протонов исследуемого соединения, относящихся к различным аминокислотным фрагментам. Анализируя и обрабатывая интегральные интенсивности данных кросс-пиков, используя методику, изложенную в работе [27], можно получить межпротонные расстояния в гептапептиде в комплексе: олигопептид – мицеллы на основе ДСН. Одномерный 1Н (500 МГц) спектр ЯМР пептида H-Asp-Ala-Glu-Phe-Arg-HisAsp-Ser-Gly-Try--Glu-Val-His-His-Gln-Lys-Leu-Val-Phe-Phe-Ala-Glu-Asp-Val-Gly-Ser24 Asn-Lys-Gly-Ala-Ile-Ile-Gly-Leu-Met-Val-Gly-Gly-Val-Val-OH в растворе фосфатного буфера (pH=7.4) и додецил сульфата натрия представлен на рисунке 2.5. Рисунок 2.5 - Одномерный 1Н (500 МГц) спектр ЯМР пептида Aβ1-40 в растворе фосфатного буфера и додецил сульфата натрия при Т= 293 К. Отнесение сигналов в 1Н ЯМР спектре пептида Aβ1-40 (H-Asp-Ala-Glu-PheArg-His-Asp-Ser-Gly-Try--Glu-Val-His-His-Gln-Lys-Leu-Val-Phe-Phe-Ala-Glu-Asp-ValGly-Ser-Asn-Lys-Gly-Ala-Ile-Ile-Gly-Leu-Met-Val-Gly-Gly-Val-Val-OH) фосфатного буфера (NaH2PO4·2H2O/ Na2HPO4·2H2O +D2O) при в растворе pH=7.4 в присутствии 10 мМ додецил сульфата натрия было проведено с помощью двумерной ЯМР 1Н-1H TOCSY спектроскопии. На рисунке 2.6 приведен двумерный ЯМР 1 Н-1H TOCSY спектр бета- амилоида Aβ1-40 в растворе фосфатного буфера и додецил сульфата натрия при Т= 293 К. 25 Рисунок 2.6 - Двумерный 1Н-1H TOCSY спектр ЯМР бета-амилоида Aβ1-40 в растворе фосфатного буфера и додецил сульфата натрия при Т= 293 К. Анализируя данные о расположении кросс-пиков в двумерном 1Н-1H TOCSY спектре и используя литературные данные о химических сдвигах протонов в аминокислотных фрагментах бета-амилоида Aβ1-40 были отнесены сигналы протонов CH, CH2 и CH3 групп аминокислот исследуемого пептида. Полученные данные о 1H ЯМР химических сдвигах исследуемого пептида приведены в таблице 2.2. 26 Таблица 2.2. ЯМР 1H химические сдвиги (Н, м.д., относительно ТМС) бетаамилоида Aβ1-40 в растворе фосфатного буфера с додецил сульфатом натрия, находящемся в мицеллярном состоянии. Т 293 К. Остаток Химические сдвиги CγH прочие CαH CβH Asp1 4,18 2.93, 2.76 Ala2 4,13 1,15 Glu3 4,12 1.87, 1.80 Phe4 4,39 2.87, 2.71 Arg5 4,14 1,46 His6 3,60 2.45, 2.35 Asp7 4,17 2.94, 2.81 Ser8 4,27 3,73 Gly9 3,76 2,19 Tyr10 4,39 2,9 Glu11 4,11 1.77, 1.72 2,05 Val12 3,88 1,89 0,73 His13 3,58 2.44, 2.33 His14 3,53 2.43, 2.29 Gln15 3,43 1.76, 1.71 2,36 6.87, 6.55 Lys16 4,17 1,50 1,30 2.79(e), 1.25(d) Leu17 4,00 1,41 1,20 0.71, 0.68 Val18 3,90 1,86 0,73 Phe19 4,28 2.95, 2.78 7.48, 6.73 Phe20 4,26 2.95, 2.78 7.50, 6.76 Ala21 4,09 1,22 Glu22 4,11 1.73, 1.69 Asp23 4,16 2.93, 2.81 Val24 3,98 1,93 Gly25 3,76 Ser26 4,34 3,71 Asn27 4,17 2.96, 2.78 Lys28 4,17 1,50 2,11 7.52, 6.76 1.59, 1.35 2.99(d) 1,99 0,80 6,95 1,30 27 2.79(e), 1.25(d) Продолжение таблицы 2.2. Gly29 3,80 Ala30 4,16 1,21 Ile31 3,99 1,71 1.30, 0.73 Ile32 4,01 1,71 1.30, 0.77 Gly33 3,74 Leu34 4,06 1,43 1,21 Met35 3,55 2.44, 2.23 2.8, 2.75 Val36 4,01 1,93 0,78 Gly37 3,84 Gly38 3,82 Val39 4,00 1,87 0,73 Val40 4,00 2,01 0,78 0.75, 0.70 Сравнивая химические сдвиги бета-амилоида Aβ1-40 в растворе фосфатного буфера с додецилсульфатом натрия, находящемся в мицеллярном состоянии и в растворе фосфатного буфера (таблицы 2.3, отчет по Проекту, 1 этап), можно видеть изменения этих величин. Отличие химических сдвигов в этих растворах обусловлено, так же как и в случае с гептапептидом Aβ16-22, изменением конформации молекулы бета-амилоида Aβ1-40 и образованием комплекса бетаамилоид - мицеллы на основе додецилсульфата натрия. Для определения межпротонных расстояний, напрямую характеризующих пространственную геометрию бета-амилоида Aβ1-40 в растворе, записывали двумерные ЯМР NOESY спектры (рисунок 2.7) с вариацией времени смешивания τm (50мс, 75 мс, 100 мс, 200 мс). Наблюдались кросс-пики в двумерных ЯМР спектрах NOESY между сигналами протонов исследуемого соединения, относящихся к различным аминокислотным фрагментам. 28 Рисунок 2.7 - Область двумерного 1Н-1H NOESY (τm=50 мс) спектра ЯМР бетаамилоида Aβ1-40 в растворе фосфатного буфера и додецил сульфата натрия, находящемся в мицеллярном состоянии. Т= 293 К. 29 3. ОБРАБОТКА ЭКСПЕРИМЕНТОВ РЕЗУЛЬТАТОВ И ДВУМЕРНЫХ ОПРЕДЕЛЕНИЕ ЯМР NOESY КОЛИЧЕСТВЕННОЙ ИНФОРМАЦИИ О МЕЖПРОТОННЫХ РАССТОЯНИЯХ В КОМПЛЕКСАХ ПЕПТИД - ПОВЕРХНОСТЬ ЛИПИДНЫХ МЕМБРАН Анализируя и обрабатывая интегральные интенсивности кросс-пиков в двумерных ЯМР спектрах NOESY [27], определены межпротонные расстояния в исследованных олигопептидах в комплексах: олигопептид – мицеллы на основе додецил сульфата натрия. Отметим, поскольку наблюдались кросс-пики в двумерных ЯМР спектрах NOESY между сигналами протонов исследуемого соединения, относящихся к различным аминокислотным фрагментам, становится возможным определить пространственное строение основной цепи олигопептидов. Данные о пространственном строении гептапептида Aβ16-22 в растворе Н2О + D2O в комплексе пептид – мицеллы приведены в таблице 3.1. В качестве калибровочной интенсивности кросс-пиков был выбран кросс-пик в ароматическом кольце фенилаланина Phe19(2/6)–Phe19(3/5) на основании того, что протоны в нём принадлежат ароматическому кольцу и не могут быть изменены при изменении внешних условий. Расстояние между этими протонами определяли различными методами и приняты равными 2.49 Å. Аналогичным образом были получены межпротонные расстояния для бетаамилоида Aβ1-40 в растворе фосфатного буфера в комплексе пептид – мицеллы на основе додецилсульфата натрия. Данные приведены в таблице 3.2. В качестве калибровочной интенсивности кросс-пиков была выбрана интенсивность кросс-пика в ароматическом кольце фенилаланина Phe4(2/6)–Phe4(3/5). Расстояние между этими протонами определяли различными методами и приняты равным 2.49 Å. 30 Таблица 3.1. Экспериментально определенные межпротонные расстояния для гептапептида NAc-Lys-Leu-Val-Phe-Phe-Ala-Glu-NH2 в растворе Н2О+ D2O в комплексе пептид – мицеллы на основе додецилсульфата натрия. Знаком (*) обозначено калибровочное расстояние. Пара протонов Межпротонное расстояние r, Å, NOESY F19(2/6)/F19(3/5) 2,49* E22(NH)/A21(NH) 3.08±0.62 F19(2/6)/F19(NH) 3.48±0.70 K16(NH)/K16(a) 3.43±0.69 K16(NH)/E22(b) 2.93±0.59 A21(NH)/A21(a) 2.97±0.59 E22(NH)/E22(b) 4.54±0.91 E22(a)/E22(b) 3.58±0.72 A21(NH)/F20(a) 2.54±0.51 F19(NH)/F19(a) 2.98±0.60 F19(NH)/V18(a) 3.10±0.62 F19(NH)/F19(b) 3.30±0.66 F19(2/6)/F19(a) 3.35±0.67 F20(NH)/F19(a) 3.27±0.65 F19(b)/F19(2/6) 3.60±0.72 V18(NH)/V18(b) 3.02±0.60 V18(a)/V18(NH) 2.80±0.56 F19(b)/F19(a) 2.85±0.57 V18(NH)/V18(g) 3.95±0.79 V18(a)/V18(b) 3.03±0.61 V18(g)/V18(a) 4.98±1.00 F20(NH)/F20(a) 2.45±0.49 F20(2/6)/F20(a) 2.14±0.43 F20(NH)/F20(b) 3.12±0.62 F20(2/6)/F20(b) 2.88±0.58 31 Таблица 3.2. Экспериментально определенные межпротонные расстояния для бетаамилоида Aβ1-40 в растворе фосфатного буфера в комплексе пептид – мицеллы на основе додецилсульфата натрия. Знаком (*) обозначено калибровочное расстояние. Пара протонов Межпротонное расстояние r, Å, NOESY F19(2/6)/F19(3/5) 2,49* D1(b)/K28 (a) 1,73±0,35 R5(g)/R5(g) 1,55±0,31 F4(2/6)/F4(3/5) 2,79±0,56 F20(b)/K28 (a) 2,08±0,42 S26(b)/F20(a) 3,30±0,66 F19(3/5)/F19(2/6) 3,84±0,77 A2(b)/I31(a) 4,31±0,86 M35(g)/M35(a) 2,25±0,45 R5(a)/R5(g) 2,92±0,58 L17(g)/Q15(a) 3,20±0,64 R5(b)/K16, K28 (e) 2,99±0,60 L34(d)/E22(b) 3,59±0,72 A21(b)/A21(a) 3,84±0,77 L17(d)/L17(b) 2,67±0,53 H13(b)/H13(a) 2,58±0,52 Y10(a)/D7(b) 2,49±±0,50 L17(d)/I31(g) 3,12±0,62 H14(b)/H14(a) 2,66±0,53 D1(b)/R5(a) 2,15±0,43 A21(b)/V24(b) 4,39±0,88 F4(a)/F4(b) 2,36±0,47 L34(b)/V24(b) 4,31±0,86 F19(a)/V24(b) 2,86±0,57 Q15(b)/A21(a) 2,80±0,56 32 4. НА ОСНОВАНИИ ДАННЫХ О МЕЖПРОТОННЫХ РАССТОЯНИЯХ ОПИСАНИЕ СТРОЕНИЯ КОМПЛЕКСОВ: ПЕПТИД - ПОВЕРХНОСТЬ ЛИПИДНЫХ МЕМБРАН В РАСТВОРЕ С целью определения пространственного строения гептапептида Aβ 16-22 в растворе Н2О + D2O в комплексе пептид – мицеллы на основе додецилсульфата натрия, проведены расчеты по методу молекулярной механики с использованием программного обеспечения DYNAMO [32]. Эти расчеты позволили однозначно определить конформер, как наиболее выгодную структуру для гептапептида. В качестве исходных экспериментальных данных использовали межпротонные расстояния, полученные из анализа интенсивностей кросс-пиков ЯМР NOESY спектров гептапептида (таблица 2.1). Координаты атомов в pdb формате пространственной структуры Aβ16-22 (NAcLys-Leu-Val-Phe-Phe-Ala-Glu-NH2) в растворе и в комплексе пептид – мицеллы на основе додецилсульфата натрия в H2O+D2O приведены в Приложении Б. Анализируя изменения ЯМР 1Н химических сдвигов протонов гептапептида при переходе от раствора боратного буфера (Na2B4O7·10H2O+D2O) к раствору Н2О+ D2O с додецил сульфатом натрия, находящемся в мицеллярном состоянии, можно на качественном уровне описать пространственное строение комплекса гептапептид NAc-Lys-Leu-Val-Phe-Phe-Ala-Glu-NH2 – мицеллы на основе додецил сульфата натрия, который представлен на рисунке 4.1. Рисунок 4.1. Строение комплекса гептапептид NAc-Lys-Leu-Val-Phe-Phe-Ala-GluNH2 – мицелла на основе додецилсульфата натрия. 33 С целью определения пространственного строения бета-амилоида Aβ1-40 в растворе фосфатного буфера в комплексе мицеллы на основе додецилсульфата натрия, были проведены расчеты по методу молекулярной механики по программе DYNAMO [32]. Эти расчеты позволили однозначно определить структуру, как наиболее выгодную для бета-амилоида Aβ1-40. В качестве исходных экспериментальных данных использовали межпротонные расстояния, полученные из анализа интенсивностей кросс-пиков ЯМР NOESY спектров (таблица 2.2). Координаты атомов в pdb формате пространственной структуры бета-амилоида Aβ1-40 в растворе и в комплексе пептид – мицеллы на основе додецилсульфата натрия приведены в Приложении С. Анализируя изменения ЯМР 1Н химических сдвигов протонов бета-амилоида Aβ1-40 при переходе от раствора к комплексу, можно на качественном уровне описать пространственное строение самого комплекса бета-амилоида Aβ1-40 – мицеллы на основе додецил сульфата натрия, который представлен на рисунке 4.2. Рисунок 4.2. Строение комплекса бета-амилоид Aβ1-40 - мицелла на основе додецилсульфата натрия. 34 ЗАКЛЮЧЕНИЕ Объектами исследований на данном этапе Проекта были: синтетический А(140) пептид нативного типа (H-Asp-Ala-Glu-Phe-Arg-His-Asp-Ser-Gly-Try--Glu-Val-His- His-Gln-Lys-Leu-Val-Phe-Phe-Ala-Glu-Asp-Val-Gly-Ser-Asn-Lys-Gly-Ala-Ile-Ile-GlyLeu-Met-Val-Gly-Gly-Val-Val-OH), его активный фрагмент А(16-22) (NAc-Lys-LeuVal-Phe-Phe-Ala-Glu-NH2). В результате проведенных двумерных ЯМР NOESY экспериментов для синтетического А(1-40) пептида нативного типа и его активного фрагмента А(16-22) в растворе показано, что применение этого метода ЯМР спектроскопии к исследованию олигопептидов, подпадающих под условие быстрого движения неэффективно; в этом случае не наблюдаются, либо наблюдаются слабые по интенсивности кросс-пики в спектрах ЯМР NOESY, что затрудняет получение количественной информации о межпротонных расстояниях; предложено информацию о пространственном строении синтетического А(1-40) пептида и его активного фрагмента А(16-22) в растворах извлекать из таблиц координат атомов этих пептидов (в pdb-формате), определенных в результате анализа величин остаточного диполь-дипольного взаимодействия (см. отчет по Проекту, 3-этап) Проведены двумерные ЯМР NOESY эксперименты для исследуемых комплексов: пептид - поверхность мицелл на основе додецилсульфата натрия в растворе; обработаны результаты двумерных ЯМР NOESY экспериментов и определены межпротонные расстояния в бета-амилоиде А(1-40) и в гептапептиде А(16-22) в этих комплексах. Описано пространственное строение комплексов: пептид - поверхность мицелл на основе додецилсульфата натрия в растворе на основании количественной информации о межпротонных расстояниях в олигопептидах в этих комплексах и качественном рассмотрении изменений химических сдвигов протонов олигопептидов при переходе от растворов к комплексам на основе додецил сульфата натрия. Полученная структурная информация позволит выявить биохимические свойства исследуемых пептидов в комплексах пептид-модель мембраны; выяснить роль мембраны при образовании промежуточных структур агрегации; описать механизм изменения свойств мембраны (проницаемости) при взаимодействии с пептидами, что является последующими задачами Проекта. 35 СПИСОК ИСПОЛЬЗОВАННЫХ ИСТОЧНИКОВ 1. Ross, C. A. Protein aggregation and neurodegenerative diseases [Text] / C. A. Ross, M.A Poirier // Neurodegeneration. - 2004. – V. 6. - P. S10-S17. 2. Rochet, J.-C. Amyloid fibrillogenesis: themes and variations [Text] / J.-C. Rochet, P. T. Lansbury // Current Opinion in Structural Biology. - 2000. –V. 10(1) - P. 60 -68. 3. Gorbenko, G. P. The role of lipid–protein interactions in amyloid-type protein fibril formation [Text] /. G. P. Gorbenko, P. K. J. Kinnunen // Chemistry and Physics of Lipids. - 2006. – V. 141.- P. 72–82. 4. Antzutkin, O. N. Amyloidosis of Alzheimer's A beta peptides: solid-state nuclear magnetic resonance, electron paramagnetic resonance, transmission electron microscopy, scanning transmission electron microscopy and atomic force microscopy studies [Text] / O. N.Antzutkin // Magnetic Resonance in Chemistry. - 2004. – V. 42. - P. 231-246. 5. Narayanan, S. Characterization of chemical exchange between soluble and aggregated states of beta -amyloid by solution-state NMR upon variation of salt conditions [Text] / S. Narayanan, B. Reif // Biochemistry.- 2005. – V. 44 .- P. 1444 1452. 6. Jarvet, J. Reversible random coil to b-sheet transition and the early stage of aggregation of the Ab(12-28) fragment from the Alzheimer peptide [Text] / J. Jarvet, P. Damberg, K. Bodell, L. E. G. Eriksson, A. Gräslund // Journal of the American Chemical Society.- 2000.- V. -122 .- P. 4261 - 4268. 7. Filippov, A. V. Diffusion and aggregation of Alzheimers's Abeta(1-40) peptide in aqueous trifluoroethanol solutions as studied by pulsed field gradient nmr [Text] / A. V. Filippov, A. V. Sulejmanova, O. Antzutkin, G. Gröbner // Applied Magnetic Resonance.- 2005. - V. 29. - P. 439-449. 8. Filippov, A.V. Effect of freezing on amyloid peptide aggregation and selfdiffusion in an aqueous solution [Text] / A. V. Filippov, A.V. Suleimanova, G. Grobner, N. Antsutkin // Colloid Journal. - 2008. – V. 70.- P. 501–506. 9. Bradley, E. K. NMR structural characterization of oligo-N-substituted glycine lead compounds from a combinatorial library [Text] / E. K. Bradley, J. M. Kerr, L. S. 36 Richter, G. M. Figliozzi, D. A. Goff, R. N. Zuckermann, D. C. Spellmeyer, J. M. Blaney // Molecular Diversity. - 1997. - V. 3. - P. 1-15. 10. Anishetty, S. Tripeptide analysis of protein structures [Text] / S. Anishetty, G. Pennathur, R. Anishetty // BMC Structural Biology - 2002. - V. 2. - P. 1472-1507. 11. Ernst, R. R. Principles of Nuclear Magnetic Resonance in One and Two Dimensions [Text] / R. R. Ernst, B. Bodenhausen, A. Wokaun. - Oxford: Oxford University Press, 1987.- 610 p. 12. Van der Ven, Multidimensional NMR in liquids: basic principles and experimental methods [Text] / Van der Ven, J. M. Frank, - N-Y: Wiley-VCH, 1995. - 399 p. 13. Jackman, L. M. Dynamic Nuclear Magnetic Resonance Spectroscopy [Text] / L. M. Jackman, F. A.Cotton, N-Y: Academic Press, 1975. - 660 p. 14. Sandstrom, J. Dynamic NMR Spectroscopy [Text] / J. Sandstrom // London: Academic Press, 1982. - 226 p. 15. Application of dynamic NMR spectroscopy to organic chemistry [Text] / M.Oki . - N-Y: Wiley-VCH, 1985. - 423 p. 16. Merrifield, R.B. Solid phase peptide synthesis. I. The synthesis of a tetrapeptide [Text] / R.B. Merrifield // J. Am. Chem. Soc. - 1963. - Vol.85. – P.2149. 17. Jones, J. Amino Acid and Peptide Synthesis [Text] / J. Jones - New York: Oxford University Press, 2002. - P.92. 18. Filippov, A. Synthesis and aggregation studies on amyloid oligomers of Alzheimer’s Abeta peptides [Text] / A.Filippov // Licentiate of Technology Thesis. Department of Chemical Technology and Geosciences. Division of Chemical Engineering. Lulea University of Technology, 2010. - 26 p. ISSN: 1402-1757 ISBN 97891-7439-069-8. Lulea University of Technology 2010. 19. Li, T. Gene transfer with echo-enhanced contrast agents: Comparison between Albunex, Optison, and Levovist in mice—Initial results [Text] / T. Li, K. Tachibana, M. Kuroki // Radiology. - 2003. - V. – 229. – P. 423–428. 20. Wolf, M. Stabilisation and determination of the biological activity of Lasparaginase in poly(D,L-lactideco-glycolide) nanospheres [Text] / M. Wolf, M. Wirth, F. Pittner, F. Gabor // International Journal of Pharmaceutics. - 2003. – V. 256. – P. 141– 152. 37 21. Mason, T. J. Practical sonochemistry: Uses and applications of ultrasound [Text] / T. J. Mason, , D. Peters. – Chichester, UK.: Horwood Publishing, 2002. – 543 p. 22. Soto, C. The alpha-helical to beta-sheet transition in the amino-terminal fragment of the amyloid beta-peptide modulates amyloid formation [Text] / C. Soto, E. Castano, B. Frangione, N. Inestrosa // Journal of Biological Chemistry. – 1995. – V. 271. – P. 3963–3967. 23. Lomas, D. A. Serpinopathies and the conformational dementias [Text] / D. A. Lomas, R. W. Carrell // Nature Reviews Genetics. – 2002. - V. 3 (10). – P. 759. 24. Stefani, M. Protein aggregation and aggregate toxicity: new insights into protein folding, misfolding diseases and biological evolution [Text] /M .Stefani, C. M. Dobson // Journal of Molecular Medicine. - 2003. – V. 81. – P. 678-699. 25. Berger, S. 200 and More NMR Experiments [Text] / S. Berger, S. Braun. – Weinheim: Wiley-VCH, 2004. - 810 p. 26. Friebolin, H. Basic one- and two dimensional NMR spectroscopy [Text] / H. Friebolin // Weinheim; Basel; New York: Wiley-VCH, 1991. - P. 344 27. Gadiev, T.A. Analysis of the spatial structure of calixarenes in solutions by 2-D NMR (NOESY) spectroscopy. [Text] / T.A. Gadiev, B.I. Khairutdinov, I.S. Antipin, V.V. Klochkov// Applied Magnetic Resonance. – 2006. – V. 30, N 2. - P. 65-73. 28. Coles, M. Solution Structure of Amyloid β-Peptide (1-40) in a Water- Micelle Enviroment. Is the Membrane-Spanning Domain Where We Think It Is? [Text] /M. Coles, W. Bicknell, A. Watson, D.P. Fairlie, D.J. Craik // Biochemistry. – 1998. – V. 37. – P.11064-11077. 29. Motta, A. Solution conformation of salmon calcitonin in sodium dodecyl sulfate micelles as determined by two-dimensional NMR and distance geometry calculations [Text] / A. Motta, A. Pastore, N.A. Goud, M.A. Castiglione // Biochemistry — 1991. — V. 30. — P.10444-10450. 30. Wang, G. Solution structure of the N-terminal amphitropic domain of Escherichia coli glucose-specific enzyme IIA in membrane-mimetic micelles [Text] / G. Wang, P. Keifer, A. Peterkofsky // Protein Science. – 2003. - V. 12. – P. 1087–1096. 31. Блохин, Д.С. Пространственное строение декапептида Val-Ile-Lys-Lys- Ser-Thr-Ala-Leu-Leu-Gly в комплексе протеин – мицеллы додецилсульфата натрия [Текст] /Д.С. Блохин, С.В. Ефимов, А.В. Клочков, А.Р. Юльметов, А.В. Филиппов, А.В. Аганов, В.В. Клочков // Ученые Записки Казанского Университета. - 2011. - Т. 153, Серия Естественные науки, книга 1. - С. 59-70. 38 32. Delaglio, F. NMRPipe: A multidimensional spectral processing system based on UNIX pipes [Text] / F. Delaglio, S. Grzesiek, G. Vuister, G. Zhu, J. Pfeifer, A. Bax// J. Biomol. NMR. – 1995. – V. 6. – P. 277-293. 39 ПРИЛОЖЕНИЕ А. Список работ, выполненных в рамках проекта, опубликованных в 2011 году. 1. Klochkov, V.V. A spatial structure of tripeptides glycylglycyl-L-histidine and glycylglycyl-L-tyrosine based on residual dipolar couplings and quantum-chemical computations [Text] /V.V.Klochkov, A.V.Klochkov, M.N.Schamsutdinov, S.V.Efimov, A.A.Krutikov, E.M.Gilyazetdinov, Y.Y. Zyavkina, V.G. Shtyrlin// Mendeleev Communications. – 2011 - Vol. 21, N 2. - P. 72-74. 2. Блохин, Д.С. Пространственное строение декапептида Val-Ile-Lys-Lys-Ser-ThrAla-Leu-Leu-Gly в комплексе протеин – мицеллы додецилсульфата натрия [Текст] /Д.С.Блохин, С.В.Ефимов, А.В.Клочков, А.Р.Юльметов, А.В.Филиппов, А.В.Аганов, В.В.Клочков // Ученые Записки Казанского Университета. - 2011. - Т. 153, Серия Естественные науки, книга 1. - С. 59-70. 3. Сахарова, А.В. Подвижность молекул и диаграмма состояния системы глицерилмоноолеат-вода по данным ЯМР [Текст] /А.В.Сахарова, А.В.Филиппов, Б.В.Мунавиров, В.Д.Скирда //Журнал физической химии. - 2011. – Т .85, N4. – С. 649-659. 4. Filippov, А. Interaction of polyacrylic acid oligomer with dimyristoylphosphatidylcholine bilayers [Text]/ A. Filippov, B. Munavirov, T. Sparrman, V. Ishmuhametova, M. Rudakova, P. Shriram, S. Tavelin// Langmuir. -2011. - V.27, N.7. P.3754-3761. 5. Blokhin, D.S. Spatial structure of the decapeptide Val-Ile-Lys-Lys-Ser-Thr-Ala-LeuLeu-Gly in water and in a complex with sodium dodecyl sulfate micelles [Text] /D.S. Blokhin, S.V. Efimov, A.V. Klochkov, A.R. Yulmetov, A.V. Filippov, O.N.Antzutkin, A.V. Aganov, V.V. Klochkov// Applied Magnetic Resonance. – 2011. – Vol. 41. – In press. 6. Усачев, К.С. Пространственное строение гептапептида Aβ16-22 в растворе и в комплексе гептапептид – модель биологической мембраны [Текст] / К.С. Усачев, А.Р. Юльметов, А.В. Филиппов, О.Н. Анцуткин, С. Афонин, А.В. Аганов, В.В. Клочков // Ученые Записки Казанского Университета. - 2011. - Т. 153, Серия Естественные науки, книга 3. – В печати. 7. Блохин, Д.С. Пространственное строение некоторых олигопептидов в растворе и комплексе: олигопептид – модель биологической мембраны [Текст] / Д.С.Блохин, 40 С.В.Ефимов, А.В.Клочков, И.З.Рахматуллин, К.С.Усачев, А.Р.Юльметов, А.В.Филиппов, А.В.Аганов, В.В.Клочков /Тез. докл. V Всероссийской конференции “Новые достижения ЯМР в структурных исследованиях” Казань, РФ. Апрель 2011 г., С. 22-23. 8. Блохин, Д.С. Пространственное строение декапептида Val-Ile-Lys-Lys-Ser-ThrAla-Leu-Leu-Gly в водном растворе и в комплексе протеин – мицеллы додецилсульфата натрия [Текст] / Д.С. Блохин, С.В. Ефимов, А.В. Клочков, А.Р. Юльметов, А.В. Филиппов, А.В. Аганов, В.В. Клочков//Тез. докл. V Всероссийской конференции “Новые достижения ЯМР в структурных исследованиях” Казань, РФ. Апрель 2011 г., С. 86-87. 9. Рахматуллин, И.З. Пространственное строение усиливающего ВИЧ гептапептида Glu-Ile-Leu-Asn-His-Met-Lys в растворе и комплексе: гептапептид – модель биологической мембраны [Текст] / И.З. Рахматуллин, Д.С.Блохин, А.Р.Юльметов, А.В.Филиппов, О.Н.Анцуткин, Всероссийской конференции А.В.Аганов, “Новые В.В.Клочков достижения ЯМР /Тез. в докл. V структурных исследованиях” Казань, РФ. Апрель 2011 г., С. 131-133. 10. Усачев, К.С. Определение структуры бета-амилоида А(10-35) (активного фрагмента бета-амилоида А(1-40)) в растворе методами ЯМР спектроскопии и анализом констант остаточного диполь-дипольного взаимодействия [Текст] / К.С. Усачев, А.В. Филиппов, А.В. Аганов, В.В.Клочков /Тез. докл. V Всероссийской конференции “Новые достижения ЯМР в структурных исследованиях” Казань, РФ. Апрель 2011 г., С. 141-142. 41 ПРИЛОЖЕНИЕ Б. Координаты атомов в pdb-формате бэта-амилоида Aβ16-22 в растворе Н2О+ D2O с додецил сульфатом натрия, находящемся в мицеллярном состоянии, рассчитанные в программе DYNAMO. В качестве входных данных использовались экспериментально полученные значения расстояний из эксперимента NOESY. № атома 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 Обозначение Аминокислота C CA O HA1 HA2 HA3 C CA CB CD CE CG HA HB1 HB2 HD1 HD2 HE1 HE2 HG1 HG2 HN HZ1 HZ2 HZ3 N NZ O C CA CB CD1 CD2 CG HA HB1 HB2 HD11 № аминокиcлоты NAC NAC NAC NAC NAC NAC LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 3 3 3 3 3 3 3 3 3 3 42 x y z 10.884 10.732 9.954 11.702 10.086 10.301 13.699 12.258 11.907 12.339 13.063 12.697 11.601 10.852 12.159 11.275 12.643 12.826 12.748 13.756 12.450 12.856 14.737 14.895 15.004 12.080 14.535 14.320 16.548 15.597 15.703 16.038 13.729 15.097 15.863 16.735 15.161 15.965 11.874 13.252 11.467 13.618 13.184 13.930 9.504 9.977 10.019 11.185 12.358 11.140 9.278 10.207 9.072 11.313 10.259 13.270 12.441 10.951 12.084 11.766 11.125 12.381 12.714 11.297 12.127 9.018 9.874 9.242 7.701 7.172 7.647 7.028 9.570 7.417 7.361 8.167 2.883 3.507 2.511 3.810 4.370 2.786 2.411 2.240 0.754 -1.410 -2.073 0.076 2.737 0.640 0.298 -1.522 -1.883 -1.549 -3.104 0.184 0.535 3.207 -2.215 -1.082 -2.744 2.835 -2.024 1.475 2.903 3.914 3.840 6.305 5.422 5.094 4.896 3.746 2.967 6.725 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 HD12 HD13 HD21 HD22 HD23 HG HN N O C CA CB CG1 CG2 HA HB HG11 HG12 HG13 HG21 HG22 HG23 HN N O C CA CB CD1 CD2 CE1 CE2 CG CZ HA HB1 HB2 HD1 HD2 HE1 HE2 HN HZ N O C LEU LEU LEU LEU LEU LEU LEU LEU LEU VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE 3 3 3 3 3 3 3 3 3 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 6 43 17.060 15.762 13.859 13.235 13.125 14.963 13.682 14.226 16.160 19.894 18.813 19.511 18.511 20.091 18.356 20.327 17.978 19.032 17.816 20.904 19.317 20.462 18.065 17.797 20.121 22.325 21.562 20.952 18.507 19.049 17.127 17.672 19.474 16.711 22.248 21.075 21.446 18.821 19.779 16.400 17.355 20.283 15.662 20.529 23.618 21.351 6.995 6.452 8.652 7.043 7.683 5.984 10.053 9.655 10.725 8.887 9.943 11.179 12.327 10.851 10.226 11.497 12.449 13.248 12.104 10.151 10.429 11.761 8.739 9.435 7.995 8.625 8.067 6.680 6.539 7.240 6.702 7.381 6.819 7.109 7.948 6.005 6.235 6.215 7.442 6.497 7.700 9.748 7.170 9.012 8.709 10.556 5.999 7.057 5.806 6.168 4.529 4.885 4.343 3.626 2.110 1.720 2.011 2.606 2.740 3.989 1.088 1.947 1.807 2.970 3.532 3.884 4.604 4.453 3.589 2.929 2.536 -0.991 0.167 -0.171 0.537 -1.747 0.263 -2.027 -0.477 -1.025 1.015 0.693 -1.008 1.512 -2.508 1.016 -3.013 -0.034 -1.263 0.557 -0.957 -3.968 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 CA CB CD1 CD2 CE1 CE2 CG CZ HA HB1 HB2 HD1 HD2 HE1 HE2 HN HZ N O C CA CB HA HB1 HB2 HB3 HN N O C CA CB CD CG HA HB1 HB2 HG1 HG2 HN N O OE1 OE2 HN1 HN2 PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE ALA ALA ALA ALA ALA ALA ALA ALA ALA ALA GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU NH2 NH2 6 6 6 6 6 6 6 6 6 6 6 6 6 6 6 6 6 6 6 7 7 7 7 7 7 7 7 7 7 8 8 8 8 8 8 8 8 8 8 8 8 8 8 8 9 9 44 22.280 22.758 20.555 21.837 19.590 20.862 21.688 19.736 23.123 23.675 22.929 20.440 22.701 18.722 20.961 20.703 18.938 21.609 21.489 19.518 19.182 17.889 18.959 17.965 17.592 17.043 20.001 20.236 19.038 23.215 21.241 20.609 18.582 19.123 20.792 20.879 21.193 18.907 18.694 21.012 20.574 23.447 18.383 18.349 23.337 23.570 9.643 8.535 7.643 6.216 6.640 5.223 7.427 5.438 10.216 8.092 8.982 8.599 6.052 6.801 4.259 8.977 4.667 9.049 10.894 13.217 11.806 11.590 11.573 11.916 10.580 12.255 10.653 10.945 14.235 14.761 14.990 15.498 15.948 15.258 15.624 16.440 14.877 14.063 15.513 12.737 13.570 14.317 15.241 17.056 14.467 16.341 -3.258 -4.176 -5.136 -3.634 -5.278 -3.769 -4.308 -4.587 -2.897 -3.813 -5.148 -5.669 -2.996 -5.910 -3.231 -1.994 -4.637 -2.036 -5.106 -3.972 -3.936 -3.103 -4.827 -2.105 -3.205 -3.532 -2.316 -3.300 -4.518 -2.853 -2.968 -1.400 -0.085 -1.291 -3.710 -1.210 -0.534 -1.335 -2.271 -2.554 -3.207 -2.499 0.885 -0.285 -2.249 -2.268 131 132 HN3 N NH2 NH2 9 9 45 24.073 24.180 15.938 14.284 -4.186 -3.722 ПРИЛОЖЕНИЕ В. Координаты атомов в pdb-формате бэта-амилоида Aβ1-40 в растворе с мицеллами додецил сульфата натрия рассчитанные в программе DYNAMO. В качестве входных данных использовались экспериментально полученные значения расстояний из эксперимента NOESY. № № аминоОбозначение Аминокислота атома ксилоты 1 C ASP 1 2 CA ASP 1 3 CB ASP 1 4 CG ASP 1 5 HA ASP 1 6 HB1 ASP 1 7 HB2 ASP 1 8 N ASP 1 9 O ASP 1 10 OD1 ASP 1 11 OD2 ASP 1 12 HT1 ASP 1 13 HT2 ASP 1 14 HT3 ASP 1 15 C ALA 2 16 CA ALA 2 17 CB ALA 2 18 HA ALA 2 19 HB1 ALA 2 20 HB2 ALA 2 21 HB3 ALA 2 22 HN ALA 2 23 N ALA 2 24 O ALA 2 25 C GLU 3 26 CA GLU 3 27 CB GLU 3 28 CD GLU 3 29 CG GLU 3 30 HA GLU 3 31 HB1 GLU 3 32 HB2 GLU 3 33 HG1 GLU 3 34 HG2 GLU 3 35 HN GLU 3 36 N GLU 3 37 O GLU 3 46 x y z 72.776 74.126 73.910 75.265 74.625 73.398 73.315 74.961 72.110 75.279 76.264 75.950 74.888 74.642 70.731 71.102 71.192 70.334 71.453 70.238 71.949 72.946 72.376 69.834 71.651 71.150 71.834 72.100 71.370 70.084 71.574 72.905 71.592 70.304 72.127 71.421 70.859 10.931 10.235 8.729 8.017 10.612 8.537 8.360 10.496 10.805 6.810 8.691 10.610 9.696 11.367 12.856 12.376 13.562 11.704 13.214 14.067 14.247 11.733 11.667 12.301 13.463 14.422 15.780 18.062 16.723 14.552 16.200 15.653 16.284 16.884 14.289 13.885 12.779 -4.832 -4.973 -5.135 -5.145 -5.852 -6.066 -4.312 -3.806 -3.805 -4.967 -5.331 -4.105 -3.142 -3.338 -7.236 -5.837 -4.890 -5.483 -3.901 -4.853 -5.241 -6.658 -5.864 -7.872 -10.119 -9.045 -9.210 -8.225 -8.098 -9.160 -10.170 -9.150 -7.137 -8.184 -7.169 -7.716 -10.768 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 OE1 OE2 C CA CB CD1 CD2 CE1 CE2 CG CZ HA HB1 HB2 HD1 HD2 HE1 HE2 HN HZ N O C CA CB CD CG CZ HA HB1 HB2 HD1 HD2 HE HG1 HG2 HH11 HH12 HH21 HH22 HN N NE NH1 NH2 O GLU GLU PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG ARG 3 3 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 47 72.985 71.761 73.516 73.555 75.004 74.972 75.109 74.995 75.132 75.029 75.075 72.993 75.464 75.549 74.911 75.152 74.951 75.193 73.552 75.071 72.967 74.066 74.151 72.766 71.907 69.697 70.497 67.554 72.299 71.852 72.349 69.616 70.201 68.024 70.559 70.005 67.780 66.746 67.146 66.387 72.445 72.866 68.334 67.344 66.985 74.295 18.309 18.816 11.079 12.528 12.938 14.416 15.462 15.691 16.737 14.301 16.852 12.610 12.218 12.971 13.529 15.374 15.779 17.625 13.978 17.828 13.412 10.191 8.873 9.495 8.621 8.452 9.209 9.087 9.546 7.622 8.588 7.413 8.528 9.403 10.251 9.115 7.147 8.040 11.038 10.243 11.589 10.844 9.045 8.007 10.210 7.651 -7.423 -9.122 -10.829 -11.307 -11.578 -13.632 -11.425 -14.237 -12.031 -12.225 -13.437 -12.225 -12.238 -10.646 -14.244 -10.349 -15.313 -11.418 -9.760 -13.899 -10.306 -11.479 -8.998 -9.147 -10.062 -11.216 -10.154 -10.281 -8.175 -9.658 -11.047 -10.933 -12.168 -12.184 -10.428 -9.196 -9.843 -8.779 -10.475 -9.136 -9.216 -9.694 -11.326 -9.580 -9.937 -9.039 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 C CA CB CD2 CE1 CG HA HB1 HB2 HD1 HD2 HE1 HN N ND1 NE2 O C CA CB CG HA HB1 HB2 HN N O OD1 OD2 C CA CB HA HB1 HB2 HG HN N O OG C CA HA1 HA2 HN N HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS ASP ASP ASP ASP ASP ASP ASP ASP ASP ASP ASP ASP SER SER SER SER SER SER SER SER SER SER SER GLY GLY GLY GLY GLY GLY 6 6 6 6 6 6 6 6 6 6 6 6 6 6 6 6 6 7 7 7 7 7 7 7 7 7 7 7 7 8 8 8 8 8 8 8 8 8 8 8 9 9 9 9 9 9 48 77.471 76.532 77.011 79.476 80.108 78.402 76.548 77.014 76.345 78.294 79.513 80.703 74.997 75.167 78.828 80.552 77.802 78.065 78.800 79.379 80.215 79.611 80.005 78.572 77.604 77.899 77.056 80.052 81.005 78.242 77.947 78.471 76.878 79.524 78.339 77.875 79.376 78.570 78.910 77.745 79.403 77.966 77.317 77.722 77.218 77.744 10.355 9.219 8.589 7.974 7.111 8.050 8.467 9.337 7.784 7.396 8.403 6.643 10.676 9.711 7.493 7.381 10.514 13.382 12.258 12.789 11.695 11.912 13.645 13.078 10.972 11.144 13.888 11.497 11.075 16.197 14.837 14.809 14.683 15.060 13.824 16.618 13.332 13.770 17.033 15.747 17.834 17.680 17.731 18.490 15.719 16.419 -8.277 -8.671 -9.980 -10.650 -8.765 -9.798 -7.897 -10.760 -10.256 -7.786 -11.641 -7.995 -8.798 -8.824 -8.602 -9.995 -7.102 -8.266 -8.990 -10.303 -10.971 -8.367 -10.102 -10.961 -10.176 -9.258 -8.757 -12.164 -10.279 -6.945 -6.322 -4.886 -6.305 -4.883 -4.471 -4.486 -6.755 -7.099 -6.337 -4.103 -9.296 -8.840 -9.702 -8.169 -8.597 -8.157 130 131 132 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148 149 150 151 152 153 154 155 156 157 158 159 160 161 162 163 164 165 166 167 168 169 170 171 172 173 174 175 O C CA CB CD1 CD2 CE1 CE2 CG CZ HA HB1 HB2 HD1 HD2 HE1 HE2 HH HN N O OH C CA CB CD CG HA HB1 HB2 HG1 HG2 HN N O OE1 OE2 C CA CB CG1 CG2 HA HB HG11 HG12 GLY TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR TYR GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU VAL VAL VAL VAL VAL VAL VAL VAL VAL 9 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 11 11 11 11 11 11 11 11 11 11 11 11 11 11 11 12 12 12 12 12 12 12 12 12 49 79.884 82.303 81.487 81.566 79.484 81.573 78.823 80.912 80.859 79.537 81.901 82.601 81.092 78.937 82.625 77.771 81.458 78.032 79.665 80.096 81.823 78.900 84.476 84.400 83.866 83.321 84.000 85.391 84.434 82.826 83.528 85.047 83.868 83.535 83.568 84.022 82.110 84.529 85.731 85.888 86.250 87.001 86.618 84.958 87.124 85.424 18.951 17.688 16.748 17.213 16.370 15.128 15.438 14.196 16.216 14.350 15.753 17.288 18.179 17.202 15.010 15.556 13.364 13.276 15.849 16.713 18.149 13.461 18.407 18.860 20.292 22.191 20.824 18.839 20.918 20.302 20.137 20.922 17.578 17.972 18.673 23.186 22.218 16.408 17.242 18.417 17.881 19.346 16.629 18.964 17.251 17.305 -9.487 -9.029 -9.910 -11.366 -12.532 -12.801 -13.360 -13.630 -12.252 -13.910 -9.840 -11.664 -11.461 -12.111 -12.587 -13.574 -14.050 -14.384 -9.307 -9.473 -7.993 -14.750 -7.217 -8.671 -8.734 -10.265 -10.163 -9.098 -8.062 -8.443 -10.849 -10.413 -10.273 -9.439 -6.430 -10.195 -10.412 -5.066 -5.495 -4.513 -3.127 -5.005 -5.448 -4.455 -3.202 -2.737 176 177 178 179 180 181 182 183 184 185 186 187 188 189 190 191 192 193 194 195 196 197 198 199 200 201 202 203 204 205 206 207 208 209 210 211 212 213 214 215 216 217 218 219 220 221 HG13 HG21 HG22 HG23 HN N O C CA CB CD2 CE1 CG HA HB1 HB2 HD1 HD2 HE1 HN N ND1 NE2 O C CA CB CD2 CE1 CG HA HB1 HB2 HD1 HD2 HE1 HN N ND1 NE2 O C CA CB CD CG VAL VAL VAL VAL VAL VAL VAL HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS HIS GLN GLN GLN GLN GLN 12 12 12 12 12 12 12 13 13 13 13 13 13 13 13 13 13 13 13 13 13 13 13 13 14 14 14 14 14 14 14 14 14 14 14 14 14 14 14 14 14 15 15 15 15 15 50 86.458 87.944 86.786 87.057 86.253 85.559 83.632 83.303 83.406 83.614 84.975 83.321 83.837 82.485 82.740 84.476 81.846 85.980 82.743 85.250 84.510 82.794 84.647 83.846 81.727 82.444 81.643 83.616 82.814 82.401 83.419 81.492 80.686 81.054 84.228 82.698 82.193 82.606 81.908 83.875 82.312 79.717 79.680 80.231 80.583 80.029 18.707 18.821 19.661 20.212 17.538 17.723 16.905 14.099 14.254 12.881 13.009 13.382 13.046 14.676 12.270 12.404 13.369 12.925 13.566 14.796 15.141 13.286 13.221 13.153 13.687 14.970 16.180 17.913 19.395 17.441 14.989 16.109 16.201 18.366 17.448 20.283 15.758 15.026 18.403 19.147 12.821 12.109 12.378 11.164 10.147 11.359 -2.464 -4.984 -6.016 -4.363 -7.528 -6.861 -4.385 -3.600 -5.114 -5.756 -8.001 -9.350 -7.234 -5.486 -5.590 -5.313 -7.883 -7.615 -10.243 -6.009 -5.467 -8.116 -9.337 -3.030 -1.095 -1.502 -1.019 -0.902 -2.265 -1.333 -1.039 0.048 -1.518 -2.682 -0.143 -2.868 -3.454 -2.951 -2.201 -1.492 -0.445 0.353 -1.148 -1.899 -4.155 -3.403 222 223 224 225 226 227 228 229 230 231 232 233 234 235 236 237 238 239 240 241 242 243 244 245 246 247 248 249 250 251 252 253 254 255 256 257 258 259 260 261 262 263 264 265 266 267 HA HB1 HB2 HE21 HE22 HG1 HG2 HN N NE2 O OE1 C CA CB CD CE CG HA HB1 HB2 HD1 HD2 HE1 HE2 HG1 HG2 HN HZ1 HZ2 HZ3 N NZ O C CA CB CD1 CD2 CG HA HB1 HB2 HD11 HD12 HD13 GLN GLN GLN GLN GLN GLN GLN GLN GLN GLN GLN GLN LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU 15 15 15 15 15 15 15 15 15 15 15 15 16 16 16 16 16 16 16 16 16 16 16 16 16 16 16 16 16 16 16 16 16 16 17 17 17 17 17 17 17 17 17 17 17 17 51 78.655 79.708 81.285 82.336 82.156 80.551 78.975 80.044 80.460 81.792 79.967 79.909 78.058 79.462 80.416 82.820 84.258 81.854 79.793 80.327 80.163 82.608 82.698 84.386 84.941 81.974 82.069 79.267 83.978 85.552 84.286 79.463 84.540 77.890 74.782 75.655 75.129 74.304 76.755 75.415 75.595 75.608 74.062 73.369 74.550 74.209 12.537 10.275 11.058 10.192 8.954 12.248 11.464 14.285 13.565 9.730 10.982 9.574 13.250 13.024 14.056 14.650 14.186 13.715 12.034 14.040 15.039 14.630 15.656 13.191 14.862 13.840 12.691 14.020 13.430 13.993 15.097 13.148 14.176 13.810 11.890 12.979 14.363 15.498 15.362 14.628 12.900 15.122 14.396 14.957 15.739 16.409 -1.450 -1.580 -1.687 -3.222 -4.370 -3.724 -3.614 -1.999 -1.480 -3.894 0.780 -4.988 3.165 2.617 3.222 3.559 3.320 2.827 2.905 4.298 2.854 4.617 3.185 3.718 3.813 1.762 3.097 0.751 1.400 1.700 1.447 1.153 1.856 4.249 2.213 2.831 2.409 0.297 0.718 0.899 3.908 3.011 2.591 0.324 -0.726 0.869 268 269 270 271 272 273 274 275 276 277 278 279 280 281 282 283 284 285 286 287 288 289 290 291 292 293 294 295 296 297 298 299 300 301 302 303 304 305 306 307 308 309 310 311 312 313 HD21 HD22 HD23 HG HN N O C CA CB CG1 CG2 HA HB HG11 HG12 HG13 HG21 HG22 HG23 HN N O C CA CB CD1 CD2 CE1 CE2 CG CZ HA HB1 HB2 HD1 HD2 HE1 HE2 HN HZ N O C CA CB LEU LEU LEU LEU LEU LEU LEU VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE 17 17 17 17 17 17 17 18 18 18 18 18 18 18 18 18 18 18 18 18 18 18 18 19 19 19 19 19 19 19 19 19 19 19 19 19 19 19 19 19 19 19 19 20 20 20 52 76.888 77.565 76.754 75.449 77.241 77.047 73.608 75.265 74.619 74.603 73.750 76.036 73.601 74.185 73.563 74.273 72.809 76.634 76.455 76.029 76.295 75.357 76.457 76.084 74.999 73.866 72.865 71.728 71.851 70.725 72.795 70.787 75.422 74.254 73.444 73.695 71.678 71.884 69.909 73.537 70.019 74.480 76.908 78.442 77.075 76.651 15.611 14.728 16.269 13.688 12.380 12.817 12.117 8.260 9.581 9.658 10.845 9.829 9.596 8.745 10.763 11.768 10.843 8.988 10.740 9.880 10.580 10.704 8.214 5.437 5.877 4.848 4.882 6.093 5.297 6.502 5.284 6.106 5.935 3.887 4.772 4.259 6.398 4.999 7.121 7.279 6.423 7.188 4.582 6.239 5.681 6.248 -0.324 1.039 1.305 0.364 1.557 2.412 1.923 1.843 1.435 -0.107 -0.567 -0.619 1.798 -0.507 -1.628 -0.367 -0.037 -0.302 -0.218 -1.698 2.269 2.016 2.147 1.242 2.220 2.226 4.549 2.752 5.464 3.657 3.197 5.009 3.211 2.527 1.234 4.877 1.718 6.510 3.313 1.596 5.699 1.848 1.564 -0.577 -0.962 -2.320 314 315 316 317 318 319 320 321 322 323 324 325 326 327 328 329 330 331 332 333 334 335 336 337 338 339 340 341 342 343 344 345 346 347 348 349 350 351 352 353 354 355 356 357 358 359 CD1 CD2 CE1 CE2 CG CZ HA HB1 HB2 HD1 HD2 HE1 HE2 HN HZ N O C CA CB HA HB1 HB2 HB3 HN N O C CA CB CD CG HA HB1 HB2 HG1 HG2 HN N O OE1 OE2 C CA CB CG PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE PHE ALA ALA ALA ALA ALA ALA ALA ALA ALA ALA GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU GLU ASP ASP ASP ASP 20 20 20 20 20 20 20 20 20 20 20 20 20 20 20 20 20 21 21 21 21 21 21 21 21 21 21 22 22 22 22 22 22 22 22 22 22 22 22 22 22 22 23 23 23 23 53 77.290 78.658 78.130 79.498 77.553 79.233 77.143 76.725 75.629 76.445 78.860 77.928 80.342 75.402 79.856 76.082 79.406 80.812 79.782 79.546 80.161 80.489 79.094 78.887 77.724 78.524 81.441 81.429 81.951 82.224 83.716 83.246 82.876 81.305 82.617 84.094 82.790 80.462 80.988 81.673 84.860 82.924 79.008 80.166 81.258 82.293 4.432 6.457 3.919 5.943 5.701 4.674 4.606 7.326 5.962 3.854 7.427 2.948 6.521 6.699 4.296 6.023 6.112 6.328 7.427 8.214 8.099 8.587 7.570 9.045 6.929 6.856 5.841 3.524 4.896 4.914 4.013 3.828 5.091 4.731 5.880 3.905 2.855 6.360 5.939 3.083 4.396 3.770 1.202 1.526 0.462 0.624 -3.964 -3.857 -4.976 -4.869 -3.404 -5.429 -1.038 -2.298 -2.524 -3.619 -3.428 -5.405 -5.215 -0.153 -6.226 0.049 -1.332 1.307 1.063 2.345 0.306 2.717 3.087 2.141 1.159 0.597 0.367 2.489 2.909 4.416 6.209 4.765 2.387 4.953 4.697 4.101 4.657 3.279 2.567 1.366 6.395 7.105 4.028 3.088 3.240 2.126 360 361 362 363 364 365 366 367 368 369 370 371 372 373 374 375 376 377 378 379 380 381 382 383 384 385 386 387 388 389 390 391 392 393 394 395 396 397 398 399 400 401 402 403 404 405 HA HB1 HB2 HN N O OD1 OD2 C CA CB CG1 CG2 HA HB HG11 HG12 HG13 HG21 HG22 HG23 HN N O C CA HA1 HA2 HN N O C CA CB HA HB1 HB2 HG HN N O OG C CA CB CG ASP ASP ASP ASP ASP ASP ASP ASP VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL GLY GLY GLY GLY GLY GLY GLY SER SER SER SER SER SER SER SER SER SER SER ASN ASN ASN ASN 23 23 23 23 23 23 23 23 24 24 24 24 24 24 24 24 24 24 24 24 24 24 24 24 25 25 25 25 25 25 25 26 26 26 26 26 26 26 26 26 26 26 27 27 27 27 54 79.803 80.815 81.740 80.546 80.712 78.738 81.944 83.419 76.220 77.201 76.452 75.356 77.437 77.574 76.005 74.948 75.774 74.571 76.907 77.904 78.194 78.583 78.326 75.435 75.421 75.378 75.672 74.368 76.912 76.266 74.980 74.695 76.044 77.094 76.344 77.984 77.342 77.301 76.288 75.952 74.373 76.569 72.000 72.597 71.659 70.341 1.512 -0.521 0.577 3.247 2.849 0.035 0.391 0.978 1.020 2.036 3.358 3.139 4.413 1.667 3.695 4.094 2.641 2.530 5.333 4.061 4.590 3.145 2.235 0.420 0.019 -0.117 -1.121 0.057 1.330 0.826 -0.875 1.142 1.375 0.449 2.398 0.457 0.793 -1.485 1.817 1.138 0.018 -0.869 3.480 2.096 1.272 1.031 2.070 3.175 4.200 4.266 3.387 4.309 0.981 2.437 4.845 5.422 5.667 6.712 6.176 6.366 4.743 7.012 7.574 6.289 6.377 7.084 5.427 4.259 4.514 5.580 1.370 2.879 3.149 3.219 2.994 3.531 0.647 -1.220 -0.547 -1.163 -0.713 -0.549 -2.154 -1.164 1.511 0.889 -1.607 -1.243 -2.214 -1.987 -1.102 -1.841 406 407 408 409 410 411 412 413 414 415 416 417 418 419 420 421 422 423 424 425 426 427 428 429 430 431 432 433 434 435 436 437 438 439 440 441 442 443 444 445 446 447 448 449 450 451 HA HB1 HB2 HD21 HD22 HN N ND2 O OD1 C CA CB CD CE CG HA HB1 HB2 HD1 HD2 HE1 HE2 HG1 HG2 HN HZ1 HZ2 HZ3 N NZ O C CA HA1 HA2 HN N O C CA CB HA HB1 HB2 HB3 ASN ASN ASN ASN ASN ASN ASN ASN ASN ASN LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS LYS GLY GLY GLY GLY GLY GLY GLY ALA ALA ALA ALA ALA ALA ALA 27 27 27 27 27 27 27 27 27 27 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 29 29 29 29 29 29 29 30 30 30 30 30 30 30 55 72.705 71.463 72.122 69.220 68.366 74.215 73.908 69.216 70.797 70.336 71.675 72.374 71.408 71.074 71.657 72.041 73.224 71.198 70.488 70.926 70.126 71.964 72.511 72.251 72.961 73.789 70.971 69.751 70.424 72.840 70.623 71.631 70.012 70.440 69.565 71.099 71.192 71.129 70.536 69.622 68.572 67.287 68.365 67.506 66.588 66.855 1.599 1.809 0.324 1.746 1.203 3.075 2.204 1.354 3.679 0.545 6.352 5.796 5.813 5.102 4.276 5.084 6.420 6.835 5.319 6.119 4.680 3.310 4.790 4.062 5.580 4.223 4.489 4.590 3.083 4.436 4.096 5.697 9.597 8.177 7.592 8.191 8.037 7.558 10.219 12.492 11.473 11.691 11.608 11.687 10.899 12.642 -2.939 -0.187 -0.872 -0.367 -1.730 -1.031 -1.358 -1.265 -2.043 -2.955 -1.607 -2.844 -4.030 -6.402 -7.550 -5.217 -3.076 -4.308 -3.752 -6.732 -6.099 -7.179 -7.966 -4.941 -5.494 -2.720 -9.505 -8.327 -8.730 -2.599 -8.608 -0.566 -0.919 -0.604 -0.364 0.251 -2.574 -1.721 -1.843 0.062 -0.369 0.416 -1.420 1.473 0.189 0.141 452 453 454 455 456 457 458 459 460 461 462 463 464 465 466 467 468 469 470 471 472 473 474 475 476 477 478 479 480 481 482 483 484 485 486 487 488 489 490 491 492 493 494 495 496 497 HN N O C CA CB CD CG1 CG2 HA HB HD1 HD2 HD3 HG11 HG12 HG21 HG22 HG23 HN N O C CA CB CD CG1 CG2 HA HB HD1 HD2 HD3 HG11 HG12 HG21 HG22 HG23 HN N O C CA HA1 HA2 HN ALA ALA ALA ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE ILE GLY GLY GLY GLY GLY 30 30 30 31 31 31 31 31 31 31 31 31 31 31 31 31 31 31 31 31 31 31 32 32 32 32 32 32 32 32 32 32 32 32 32 32 32 32 32 32 32 33 33 33 33 33 56 68.674 69.058 70.258 71.219 70.782 70.191 68.228 68.883 69.916 71.650 70.897 68.981 67.495 67.742 69.095 68.208 69.041 70.766 69.750 69.267 69.802 72.128 72.298 70.911 69.881 67.420 68.466 70.057 70.912 70.028 67.719 66.464 67.334 68.337 68.333 69.674 71.106 69.518 69.857 70.573 72.544 74.588 74.548 74.940 75.169 72.951 9.576 10.115 13.128 13.175 13.594 15.016 16.362 14.975 15.549 13.610 15.668 17.120 16.405 16.542 14.669 14.264 15.065 15.348 16.615 12.112 12.646 13.773 11.030 11.669 10.629 10.157 11.156 10.386 12.506 9.699 9.151 10.373 10.243 11.294 12.104 11.232 10.256 9.495 11.710 12.150 10.069 9.689 11.032 10.917 11.729 12.328 0.570 -0.153 -0.777 3.305 1.904 1.983 2.801 2.786 0.570 1.261 2.479 2.970 3.593 1.854 3.800 2.332 0.163 -0.065 0.615 1.996 1.373 3.882 5.186 5.185 5.673 5.909 5.396 7.178 5.867 5.141 5.651 5.455 6.982 4.333 5.898 7.730 7.403 7.464 3.347 3.850 4.457 6.784 6.082 5.082 6.624 6.563 498 499 500 501 502 503 504 505 506 507 508 509 510 511 512 513 514 515 516 517 518 519 520 521 522 523 524 525 526 527 528 529 530 531 532 533 534 535 536 537 538 539 540 541 542 543 N O C CA CB CD1 CD2 CG HA HB1 HB2 HD11 HD12 HD13 HD21 HD22 HD23 HG HN N O C CA CB CE CG HA HB1 HB2 HE1 HE2 HE3 HG1 HG2 HN N O SD C CA CB CG1 CG2 HA HB HG11 GLY GLY LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU LEU MET MET MET MET MET MET MET MET MET MET MET MET MET MET MET MET MET VAL VAL VAL VAL VAL VAL VAL VAL 33 33 34 34 34 34 34 34 34 34 34 34 34 34 34 34 34 34 34 34 34 35 35 35 35 35 35 35 35 35 35 35 35 35 35 35 35 35 36 36 36 36 36 36 36 36 57 73.200 74.244 75.301 75.084 76.217 78.684 77.717 77.600 74.150 76.156 76.096 79.656 78.538 78.622 78.754 77.148 77.335 77.736 75.269 75.007 75.737 74.429 75.166 74.631 76.227 75.516 76.217 74.641 73.619 77.206 76.167 76.058 75.441 76.542 74.653 74.997 75.014 74.968 72.476 72.350 70.864 70.081 70.725 72.706 70.473 70.559 11.563 8.664 8.744 8.449 7.559 7.315 9.588 8.095 7.919 7.532 6.558 7.697 7.427 6.269 9.890 10.167 9.762 7.954 10.532 9.687 7.874 9.387 10.357 11.774 15.325 12.774 10.340 12.004 11.838 15.094 15.010 16.390 12.598 12.651 10.624 9.970 8.418 14.458 7.326 8.782 9.181 8.345 10.664 8.877 9.002 8.411 6.003 6.194 10.295 8.812 8.267 7.932 8.339 8.682 8.702 7.189 8.653 8.204 6.868 8.195 8.384 9.051 7.344 9.745 8.466 8.045 11.050 13.024 12.106 12.324 10.980 11.578 12.350 13.378 11.950 11.371 9.949 11.040 10.515 11.892 10.067 10.710 13.510 11.950 13.689 14.125 14.082 15.096 14.430 15.140 13.090 16.062 544 545 546 547 548 549 550 551 552 553 554 555 556 557 558 559 560 561 562 563 564 565 566 567 568 569 570 571 572 573 574 575 576 577 578 579 580 581 582 583 584 585 586 587 588 589 HG12 HG13 HG21 HG22 HG23 HN N O C CA HA1 HA2 HN N O C CA HA1 HA2 HN N O C CA CB CG1 CG2 HA HB HG11 HG12 HG13 HG21 HG22 HG23 HN N O C CA CB CG1 CG2 HA HB HG11 VAL VAL VAL VAL VAL VAL VAL VAL GLY GLY GLY GLY GLY GLY GLY GLY GLY GLY GLY GLY GLY GLY VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL VAL 36 36 36 36 36 36 36 36 37 37 37 37 37 37 37 38 38 38 38 38 38 38 39 39 39 39 39 39 39 39 39 39 39 39 39 39 39 39 40 40 40 40 40 40 40 40 58 70.061 69.071 69.680 71.247 71.150 72.731 73.147 73.134 71.371 71.901 71.310 72.926 71.336 71.846 70.162 70.989 71.872 71.332 72.757 73.223 72.272 71.486 69.012 68.737 67.286 66.924 67.149 68.851 66.620 67.662 66.900 65.952 66.150 67.335 67.866 69.334 69.677 69.369 68.230 69.084 68.748 67.256 69.098 70.125 69.324 67.062 7.314 8.720 10.936 11.257 10.847 10.431 9.646 6.528 4.622 5.622 5.557 5.375 7.658 6.982 4.412 3.638 3.026 2.228 2.618 4.207 4.002 4.206 3.520 4.079 3.743 4.459 2.231 5.152 4.068 4.230 5.525 4.126 2.001 1.728 1.897 3.064 3.526 2.350 2.700 3.921 5.047 5.382 4.595 3.659 5.927 6.294 14.774 15.167 14.436 13.693 15.406 12.849 13.261 14.356 13.076 12.067 11.166 11.834 12.078 12.571 13.174 15.898 14.828 14.341 15.294 13.712 13.831 16.870 18.085 16.693 16.307 15.005 16.111 16.717 17.093 14.251 15.175 14.669 15.770 17.048 15.375 14.930 15.724 18.230 20.808 20.479 21.476 21.395 22.897 20.588 21.226 21.940 590 591 592 593 594 595 596 597 598 HG12 HG13 HG21 HG22 HG23 HN N OT1 OT2 VAL VAL VAL VAL VAL VAL VAL VAL VAL 40 40 40 40 40 40 40 40 40 59 66.679 66.973 68.987 70.120 68.437 68.562 68.848 67.102 68.713 4.577 5.515 5.427 4.245 3.796 5.275 4.354 2.647 1.841 21.825 20.361 23.577 22.922 23.198 18.934 19.106 20.346 21.528